 国家高新企业 | ISO9001认证
国家高新企业 | ISO9001认证 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康

在过去的几十年里,生物技术和计算技术的巨大进步彻底改变了我们对微生物群落的理解。特别是,基于16S rRNA和宏基因组测序的研究进一步验证了一个事实,即微生物群不是静态的实体,而是动态的生态系统,身在其中的组成成员之间,以及成员与环境之间,都在以各种方式进行相互作用。
在这些研究过程中产生了大量的关于微生物相互作用的机制和环境依赖性的数据,也出现了许多专门储存单一类型或进行了部分分类整合的数据库。但由于是以不同的标准去整理的数据,所以很难共享。
//
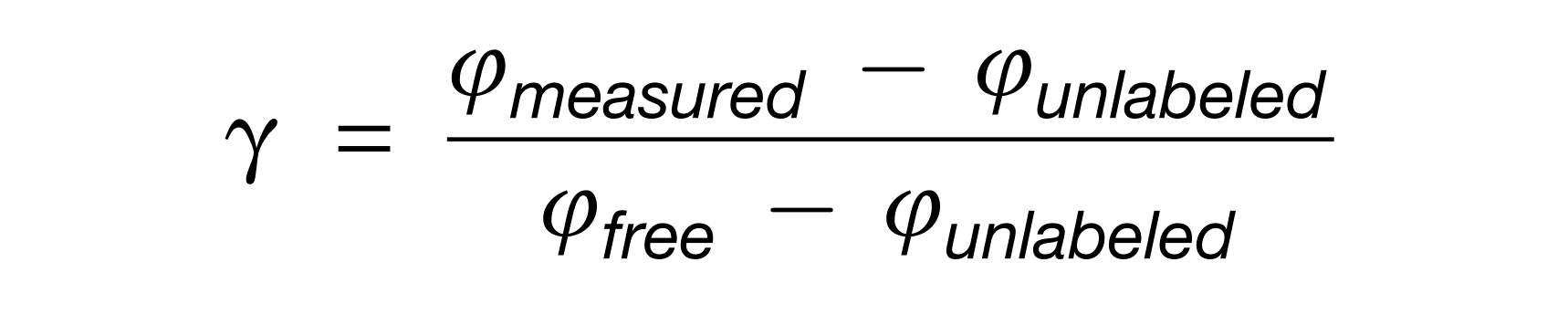
本文研究人员提出了一套体系,用于微生物组数据管理,简称FAIR (findable, accessible, interoperable, and reusable),即可查询、可访问、可交互和可重复使用。这些原则于2016年首次正式提出。
首先基于可查询和可访问的原则,需要数据库,它能存储现有数据也能继续转化新的数据。这个平台可以是自己从零开始设计,可以是使用现成的数据库构建工具作为辅助(比如mako软件),也可以是直接使用已有的数据库,比如GloBI。
关于数据获取,文中列出了一些资源,都是目前广泛使用或者比较独特的,如下表:

其次基于交互性和可重复使用的原则,需要将大数据拆分为一个个子集,每一个子集都应该是能满足大量用户的第一需求或者说是第一想了解的信息点。基于此,研究人员提出了四类元数据作为起点:
1. 微生物实体
应提供参与相互作用的每种微生物的物种(和菌株,如果相关)名称。
示例:
2. 相互作用的推理方法
使用不同的方法来指示高度相关的元数据。
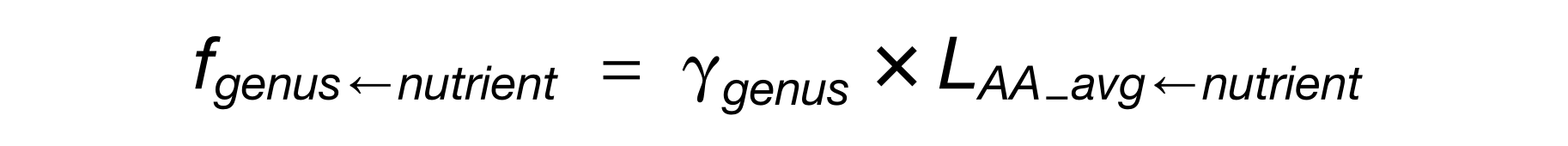
示例:

3. 相互作用的上下文语境
一些环境背景,如生物群落(例如,宿主相关的、合成的)。

示例:

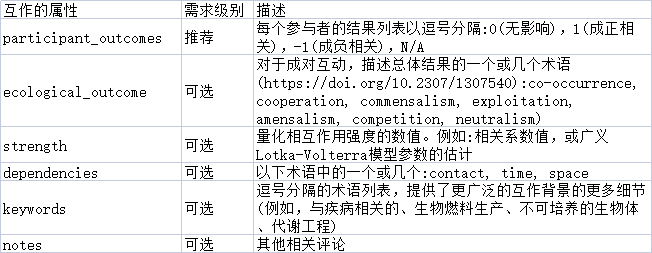
4. 属性,用于定义互作关系的类型
如合作、对抗、关联、成对或高阶等。
示例:

最后,文章中给出了依照FAIR体系研究微生物互作关系的可拓展性。
如下图,通过研究微生物相互作用和相关性(A部分)而产生的多种类型的元数据(B部分),这些元数据(DT),例如真菌细菌或噬菌体细菌的培养实验数据(DT1),扩增子序列或OTU计数的相关性(DT2),两个或两个以上物种基因组尺度代谢网络的通量平衡模型(DT3),直接比对到特定数据库(DT4)。
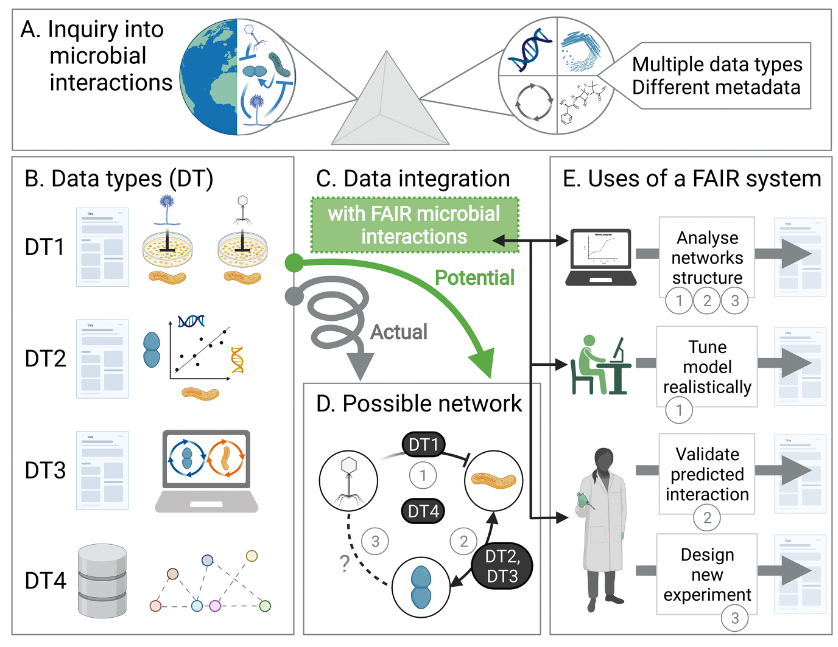
遵循FAIR里的四个原则来解析微生物互作内容(C部分),解析结果如D部分,得到一个可能的相互作用网络关系图谱。
同时,C部分的内容可以横跨不同领域,为不同领域的专家提供信息,例如E部分,建模者可以更容易确定具体的互作以便真实的模拟生态动力学,而实验人员可以评估是否有新的互作在其他宿主或上下文中被报告。
以上内容,是研究人员基于自身经验归纳总结的,研究人员也指出在此概述的实践、标准和示例并不是详尽无遗的,文章旨在促进进一步讨论如何改进微生物相互作用及其属性数据的访问和可用性。希望能进一步的激励各位科学家,共同创建一个共享开放的,拥有统一标准的微生物相互作用资源站。
参考文献:
Pacheco AR, Pauvert C, Kishore D, Segrè D. Toward FAIR Representations of Microbial Interactions. mSystems. 2022 Aug 25:e0065922. doi: 10.1128/msystems.00659-22. Epub ahead of print. PMID: 36005399.
谷禾健康
为了使研究更系统更全面,越来越多的研究人员追求在多组学背景下解释分子数据。
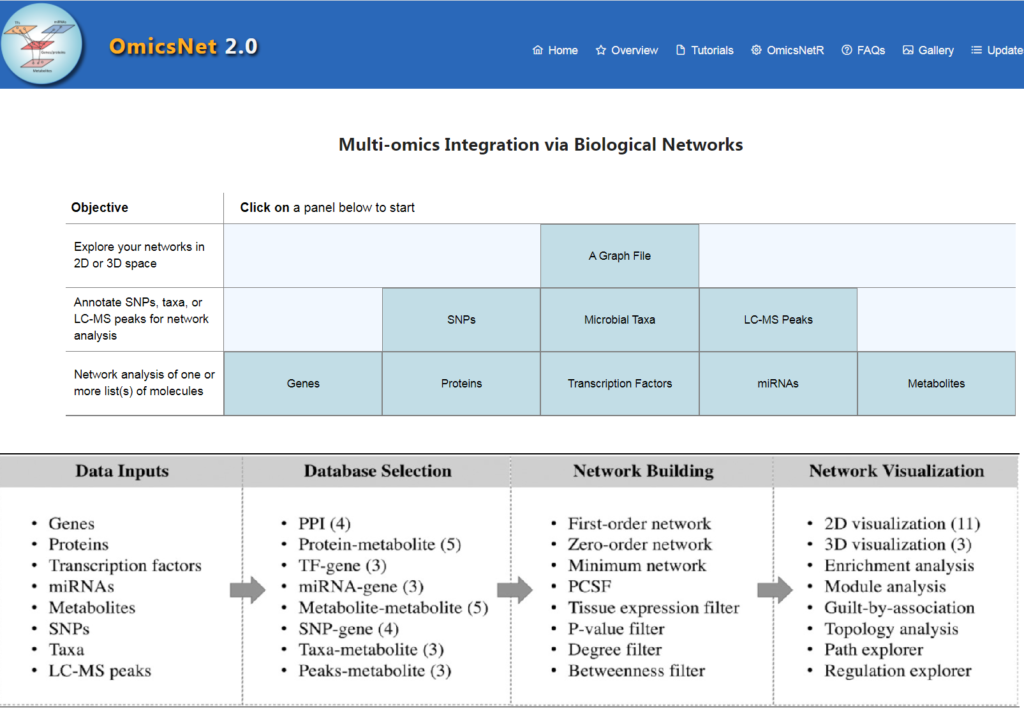
OmicsNet (www.omicsnet.ca) 顺应而生,这是一个在线平台,允许用户轻松地构建、可视化和分析多组学网络。
OmicsNet 2.0在原有基础上进行了升级,主要改进了三个方面:
(1) 可视化分析中2D图形布局选项增加了11个,以及一个新颖的3D模块。
(2) 支持三种新的组学类型:
(3) 分析时将同时输出使用的R包OmicsNetR和历史命令,并生成共享链接可在网页查看和交互操作,从而提高研究的可重复性。
研究中使用OmicsNet 2.0对炎症性肠病(IBD)的数据进行多组学分析。
OmicsNet 2.0工作流程

如图,主要为4个步骤:
OmicsNet 2.0 的改进
1、更新了分子相互作用数据库
包括PPI数据库(STRING,InnateDB和IntAct),TF-target数据库(TRRUST和JASPAR), miRNA-target数据库(TarBase和miRTarBase),代谢数据库(KEGG, Recon3和AGORA)。
2、支持三种新的组学类型
可支持来自遗传变异研究的SNP矩阵、来自非靶向代谢组学的 LC-MS 峰和来自微生物组分析的物种丰度矩阵数据的输入及互作分析。
对于那些对影响基因调控的变异感兴趣的用户,可以分别基于ADmiRE和SNP2TFBS将SNPs映射到miRNAs或TF结合位点。由此产生的网络可以通过proteins、miRNAs或TFs进一步扩展,以了解潜在的影响。
对于代谢组学分析,使用最近发布的NetID算法对LC-MS峰(m/z, retention time, intensity和p-value)进行数据处理,可选三种数据库(KEGG, PubChemLite_BioPathway和HMDB)进行注释。
使用Rcpp/C++ 引擎重新编写了核心算法,使其速度提高了10 倍以上。使用 lpsymphony 包进一步优化了整数线性规划。
直接上传物种丰度矩阵,可以使用贝叶斯逻辑模型预测潜在代谢物,该模型使用超过 6000 个高质量基因组规模的代谢模型进行训练。
3、代码开源
开放R包OmicsNetR(https://github.com/xia-lab/OmicsNetR),显示分析期间所执行的R命令。结果可输出为网页链接,有效期一个月。
4、减少假阳性
对于来自人类和小鼠的数据,提供了一个基于ENCODE、基因型-组织表达(GTEx)或人类蛋白图谱(HPA)的基因表达数据的过滤器,这些过滤器帮助研究人员专注于与生物相关的相互作用,并减少假阳性。
5、支持 2D 和 3D 网络可视化
2D模块可支持11种图形布局,3D模块可支持交互。图形化主要基于 igraph 包和 graphlayouts 包。
使用OmicsNet 2.0进行的IBD多组学研究
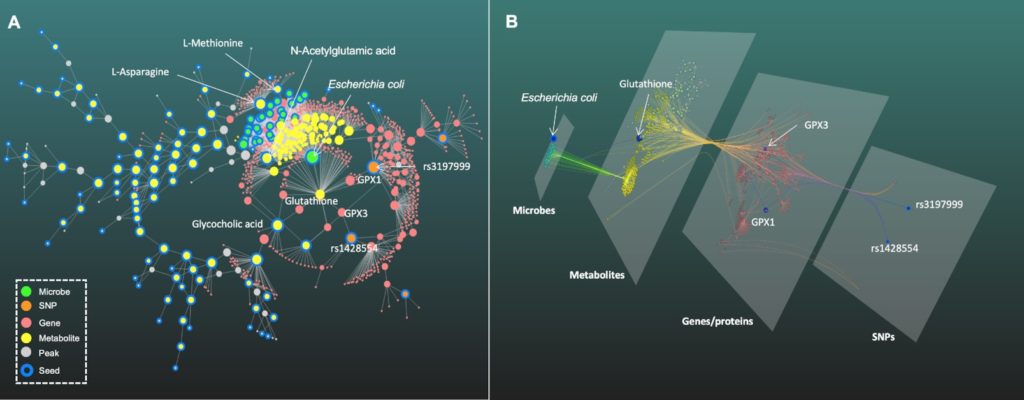
图A为2D视图,图B为3D视图
将获得的物种丰度矩阵,SNP矩阵和LC-MS峰数据上传至OmicsNet 2.0平台。
利用AGORA数据库预测潜在的微生物代谢物(potential score:0.9)。
利用PhenoScanner对基于eQTLs的基因进行SNP定位。
利用KEGG数据库对LC-MS峰进行注释。
通过添加代谢物-蛋白质相互作用,从SNPs和LC-MS峰生成的个体网络进一步扩大,这样三个网络可以在代谢组学层合并。
p-value过滤:(cut-off: 0.2)来排除p值较大的LC-MS峰值所贡献的节点。其网络连接基于其相关代谢物、基因或蛋白质。
如图A,生成的子网络1包含六种类型的节点(物种、SNPs、代谢物、SNPs或代谢物相关的基因/蛋白质),结果显示出glutathione是宿主-微生物相互作用中的重要交互作用点。
由此推测包括大肠杆菌在内的几种微生物会产生glutathione,两个SNPs(rs3197999和rs1428554)通过编码谷胱甘肽过氧化物酶(GPx)的基因GPX1和GPX3与代谢产物相关。
与此呼应的是,在以往的研究中证明大肠杆菌和相关物种在IBD中的过度表达可能是由于它们能更好地产生glutathione以抵抗氧化应激。
图B的3D分层网络提供了一个直观的多组学整合视角,突出显示了连接微生物组和宿主遗传的glutathione的流动路径。
OmicsNet 2.0与其它工具的比较
如下表,比较OmicsNet 2.0与其他基于web的多组学集成工具的关键特性。
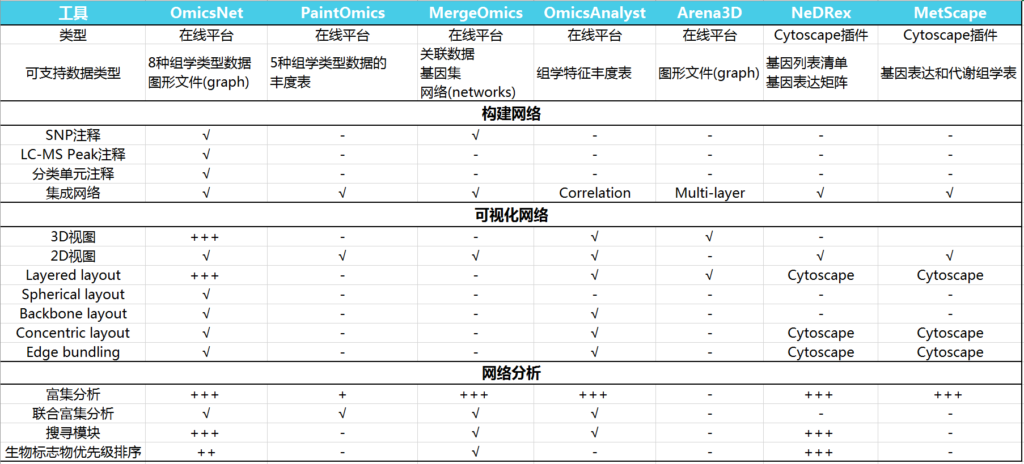
‘√’表示支持,‘-’ 表示缺失,“+”表示定量评估(“+”越多表示更好的支持)
附带各工具的官网及主要特点描述:
PaintOmics
(http://www.paintomics.org)
专注于在视觉上呈现多组学数据的探索分析,包括转录组学,代谢组学,表观基因组学,miRNA和转录因子,将其映射到KEGG通路联合分析。
MergeOmics
(http://mergeomics.research.idre.ucla.edu)
整合了来自单个组学层面的关联研究的汇总统计数据,以及不同功能基因组学数据,以获得机理上的见解,最近还加入了多组学信息的药物重新定位(drug repositioning)。
OmicsAnalyst
(https://www.omicsanalyst.ca)
基于输入的数据,利用多元统计、相关性分析和聚类方法,结合网络、热图和散点图进行多组学分析。
Arena3D
(https://www.arena3d.org)
擅长使用基于3D的分层布局对多层网络进行交互式可视化,适用于多组学网络数据。
NedRex
(https://nedrex.net)
是一个Cytoscape插件,专注于疾病模块识别和药物再利用,使用各种模块识别和优先排序算法。
MetScape
(http://metscape.ncibi.org)
是一个Cytoscape插件,通过构建和分析不同类型的含有酶、代谢物和/或反应的网络,专注于基因表达和代谢组学数据的集成和可视化。
OmicsNet
(https://www.omicsnet.ca)
通过将多个分子互作数据库与强大的2D/3D可视化网络分析相结合,对于多组学集成分析和结果解释有更好的理论基础,对以上工具的不足做了补充。
结 语
OmicsNet 2.0是一个基于网络的多组学分析平台,支持2D和3D网络可视化探索。
在1.0版本中强调基于web的3D网络可视化。
在2.0版本中,进一步改进了其可视化分析系统,添加了一个功能完整的2D网络可视化系统,并支持三种新的组学数据输入(SNPs、微生物分类单元和LC-MS峰),目前的生物信息学工具无法很好地支持这些数据。
除了富集分析、搜寻模块和最短路径分析,用户还可以使用重启随机游走算法(Random Walk with Restart)搜索候选疾病标记物。最后,2.0版本通过发布底层的R代码和共享链接,改进了工具的可再现性和透明度。
使用IBD多组学数据的案例研究表明,OmicsNet 2.0可以揭示与原始出版物和后续出版物以及IBD文献一致的有意义的模式、关系和功能。总之,OmicsNet 2.0解决了对多组学数据进行分析的一些需求。
参考文献:
Zhou G, Pang Z, Lu Y, Ewald J, Xia J. OmicsNet 2.0: a web-based platform for multi-omics integration and network visual analytics. Nucleic Acids Res. 2022 May 26:gkac376. doi: 10.1093/nar/gkac376. Epub ahead of print. PMID: 35639733.

谷禾健康

尽管地球上微生物类群的繁多,但只有一小部分得到了培养和有效命名。因为大多数菌无法在非常特定的条件下培养分离鉴定。
在过去十年中,宏基因组研究的重要性已经凸显,因为它能够评估细菌基因库并发现当前实验室培养技术无法掌握的新细菌基因组。这些数据对于扩大我们对地球上微生物多样性的理解至关重要。
由于宏基因组测序数据由来自多个物种和菌株的 DNA 序列片段组成,通常有数千个来自不同生命领域,因此此类分析的主要挑战是正确确定每个 DNA 序列片段的真实来源。不幸的是,这些步骤容易出错,因此必须对结果进行严格审查,以避免发布不完整和低质量的基因组。
最近,比利时研究人员新开发MAGISTA,这是一种评估宏基因组基因组组装质量的新方法,基于随机森林的方法估计MAGs的完整性和污染度,解决了当前基于参考基因的方法经常被忽视的一些缺陷。
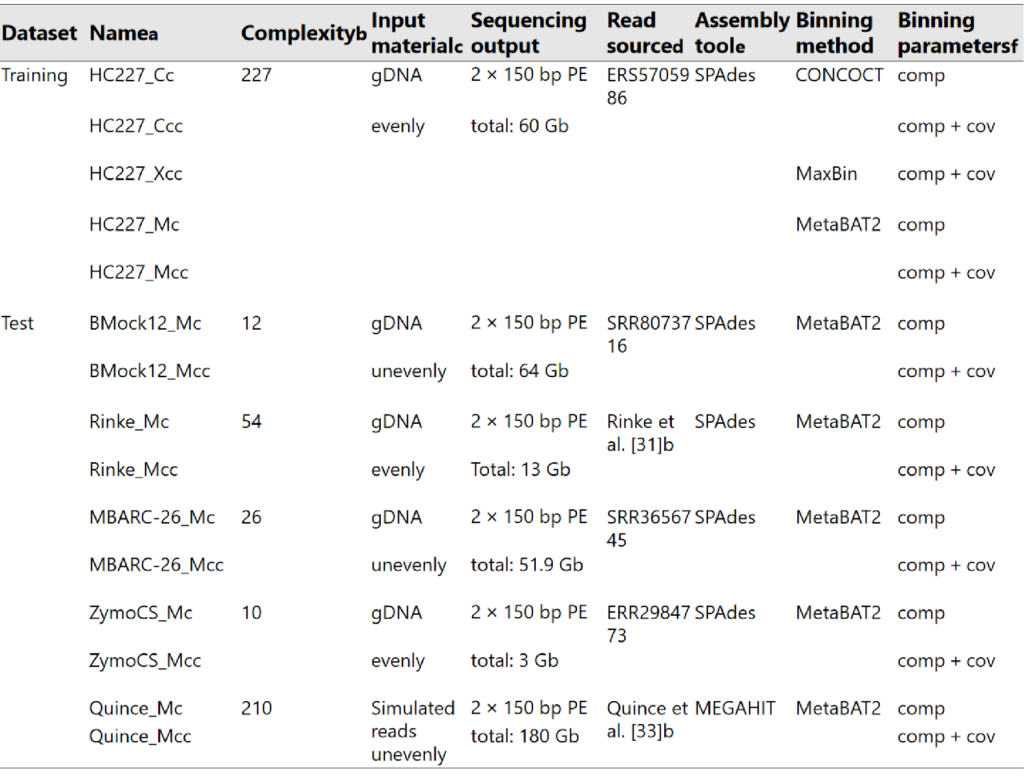
MAGISTA是基于宏基因组bins内的contig片段之间的无对齐距离分布,而不是一组参考基因。该方法利用了来自整个 bin 的信息。为了正确评估此方法,并说明基于参考的工具的缺点,最近,比利时研究人员构建了一个高度复杂的 DNA 模拟群落,由 227 个细菌菌株组成,并且具有不同程度的相似性。

方 法
训练集来(HC227)自 227 个细菌菌株,测试数据集由五个公开可用的短读(short reads)子集构成,其中四个含有来自复杂度相对较低的基因组 DNA 模拟群落的reads。具体情况如下图所示。
Complexity列指示菌株数;Assembly tool列表示所使用的用于组装的软件;Binning method列表示所使用的用于分箱的工具;Binning parameters列表示所使用的用于评估分箱质量的指标,comp为完整度,cov为覆盖率。
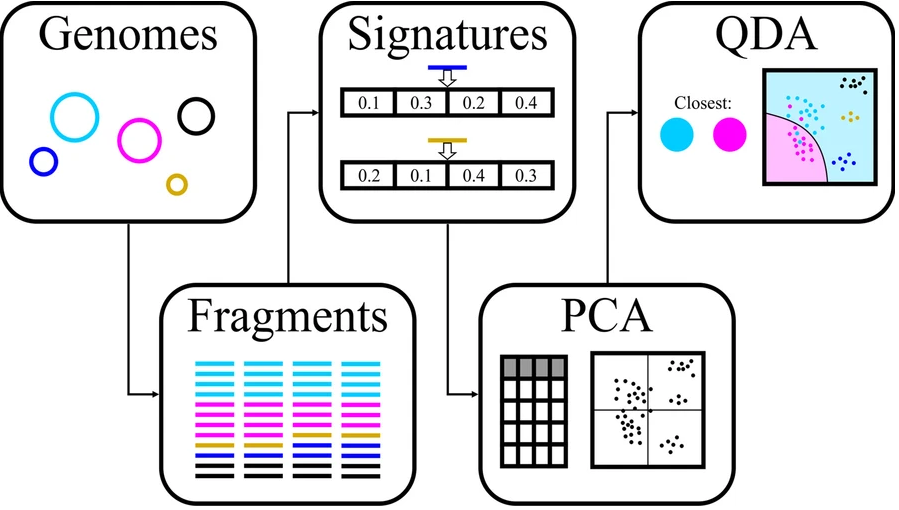
MAGISTA计算步骤:
输入binning后的每个bins
-●-
第 1 步:选择适合的片段大小与距离计算方法
-●-
首先将每个 bin 中的每个 contig 拆分为固定长度的片段,然后使用四种不同的方法(即 PaSiT4、MMZ3、MMZ4 和 Freq4)计算一个 bin 中的片段之间的所有距离。对于每种方法,都选择了特定的片段长度,以便为不同的生物产生不同的特征分布。
每种方法的最终片段长度的选择是通过不同方法分析整合决定的,方法如下图所示。每组的设计中至少两个基因组来自同一个家族,两个基因组来自相同的顺序但来自不同的家族。这些基因组被人为地分成所需长度的片段,并为每个片段计算目标特征。
对于每组五个基因组,混合所有片段并根据它们的特征进行主成分分析(PCA),然后进行二次判别分析,用于生成分类器,旨在区分每组中重叠最多的两个基因组。对该分类器的准确度取平均值,结果用于选择方法和片段长度的最终组合。
-●-
第 2 步:模型中特征变量的选择
-●-
为每种方法选择片段长度后,使用平均值、标准差、偏度、峰度和中位数以及 2.5%、5%、10%、90%、95% 和 97.5% 百分位数计算距离分布。此外,还计算了 1 kb 片段的 GC含量分布。以及每个bin的大小,共计66个特征变量。
-●-
第3步:模型构建
-●-
使用 R (v 4.0.3) 包“RandomForest”中的“RandomForest”函数和默认参数训练随机森林模型。同时使用R包lm再建立一个线性模型执行线性回归,输入经对数转换后的特征变量值,用于交叉验证分析。
主 要 结 果
一个高度复杂的基因组DNA模拟群落
由来自 227 个细菌菌株的基因组 DNA 组成,这些菌株属于8 个门(Actinobacteria, Bacteroidetes,Deinococcus-Thermus, Firmicutes,Fusobacteria,Planctomycetes, Proteobacteria和Verrucomicrobia),18 类,47目,85科,175属,197种。
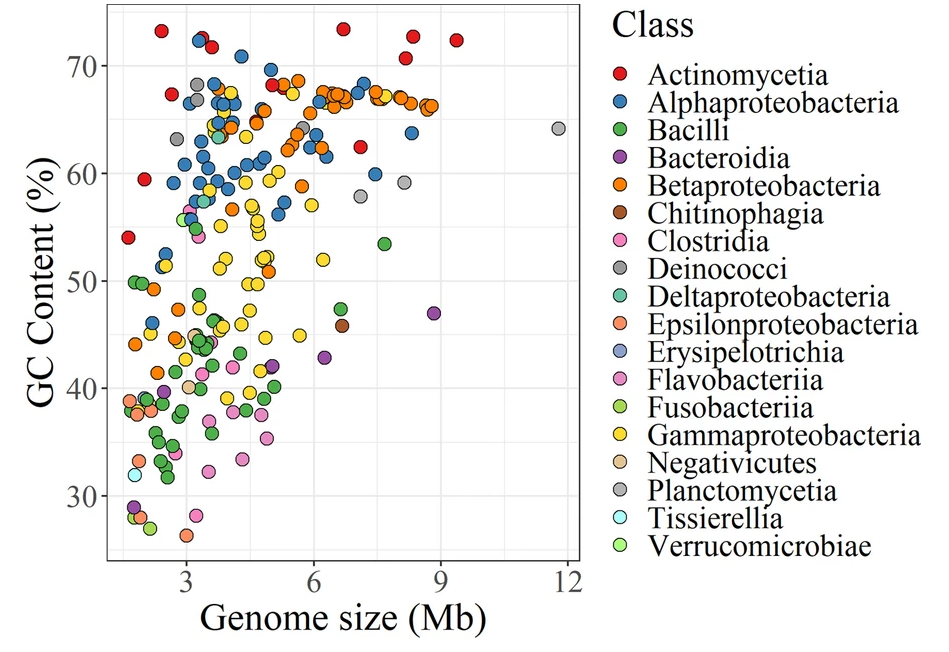
编辑
上图为模拟群落中的细菌菌株的基因组大小和GC含量(从26.3%到73.4%)散点图;
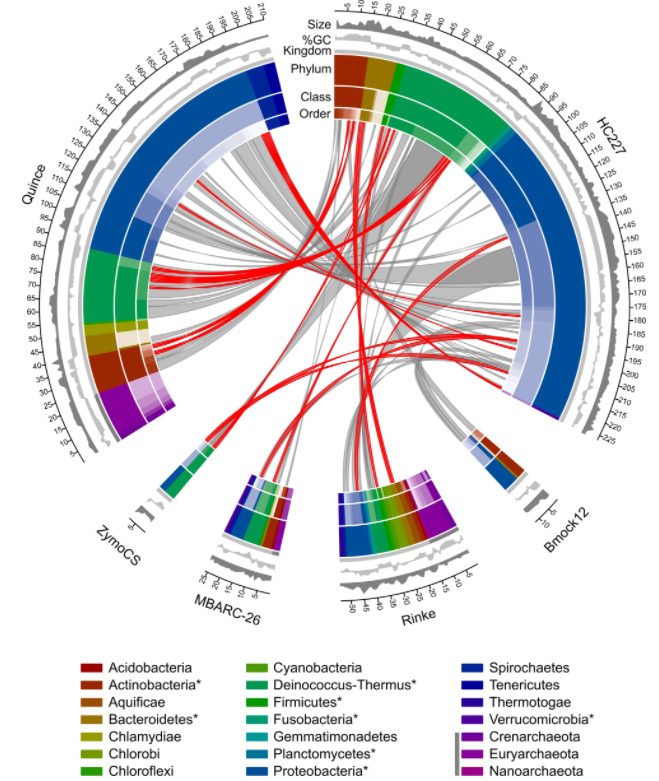
编辑
图为训练集与测试集中物种之间的关系图。红色线条表示在训练集中存在的菌种,灰色线条表示在训练集中存在的菌属。环状图中的不同颜色代表不同分类水平。图例中存在于训练集中的菌门用*标记,存在于古生菌的菌门用深灰色色带标记。
CheckM中基于单拷贝标记基因(SCMG)来评估 bin 质量的存在的缺陷
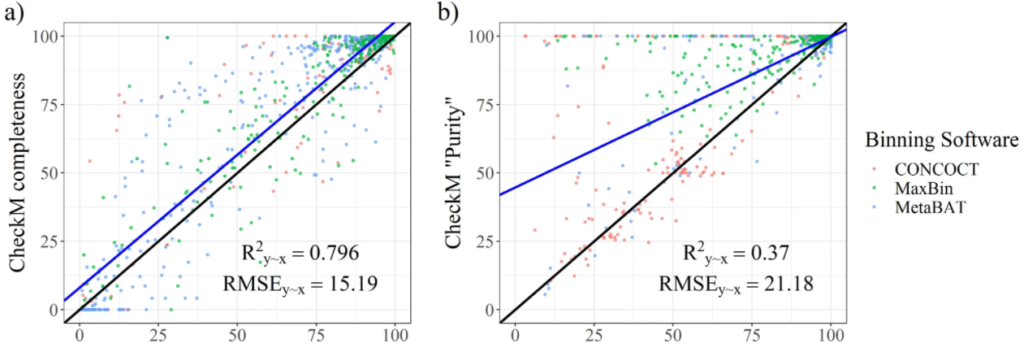
图a和b分别为从CheckM中输出的完整性指标和污染度。使用R^2y∼x(解释方差的百分比),RMSE(相对于实际值的均方根误差)两个参数评估结果。结果表示CheckM高估了bin的质量。许多受污染的bins被预测为接近未受污染。
使用MAGISTA分析模拟群落中的bins
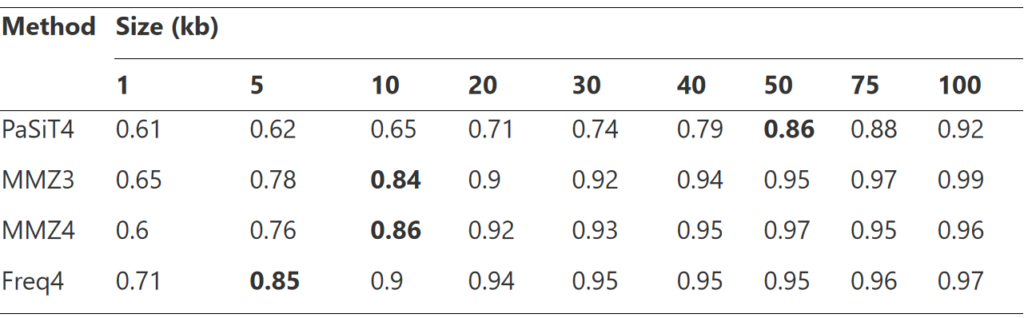
首先选择最佳片段大小用于计算距离分布,如上图所示,考虑了 1、5、10、20、30、40、50、75 和 100 kb 的片段,最终选择了粗体所示的片段大小。
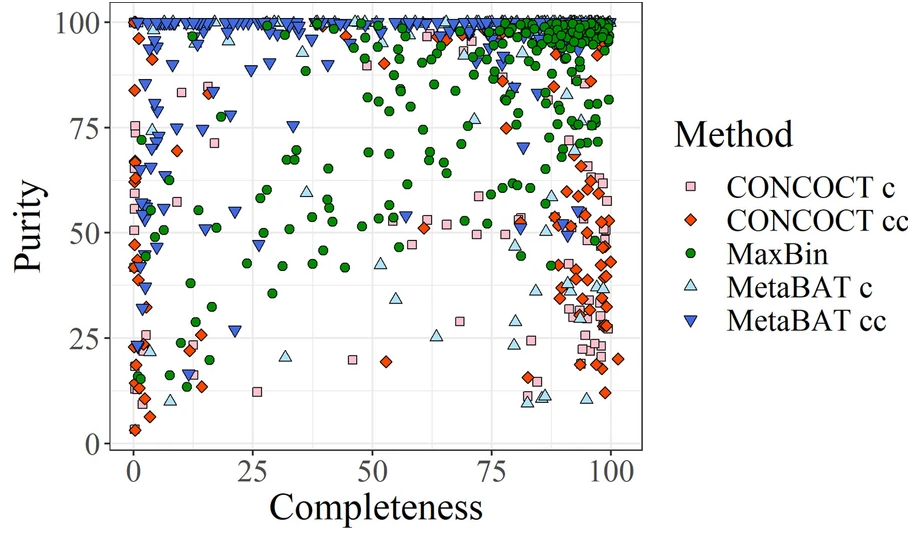
图为concont、MetaBAT和MaxBin产生的bins的完整性和污染度信息。
由于通过模拟生成这样的数据集并不能准确地表示真实的结果,所以使用了binning软件的结果,提供了一组不同质量的真实的bins。训练数据集的完整性和未污染度均在90%以上。
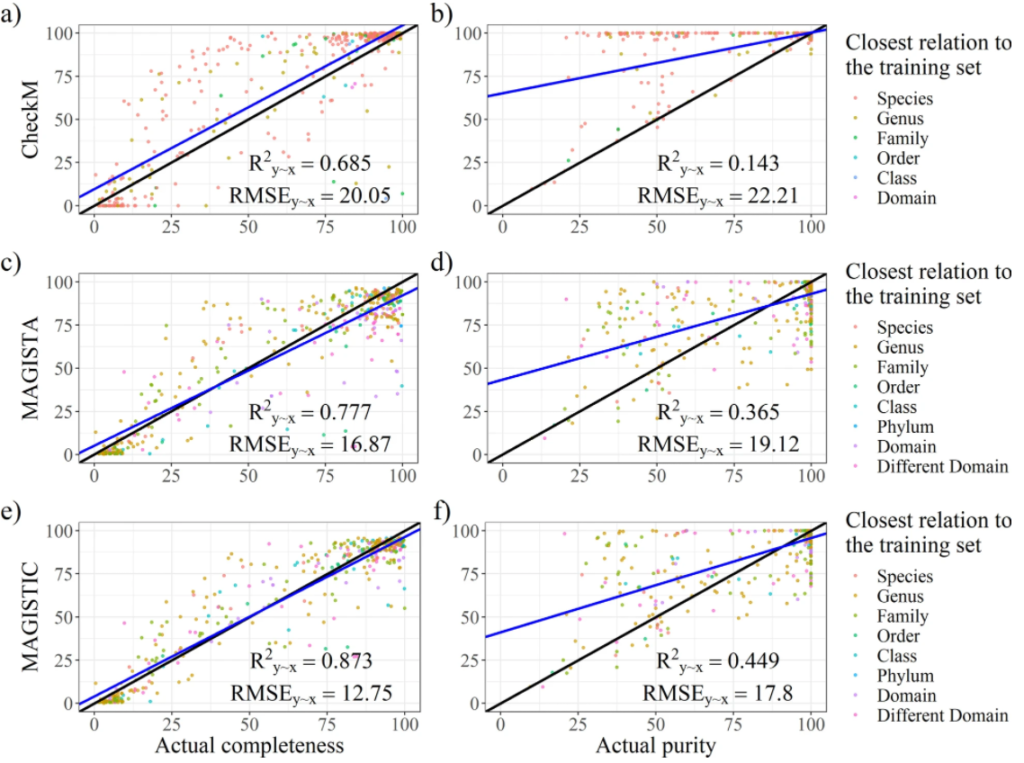
最后是模型构建,建立完整性和污染度的预测模型。并进行了模型评估,如图所示。分别对CheckM、MAGISTA 和 MAGISTIC测试了其性能。CheckM是现在主流的一款评估bin质量的工具。MAGISTIC是一款结合了CheckM和MAGISTA 的工具。使用解释方差的分数(R2y∼x)和均方根误差(RMSE)作为评估性能的指标。对于完整性的预测,MAGISTA 优于 CheckM。对于污染度的预测,MAGISTA 的表现优于 CheckM。
结 论
研究人员开发了一种新的用于预测高度复杂的宏基因组组装基因组bin的质量的方法,MAGISTA。是基于 SCMG 的低复杂性宏基因组方法的一个同样好的替代方法。除了MAGISTA之外,还通过结合CheckM的结果,使用MAGISTIC生成了一个更准确的预测。
研究人员在文章中指出MAGISTA 和 CheckM 都没有达到足够的准确度来被认为是可靠的。MAGISTIC 产生了比 MAGISTA 更好的结果。
在附加分析中,将测试集分为了两个子集,从真实和模拟reads中获得的bins,对此再进行分析,结果表示,CheckM 对于“真实”子集表现良好(但相比MAGISTA 和 MAGISTIC还是较差),对于“模拟”子集部分表现较差。而MAGISTIC相比MAGISTA会更准确些。但是文章中并没有详细说明MAGISTIC的工作流程。
查看作者在github上公开的软件说明,地址如下。但是没有说明和给出输出文件的内容。个人认为还不太成熟。
https://github.com/LM-UGent/MAGISTA
参考文献:
Goussarov G, Claesen J, Mysara M, Cleenwerck I, Leys N, Vandamme P, Van Houdt R. Accurate prediction of metagenome-assembled genome completeness by MAGISTA, a random forest model built on alignment-free intra-bin statistics. Environ Microbiome. 2022 Mar 5;17(1):9. doi: 10.1186/s40793-022-00403-7. PMID: 35248155; PMCID: PMC8898458.

谷禾健康

微生物物种的遗传变异研究通常包括单核苷酸多态性(SNPs)、结构变异(structural variants ,SV)和可移动遗传元件(mobile genetic elements,MGEs)。
在宏基因组中,SNP被用来量化种群结构、追踪菌株和鉴定微生物表型的遗传决定因素。然而,现有的基于比对的宏基因组SNP检测方法需要高性能的计算和足够的覆盖深度来区分SNP和测序错误。
为了解决这些问题,美国加利福尼亚大学研究人员使用高质量基因组,构建了 909 个人类肠道物种中 1.04 亿个 SNPs 的目录,并使用针对该目录的独特 k-mers 表征来自 7,459 个样本的肠道菌群的全球种群结构,开发了GenoTyper for Prokaryotes(GT-Pro),可以对宏基因组的这些 SNPs进行快速基因分型的方法。该研究成果近日公开在《Nature Biotechnology》发表。
该方法与使用读长对齐的方法相比,GT-Pro 更准确,速度快两个数量级,作者构建了一个GT-Pro数据库,基于大约25,000个宏基因组样本,并展示了GT-Pro如何用于数千种菌群的菌株水平探索,可以实现在个人电脑上快速高效地对数百万个SNP进行宏基因组分型。

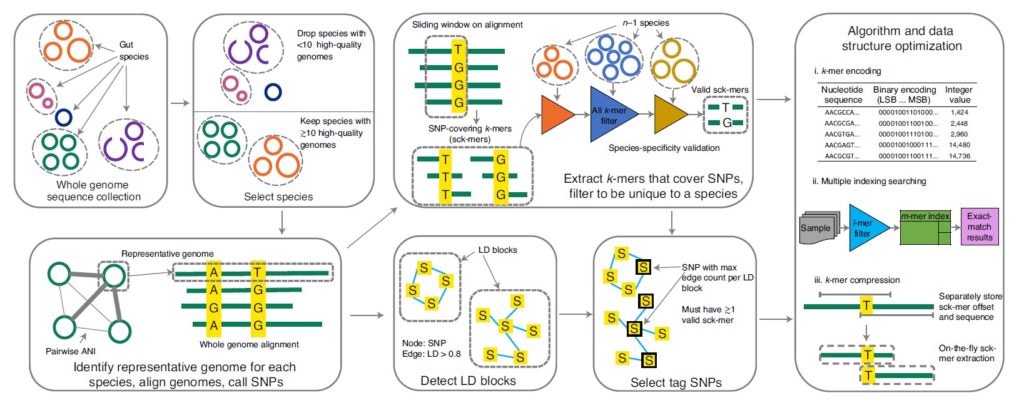
如图,按箭头方向所示。
首先从全基因组序列中识别高质量基因组的物种(去除<10 个高质量基因组的物种,高质量基因组:≥90% 的完整性和≤5% 的污染),对于每个物种,一个有代表性的基因组是根据平均核苷酸一致性(Average Nucleotide Identity,ANI)和组装质量指标选择的,确定代表性基因组后,对每个物种,通过MUMmer软件将每个同种基因组(conspecific genome )与代表性基因组比对,确定SNP,在这些SNP中选择常见的双等位基因SNP用于分型(site prevalence ≥90% and minor allele frequency >1%)。
接下来提取覆盖SNPs的k-mers(sck-mers),过滤出独有的物种,同时检测LD块,并选择具有物种特异性的sck-mers的SNPs和该块中其它SNP的最高LD。LD块为基于跨基因组的共现模式将 SNP 聚类成linkage disequilibrium block。检测LD块使用R2 阈值 (0.81) 。具有物种特异性的sck-mers即删除了两个或多个物种共有的任何sck-mer。
最右边的方框里简要是GT-Pro的算法和数据结构的优化方法。也是该研究的主要目标之一,正是利用了该方法构建的SNP索引才能实现快速地分型。
首先是k-mers编码,选择了k=31,以便使用64位整数编码,通过这一步骤,GT-Pro 数据库缩小了四倍。
其次是多索引检索和进一步压缩SNP数据结构。
优化后的GT-Pro数据库由两个表组成:
(1)10.6 GB的sck-mers表,包含每个k-mer的4字节条目;
(2)2.4 GB的sc-span表,包含每个等位基因的24字节条目。
所需的总存储空间为13 GB,是原始sck-mer表的bzip2压缩的两倍。也使得GT-Pro可以在个人计算机中高效运行。
1.从模拟宏基因组中准确识别SNP
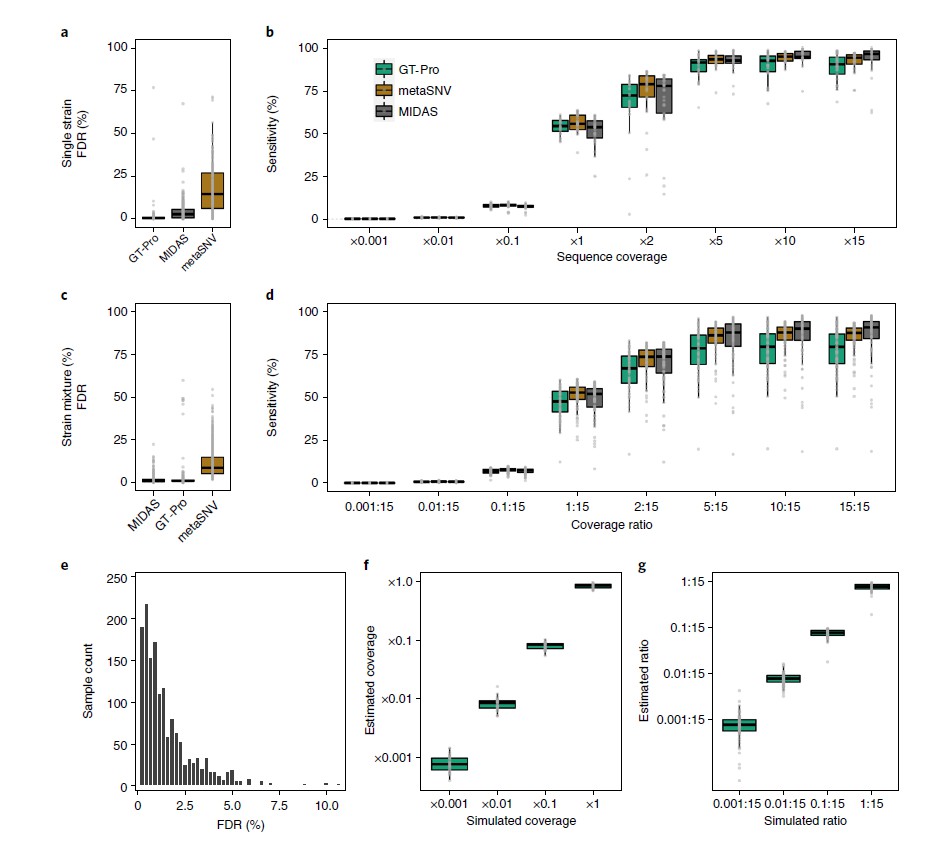
比较GT-Pro、MIDAS和metaSNV宏基因分型的准确性,使用232个未用于开发这些方法的人类肠道分离株的模拟宏基因组(大约2600万次reads)。
图a为FDR比较,假阳性指不正确的基因型,是由测序错误和读数映射到错误位点导致的。假阴性指缺失的基因型,在没有读数映射时产生。在宏基因组中,FDR最低的是GT-Pro(中位数,0.4%),而 metaSNV 最高(中位数,14.5%)。
图b为对图a的灵敏度调查,用于直接比较不同方法。敏感性是指在GT-Pro数据库中检测到分离株基因组(参考和非参考等位基因)中存在基因型的概率。结果表示,随着覆盖度的加深,GT-Pro的灵敏度损失较小。
图c为比较三个工具在一对同种分离株但不同覆盖率下的FDR,目的是检查宏基因组分型方法对菌株混合物的表现。其中一个菌株始终为15倍的覆盖率,另一个菌株的覆盖率从 0.001 到 15 倍不等。FDR包括纯合位点和杂合位点。
总体而言,GT-Pro的 FDR与 MIDAS 相似但低于 metaSNV。
图d为对图c的灵敏度调查,敏感性是指正确判断reads所模拟的基因组的基因型(纯合位点和杂合位点)的概率。GT-Pro 的灵敏度低于基于比对的方法,基于比对的宏基因分型通常使用覆盖率和等位基因频率过滤来减少错误的杂合性调用。
图e为基于图a中模拟的等位基因,从tag SNPs推算的基因型的FDR。结果表示大多数物种的 FDR 较高但仍低于 5%。
图f和图g,为了探索 GT-Pro 是否能用于定量估计物种丰度,使用从单个分离株和对同种分离株中模拟的宏基因组,比较了sck-mer匹配reads的平均数量和已知的基因组覆盖率。结果表示GT-Pro等位基因的调用和计数可以用一个小的校正因子来估计物种和菌株的相对丰度。
所有的结果表示,在模拟宏基因组的测试中,metaSNV 和 MIDAS 对于丰富的物种(>5×覆盖度)和保守位点表现良好,但 GT-Pro 对典型覆盖率值、非参考和杂合位点更准确和敏感,同时对错配和测序错误更为稳健。只是,与 metaSNV 和 MIDAS 相比,GT-Pro 无法检测其数据库中缺少的新 SNP。
结论是,在保守的基因组区域仔细选择sck-mers能使 GT-Pro 能对来自鸟枪法宏基因组数据的已知 SNPs 进行敏感和特异性的基因分型。
2.从模拟宏基因组中准确识别SNP
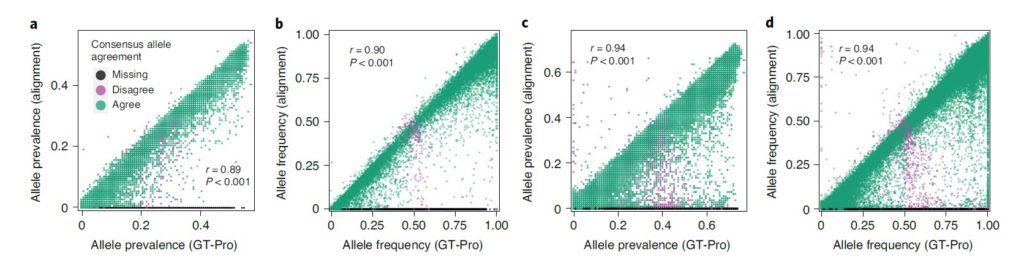
使用GT-Pro对肠道微生物组样本进行宏基因组分型,结果与基于比对的MIDAS宏基因组分型比较。
图a和b分别为流行率(prevalence)、平均等位基因频率(Average allele frequency)
图c和d类似图a和b,只是物种不同。
每个点代表一个 SNP,颜色表示两种方法的共有等位基因(即样品中最常见的)是否相同(绿色),两种方法都返回某些样品的基因型,但共有等位基因不同(紫色)或仅GT-Pro 返回基因型(黑色)。
结果表示对于高覆盖率物种,基于比对的方法能检测到GT-Pro数据库中没有的SNP,而GT-Pro 在中低覆盖率物种中检测到更多SNP位点。这部分结果也与模拟宏基因组测试时的结论一致。
1.使用GT-Pro的SNP估算结构变异
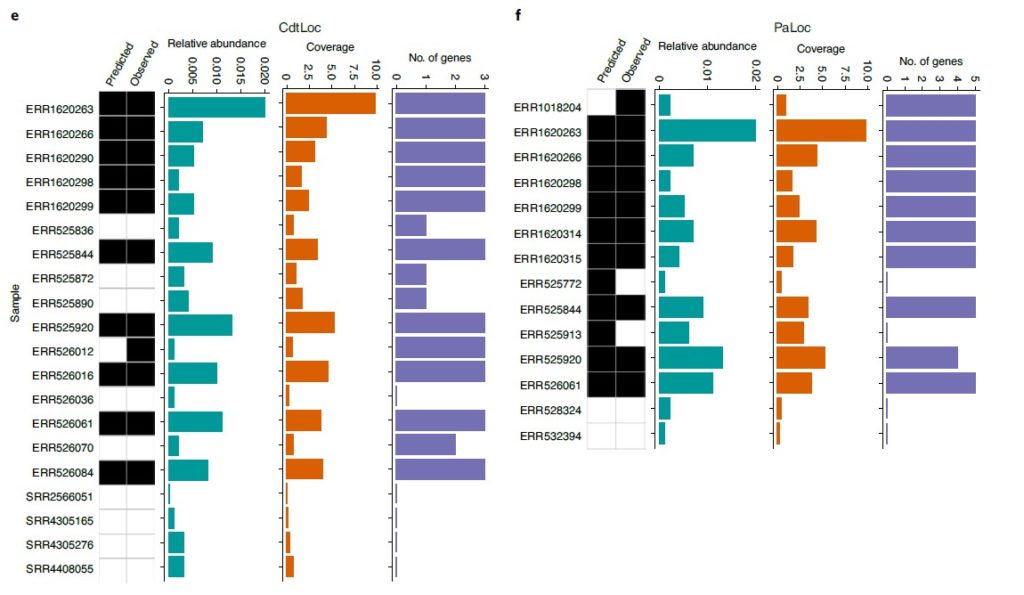
研究人员试图使用GT-Pro的SNP推断附近基因或操纵子的存在,从而作为结构变异的生物标志物。
首先对艰难梭菌的毒性控制位点CdtLoc和PaLoc的侧翼区域使用GT-Pro检索SNP。
接着用艰难梭菌的参考基因组训练了一个随机森林分类器,用于预测来自混合群组(n = 7,459)的人类肠道宏基因组中存在/不存在艰难梭菌毒素基因位点。
图e和f分别代表CdtLoc基因和PaLoc基因,对每个样本,最左边的热图,第一列为预测的,第二列为基于比对方法得到的,黑色表示存在,白色表示不存在。
从左到右的条形图分别指艰难梭菌的相对丰度、全基因组序列覆盖率,从毒素位点检测到的基因数目,所有这些都是通过比对到艰难梭菌的代表性基因组来估计的。结果表示预测到艰难梭菌毒素位点的概率>0.6。
对CdtLoc的几个预测与宿主的表型相关(P < 0.001),包括5名艰难梭菌阳性和CdtLoc(+)的克罗恩病患者,这与该人群对艰难梭菌病理的高易感性相一致。与此相反,CdtLoc基因座在大多数可检测到艰难梭菌的健康婴儿中没有被预测,这与婴儿期艰难梭菌常见的无症状定殖一致。这些结果表明,GT-Pro可以预测具有临床相关性的linked structural variants。
2. 使用 GT-Pro 捕获新的种内遗传结构
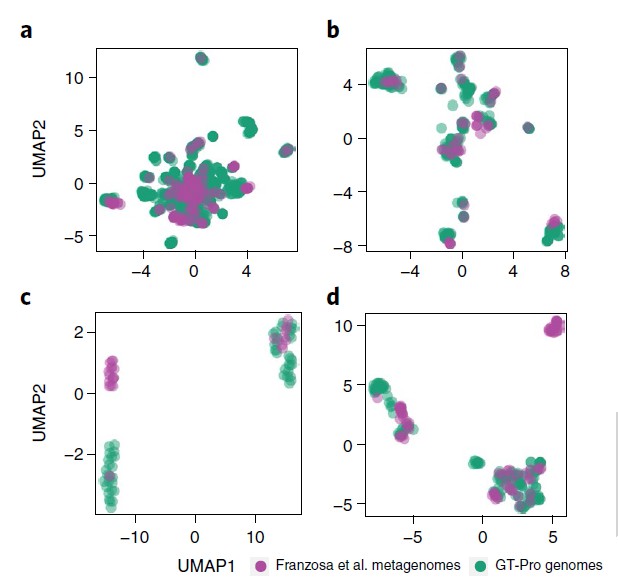
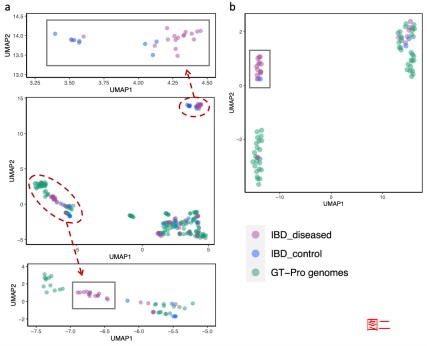
GT-Pro 可以对从参考基因组中鉴定的已知 SNP 进行宏基因组分型分析。但研究人员认为GT-Pro还有更广阔的发展,假设GT-Pro可以基于 SNP 等位基因的不同组合检测新的菌株变异。
为了验证该假设,研究人员使用GT-Pro 对最近发表的北美炎症性肠病 (IBD) 队列 的 220 个粪便宏基因组中发现的物种进行基因分型。使用UMAP降维分析,每个图都是将UMAP应用于一个物种GT-Pro SNPs基因型矩阵的结果。每个点代表该物种的一个菌株(杂合宏基因组的主等位基因)。紫色为队列样本,绿色为GT-Pro基因组。
结果表明 GT-Pro 的数据库代表了这些个体的常见菌株多样性,对于大多数物种,如图一的a和b,粪便样本组与参考基因组聚集在一起,相比之下,对于少数物种。
如图二的c和d,分别是新的亚种,观察到基因型与数据库中任何参考基因组不同的粪便样本群,包括一些富含IBD患者的样本。这说明可以使用 GT-Pro 常见 SNP 发现新的亚种遗传结构。
3. GT-Pro 探索全球人类肠道微生物组遗传变异
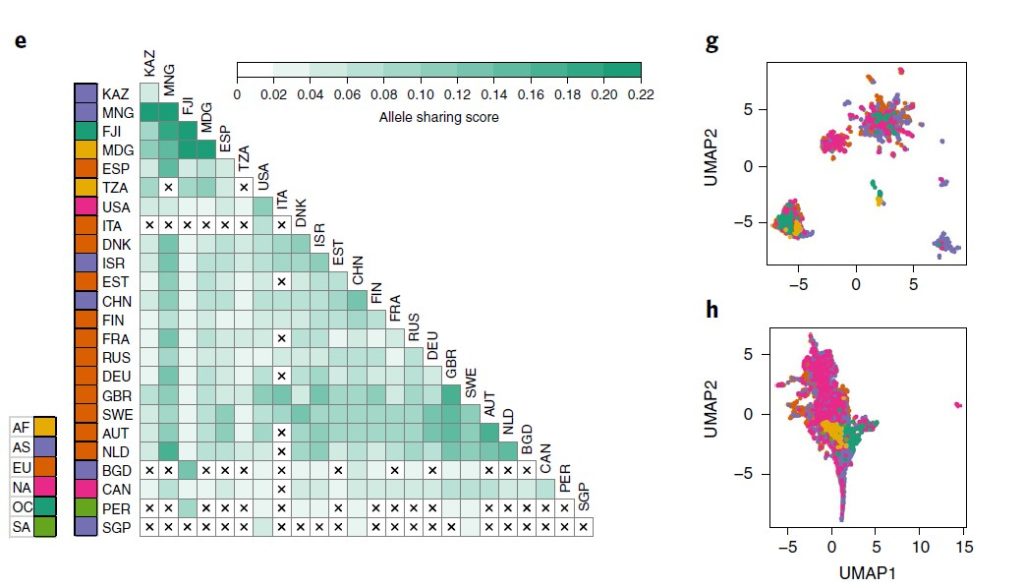
来自六大洲 31 个地点的 7,459 个肠道样本中发现的 881 个物种的 5180 万个 SNP的多个物种的种内遗传变异荟萃分析。
图e来自不同国家的宏基因组间的等位基因平均共享分数的热图。打叉单元格表示由于样本对不足(<5,000)而导致分数缺失。
图f为78 个常见物种的洲际种群分化分析(大陆内部与大陆之间的遗传相似性,用 F 统计检验测量亚种群 (FST) 中捕获的总遗传变异的比例)。
每个箱线图代表一个物种的洲际 FST 分布,按中位数排序。图g为通过直肠Agathobacter rectalis(物种ID 102492)的GT-Pro宏基因组基因型中的种内遗传变异捕获的地理模式的示例。
图h为为基于图g中相同样本的物种相对丰度的UMAP 分析。每个点都是一个宏基因组样本。颜色与图e示意一致。
结果表示,等位基因共享与工业化程度以及宿主关系明显关联;洲际种群间的分化程度有巨大差异,具有高FST的物种显示出明显的宿主集群,但不是所有宿主集群都与地理相关。
这与菌株在宿主中殖民的生活方式和环境的作用相一致。相比之下,在基于物种相对丰度的UMAP分析中,宿主间并没有明显集群,这表明宏基因组基因型可能揭示了在丰度分析中缺失的微生物生态学和微生物群落-宿主关系。
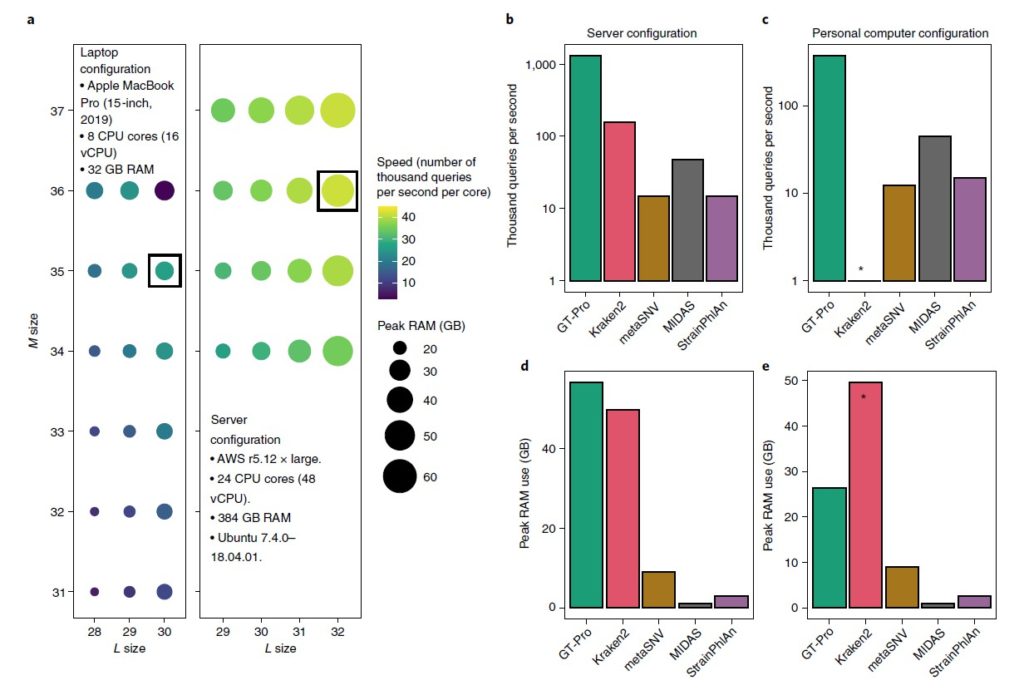
图a评估GT-Pro在笔记本电脑(左)和服务器环境(右)中的计算性能,以bits为单位。颜色表示处理速度,圆圈大小为RAM使用峰值。黑色方框表示最优状况。
图b-c为GT-Pro与metaSNV、MIDAS、StrainPhlAn、 Kraken2之间的速度比较,分别在服务器环境和笔记本电脑下比较。
图d-e为RAM使用峰值的比较,分别在服务器环境和笔记本电脑下比较。* 由于超出可用 RAM,Kraken2 无法在笔记本电脑中运行。
这些分析表明,与其他方法相比,GT-Pro 在服务器上大约快 8.5-570 倍,在笔记本电脑上快 8.3-163.6 倍。平均而言,处理每个宏基因组只需要在服务器上不到 4 秒,在笔记本电脑上大约需要 13 秒(平均为 497 万次读取)。虽然 GT-Pro 比其他方法更快,但它在服务器上需要 1.1-53.7 倍的 RAM和笔记本电脑上的 2.9-29.2 倍的 RAM(不包括内存不足的 Kraken2)。因此,只要计算机具有足够的 RAM,GT-Pro 数据结构和算法就可以极大地加速宏基因组分型。
研究人员在该文章中使用GT-Pro大约分析了2.5万个宏基因组,展示了GT-Pro是如何快速准确的识别SNP以及探索结构变异、种内遗传变异等。GT-Pro不使用基于比对的方法,而是类似于Kraken2,通过编码k-mers来快速检索,并适用于个人计算机或服务器环境。
但是它也不是完美的,目前GT-Pro存在的不足和如何应对:
第一,GT-Pro 数据库并未捕获所有人类肠道微生物多样性:但是通过基因组测序,会持续扩大SNPs的数量和涵盖的物种。
第二,GT-Pro 类似于基因分型阵列,因此不能识别新的 SNP,这需要其他方法,例如基于比对的宏基因组分型或单细胞基因组测序。
第三,由于基因组集合中存在高度相关的物种,少数物种缺乏物种特异性的 sck-mers。替代策略,例如使用更长的 k-mer 或不太常见的 SNP,可以对这些物种使用 GT-Pro 。
第四,尽管非常严格的挑选了用于构建GT-Pro的基因组和SNPs,但不可能完全排除错误(例如,不完整、污染和物种错误分类)。
最后,GT-Pro 不直接对结构变异进行基因分型。
“
考虑几个GT-Pro的未来发展方向,比如:
将GT-Pro与下游算法结合起来,以识别代表新微生物菌株的SNPs簇,或准确标记参考数据库中已知菌株的SNPs;
将GT-Pro的计算框架扩展到其他微生物环境中;为短插入缺失和结构变异开发无比对宏基因组分型;
将微生物组应用于精准医学,综合识别与疾病或其他特征(如致病性、抗菌耐药性、药物降解)相关的SNPs;
将GT-Pro用于检测污染、重组和跟踪变化,比如变异或菌株随时间、宿主生活方式和地理位置的变化。
主要参考文献
Shi ZJ, Dimitrov B, Zhao C, Nayfach S, Pollard KS. Fast and accurate metagenotyping of the human gut microbiome with GT-Pro. Nat Biotechnol. 2021 Dec 23. doi: 10.1038/s41587-021-01102-3. Epub ahead of print. PMID: 34949778.
谷禾健康
分类分析的研究,依赖于高质量的序列分类参考数据库,然而,目前已有记录公共序列数据库中出现错误,这些错误可能导致下游结果出错。不同的参考数据库对生物数据的分类结果差别很大,但缺乏客观评价单个数据库质量的标准。
有人选择自行构建特定于环境的数据库,但生成这样的数据库在技术上具有挑战性,导致了研究人员难以获取适当参考材料,或者对专有资源和服务有很大的依赖性。
为了满足可重复的生物信息学工作流程,以简化数据库生成和管理,来自阿肯色大学的Michael等人开发了一款新的工具——RESCRIPt. 该文章最近发表在《PLOS COMPUTATIONAL BIOLOGY》上。

RESCRIPt是一个独立的python3软件包,也是QIIME 2插件。用于参考序列分类数据库的可重复构建和管理,主要功能是格式化主流的公共数据库内序列用以自建分类数据库,由于处理步骤是透明化的,所以用户可以为不同的研究应用创建参考材料。
次要功能有评估、比较和交互探索参考数据库的定性和定量特征的功能。RESCRIPt使用QIIME 2文件格式,对每个处理步骤都生成专一的文件存储,使用户可以随时追溯任一计算步骤。
文章中,作者使用RESCRIPt对几个常用的16S rRNA基因、ITS和COI序列的参考数据库利用RESCRIPt进行了评估,并探讨了RESCRIPt目前存在的问题和未来的目标。
RESCRIPt处理和管理参考数据库的工作流程
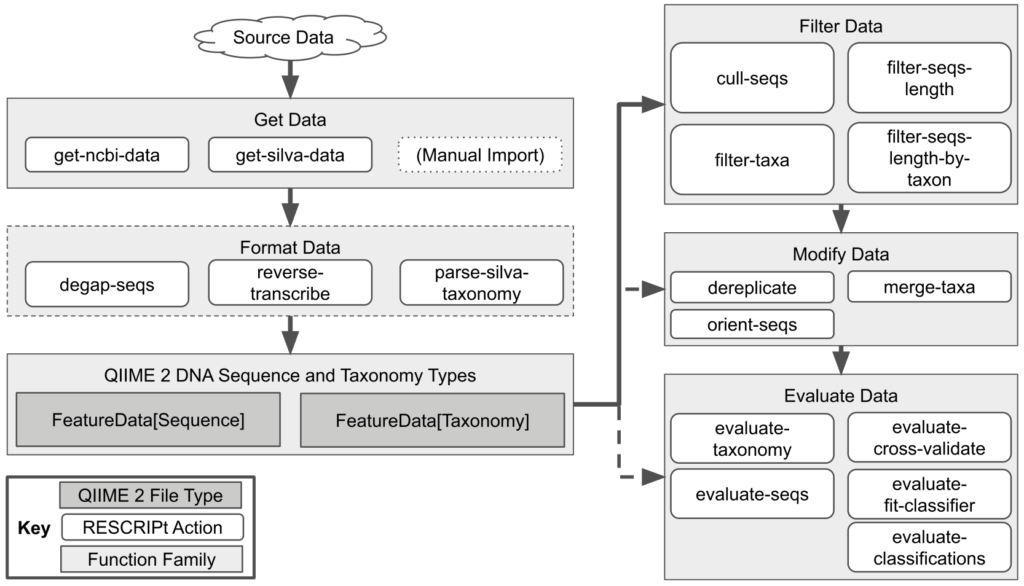
实线箭头表示建议的流程。虚线的箭头和边框表示自定义工作流程时的可选步骤。
RESCRIPt可以有效和透明的构建任何存在源数据的扩增子的参考数据库,以及来自NCBI的全基因组。
“Get Data”:获取源数据,可以直接从SILVA和NCBI GenBank数据库中自动下载序列和分类。
“Format Data”:格式化数据,包括基本的序列操作、逆转录和解析分类。
“Filter Data”:过滤数据,根据序列的质量或长度过滤以及根据分类和分类单元所在的序列长度过滤。
“Modify Data”:修改数据,去重复、合并分类或聚类。
“Evaluate Data”:评估, 对序列的一般质检,以及对分类准确率的评估。
详细的操作命令,见:
bokulich-lab/RESCRIPt: REference Sequence annotation and CuRatIon Pipeline (github.com)
RESCRIPt比较评估目前常用的四种16S rRNA基因数据库,分别为SILVA、Greengenes、GTDB和NCBI-RefSeq
从结果上看,在这些数据库中,SILVA数据库展示了最多的唯一序列和物种数,但是SILVA缺乏种水平的分类管理,其在种水平的分类准确率为0.73,远远低于其他16S rRNA基因数据库。相比之下,SILVA在属水平上的分类准确率要高得多。
NCBI-RefSeq的参考序列质量最高,分类准确率为0.94。
GTDB表现出略低的分类准确率0.92。
Greengenes13_8含有大量独特的序列和与SILVA相似的序列信息熵,但在属(54%)和种(90%)水平上有许多没被注释的序列。这表明该数据库中的大量序列在遗传上相似(≥98%),但在分类上是不同的,产生了不明确的标签。
各数据库的序列信息
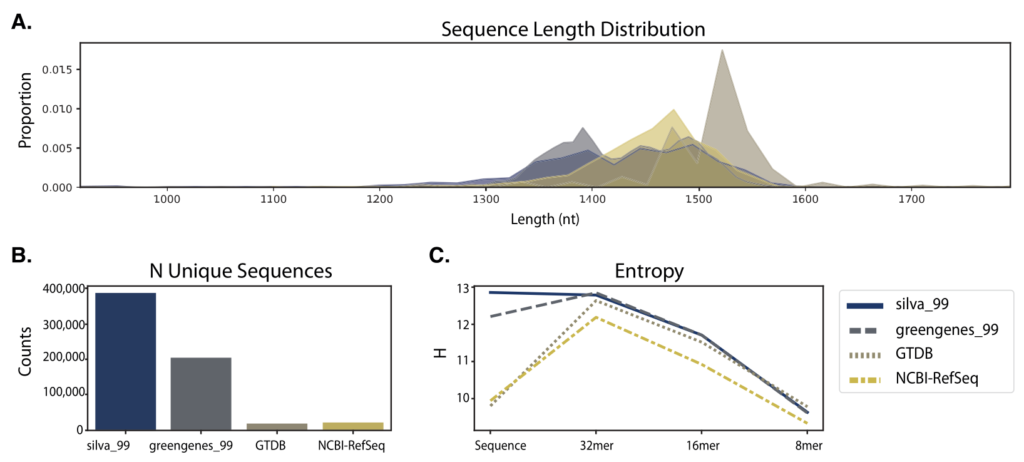
图A. 序列长度分布(去除异常值后);
图B. 每个数据库中唯一序列的数量;
图C. 每个数据库中全长序列和不同kmer长度的熵。
各数据库的分类信息和模拟分类的准确率比较
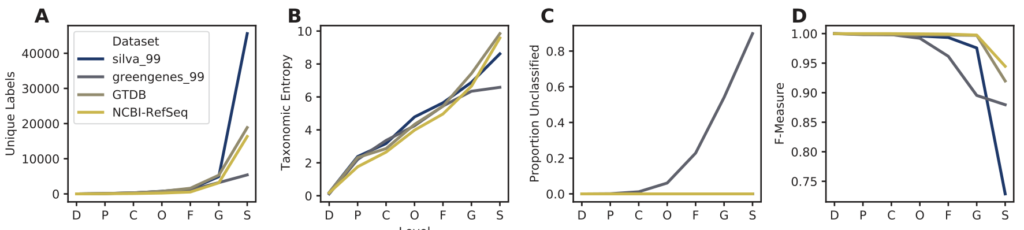
图A.唯一分类标签的数量。 图B.分类熵。
图C.在每一层级上未分类物种的比例。 图D.分类准确率。
横轴表示分类水平域门纲目科属种。
各数据库的分类覆盖率比较
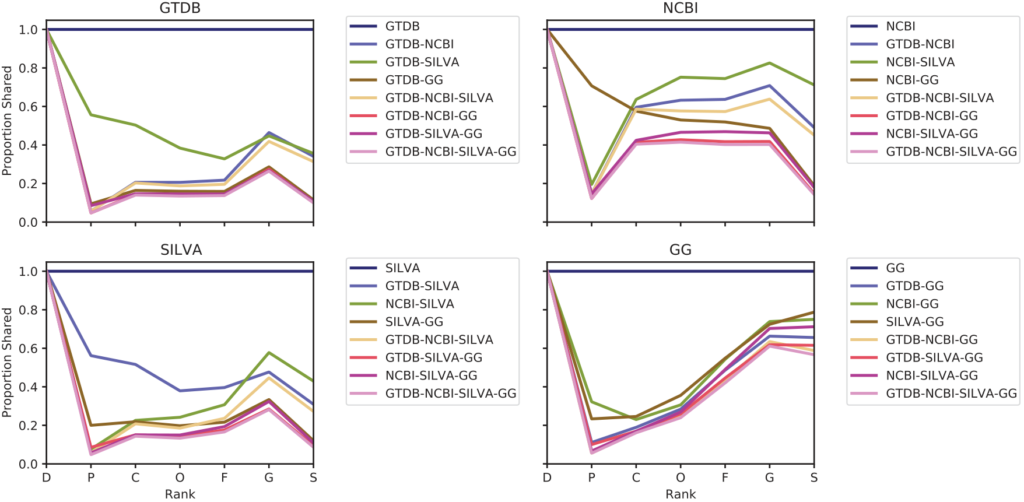
每张子图表示该数据库与其他数据库在每个分类水平上共享的分类群比例。图例指出了要相互比较的数据库。
RESCRIPt比较评估不同过滤步骤对16S rRNA基因SILVA数据库的影响
RESCRIPt使用get-silva-data命令获取SILVA序列和分类文件。“get-silva-data”命令允许选择下载哪个版本的数据库,是否下载LSU、SSU序列或SSU NR99序列,以及使用哪个分类水平和分类解析的选项等其它选项。
对16S rRNA基因SILVA数据库中每个连续序列使用不同RESCRIPt的质量过滤步骤后的序列信息比较
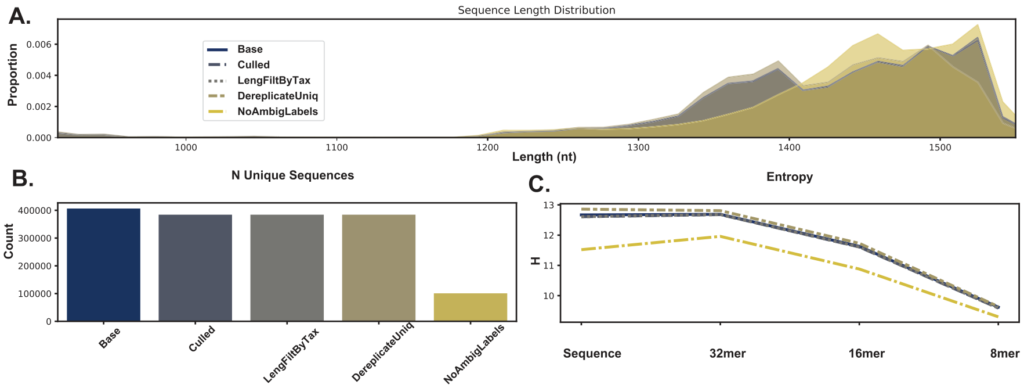
图A.序列长度分布。图B.唯一序列的数量。
图C.全长序列和不同kmer长度的熵。
图例中Base指完整的NR99 SILVA数据库;Culled指在序列中去掉8个或更多的均聚物(homopolymers)和/或5个具有歧义的碱基(ambiguous bases);
LengFiltByTax指基于分类学对数据进行序列长度过滤,即去除长度小于900 bp和小于1200 bp的古菌和细菌序列;
DereplicateUniq指使用“uniq”模式对分类和序列去重,即任何具有不同分类的相同序列将不会被合并;
NoAmbigLabels指任何与具有歧义的标签(通常在较低的分类级别) 相关的序列都从数据集中删除。
结果表示Culled和LengFiltByTax步骤对序列的影响是有益的,而NoAmbigLabels方法会过多丢失序列信息。
各过滤步骤下序列分类信息和模拟分类准确率的比较
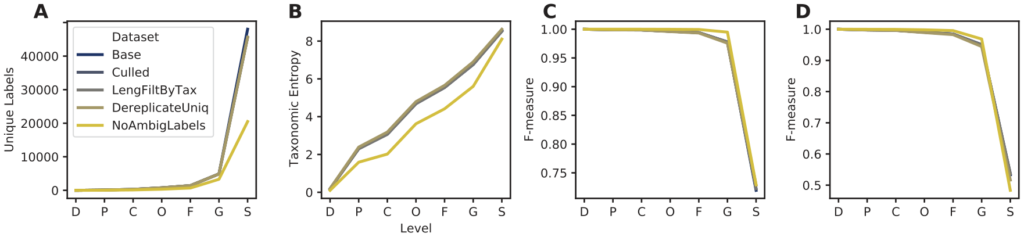
图A. 唯一分类标签的数量。图B.分类熵。
图C. 无需交叉验证的最佳分类准确率(当真实标签已知,但分类准确率可能被数据库中其他类似的命中混淆时,模拟可能的最佳分类准确率)。
图D. 使用交叉验证的分类准确率(在不知道正确标签的情况下模拟真实的分类任务)。
横轴表示分类水平域门纲目科属种。除了NoAmbigLabels的分类注释外,质量过滤对分类准确率的影响微乎其微。
RESCRIPt评估在多个OTU%相似性阈值下聚类的Greengenes数据库(13_8版本)的多个数据库质量特征
结果表示相似性阈值的降低导致了信息丢失,在属和种水平上,唯一分类标签的数量迅速减少。相反,相似性阈值的增加使得分类准确率上升。
这表明,即使选择了认为合适的相似度阈值也可能对数据库的信息内容和分类准确率产生负面影响。但作者还是建议不要在任何标记基因序列数据库中使用相似度<99%的OTU聚类。
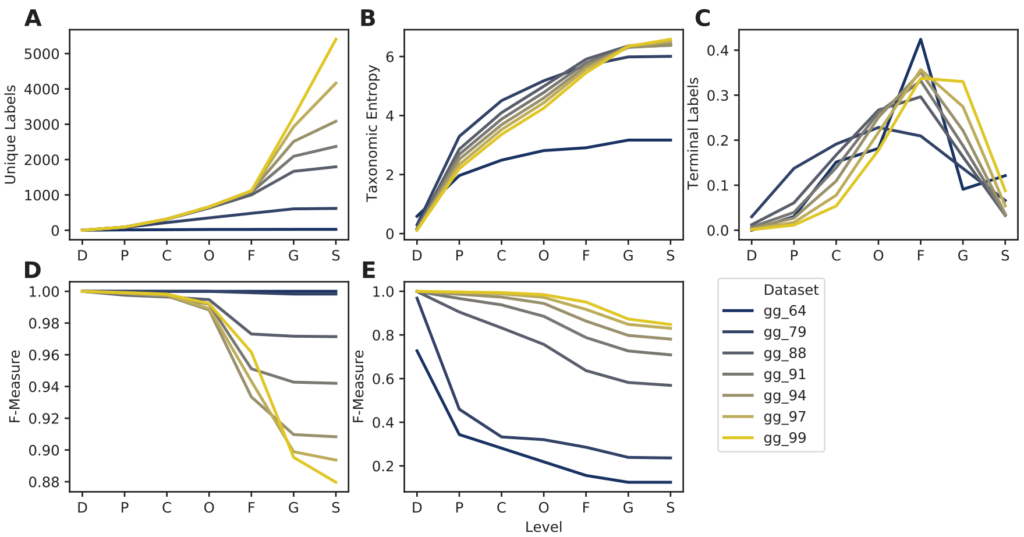
图A. 唯一分类标签的数量。 图B.分类熵。
图C. 在每一层级里分类单元的数目。
图D. 无需交叉验证的最佳分类准确率(当真实标签已知,但分类准确率可能被数据库中其他类似的命中混淆时,模拟可能的最佳分类准确率)。
图E. 使用交叉验证的分类准确率(在不知道正确标签的情况下模拟真实的分类任务)。
横轴表示分类水平域门纲目科属种。图例指示不同的OTU%相似性阈值。
RESCRIPt评估不同处理步骤下的UNIT ITS真菌序列数据库
结果表示OTU聚类方法里,97%比99%比动态聚类,对结果的影响最小。含所有真核生物的数据库所包含的序列是仅含真菌序列数据库的两倍多,但其分类准确率是最低的。
而只含目水平或更低级别分类水平的真菌序列数据库在分类准确率上提升最大。
对UNIT ITS数据库的三种类型UNIT_97,UNIT_99,UNIT_dynamic数据库分别进行划分
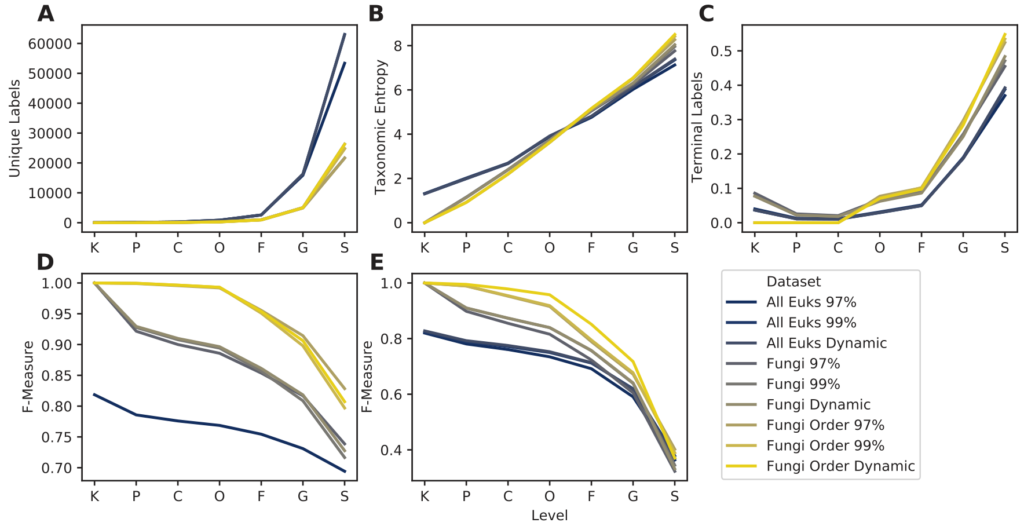
Euks表示含所有真核生物序列,Fungi表示只含真菌序列,Fungi Order表示只含目水平或更低级别分类水平的真菌序列。
图A. 唯一分类标签的数量。 图B. 分类熵。
图C. 在每一层级里分类单元的数目。
图D. 无需交叉验证的最佳分类准确率(当真实标签已知,但分类准确率可能被数据库中其他类似的命中混淆时,模拟可能的最佳分类准确率)。
图E. 使用交叉验证的分类准确率(在不知道正确标签的情况下模拟真实的分类任务)。横轴表示分类水平域门纲目科属种。
RESCRIPt评估用于后生动物分类鉴定的COL基因数据库
首先比较评估了不同序列处理步骤下的BOLD COL基因数据库(BOLD全称Barcode of Life Data Systems)。
结果表示聚类序列大大减少了未修剪和引物修剪的BOLD COI数据集中唯一序列的数量,经引物修剪也会降低唯一序列的数量。且在种水平上表现最明显。聚类和引物修剪也降低了分类准确性。数据表明OTU聚类不利于COI基因分类。
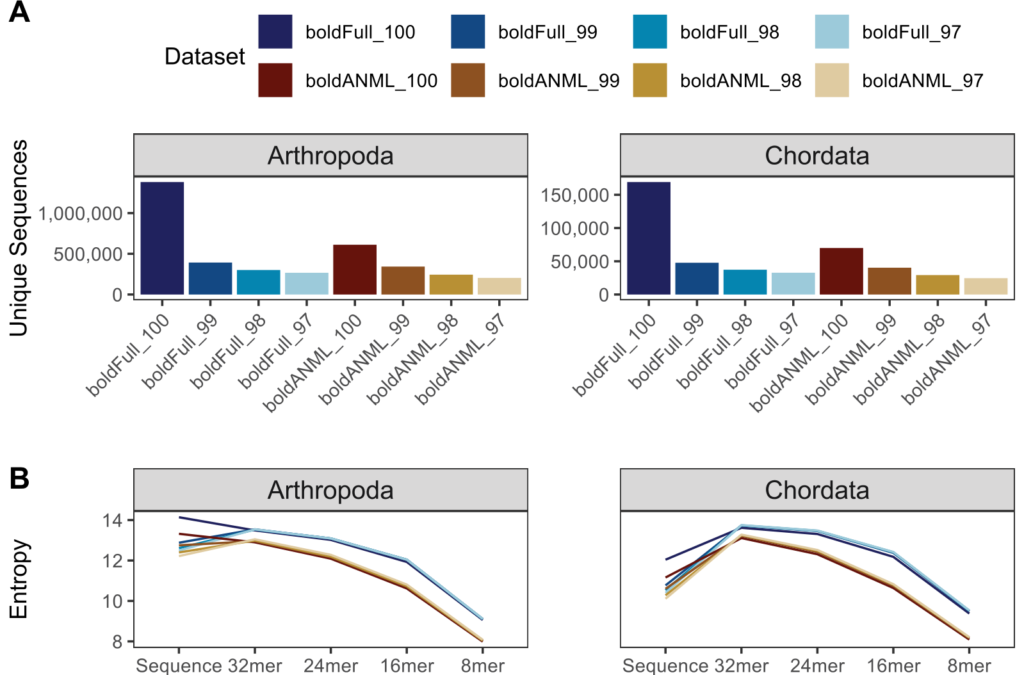
图例中Full表示未修剪的全长序列,ANML表示经引物修剪后的序列,后边接的数字表示相似性聚类阈值。Arthropod指节肢动物,chordate指脊索动物。图A.唯一分类标签的数量。图B.不同kmer长度的分类熵。横轴表示不同数据库。
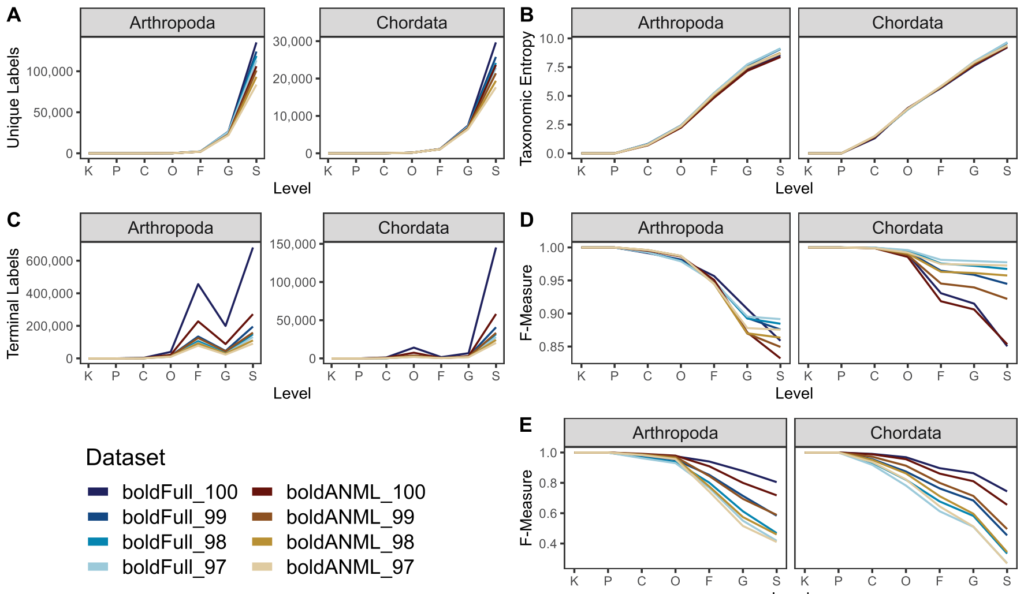
图A.唯一分类标签的数量。图B.分类熵。
图C.在每一层级里分类单元的数目。
图D. 无需交叉验证的最佳分类准确率(当真实标签已知,但分类准确率可能被数据库中其他类似的命中混淆时,模拟可能的最佳分类准确率)。
图E. 使用交叉验证的分类准确率(在不知道正确标签的情况下模拟真实的分类任务)。横轴表示分类水平域门纲目科属种。
其次评估比较了从BOLD或NCBI GenBank获得的去重复和引物修剪的COL基因数据库。
数据表明,整体看NCBI的唯一序列较少,但局部看,NCBI在属水平和种水平上有更多唯一序列。从分类准确率看,NCBI相对于BOLD,从科到种水平都有提高。
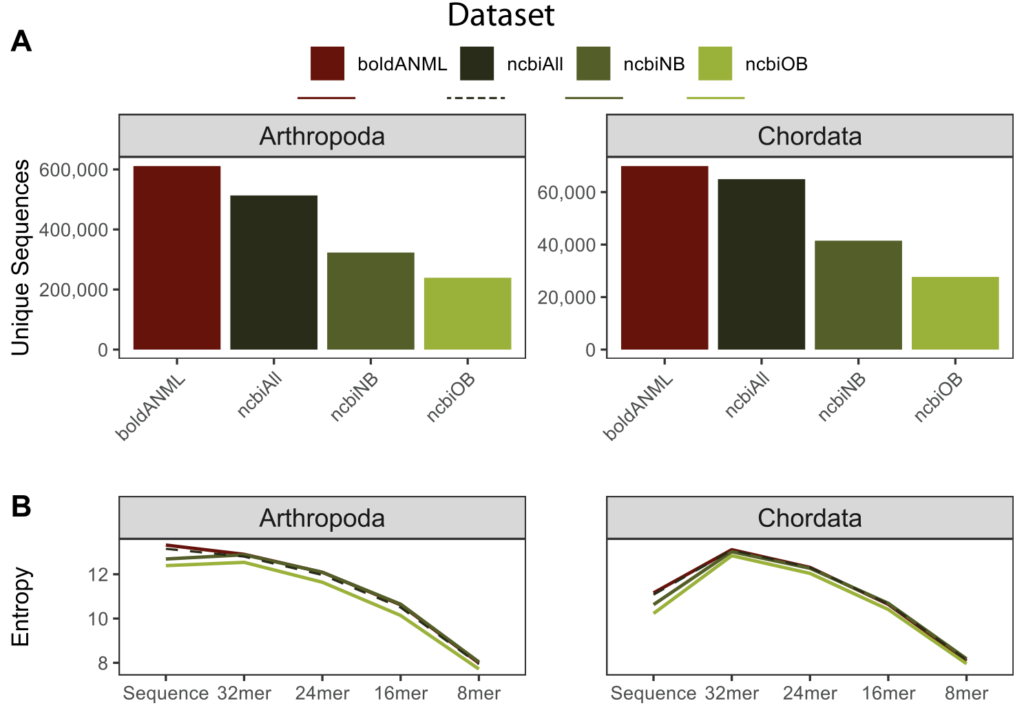
数据集分别为boldANML(BOLD COL基因数据库)、ncbiAll(ncbiNB与ncbiOB的集合)、ncbiNB(不含BOLD COL基因序列的NCBI GenBank COL基因数据库)、ncbiOB(含BOLD COL基因序列的NCBI GenBank COL基因数据库)。图A.唯一分类标签的数量。图B.不同kmer长度的分类熵。横轴表示不同数据库。
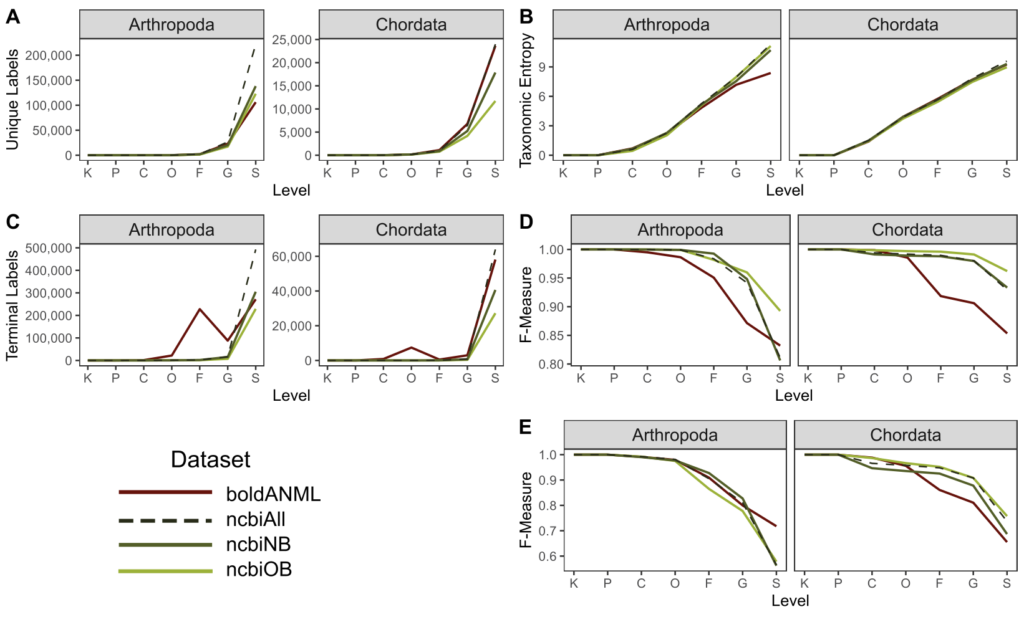
图A. 唯一分类标签的数量。 图B. 分类熵。
图C. 在每一层级里分类单元的数目。
图D. 无需交叉验证的最佳分类准确率(当真实标签已知,但分类准确率可能被数据库中其他类似的命中混淆时,模拟可能的最佳分类准确率)。
图E. 使用交叉验证的分类准确率(在不知道正确标签的情况下模拟真实的分类任务)。横轴表示分类水平域门纲目科属种。
RESCRIPt旨在为研究人员提供可重现的核苷酸序列和分类学数据库生成、整理和评估的工具。它不是一个数据源,也不是分类学、系统学或数据质量方面的权威,并且RESCRIPt生成的评估结果也不是质量或准确性的可靠指标。
与任何生物信息学方法一样,RESCRIPt输出的质量取决于其输入的质量和用户作出的处理决策。一般来说,用户应该使用多个指标来指导他们对RESCRIPt结果的解释,但在对数据库质量作出结论之前,还需要了解输入数据的组成。
RESCRIPt目前的版本已经兼容宏基因组数据库。未来将计划提供更多的基因组和宏基因组功能。例如用于(元)基因组距离估算的ANI和MASH方法,以及用于(元)基因组数据库分类精度估算的方法。会增加从学界里常用的公共在线数据库中获取序列和分类的方法。
RESCRIPt作为一个Python3软件包和QIIME 2插件,可以用conda安装也可以docker运行,或者在已有的qimme2环境中安装。
通过RESCRIPt工具可以独立完成序列的获取、修剪、过滤、去重、聚类,整合为数据库,并且可以对多个数据库进行评估比较。每个处理步骤会有独立的日志文件生成和中间文件生成,便于溯源和重现该流程。只是庞大的数据库和庞大的功能在计算资源消耗这方面肯定不容小觑,虽然文章中没有提及这方面的内容,但作为使用者不能忽视。
关于安装和测试使用还是要仔细阅读官方手册,地址:
参考文献:
Robeson MS 2nd, O’Rourke DR, Kaehler BD, Ziemski M, Dillon MR, Foster JT, Bokulich NA. RESCRIPt: Reproducible sequence taxonomy reference database management. PLoS Comput Biol. 2021 Nov 8;17(11):e1009581. doi: 10.1371/journal.pcbi.1009581. PMID: 34748542; PMCID: PMC8601625.

谷禾健康

在细菌全基因组测序数据分析中,区域(regional)和功能注释已经成为常规操作。一个完整的基因组注释是下游分析的基础,而注释的准确性和全面性往往影响研究结果。
最近来自德国一个生物信息学团队们开发了Bakta,一款新的命令行工具,用于自动化和标准化的细菌基因组注释。该文章最近在《MICROBIAL GENOMICS》公开发表。

作者认为现有的各种注释软件工具都在以下问题留下了改进的空间:
1. 尽管早在20年前就发现了以前被忽视的保守的短ORF(sORF),但它们既不能预测也不能检测到短于29个氨基酸的小蛋白的编码序列(CDS),因为在基因预测工具中实施了基因长度截断,以减少错误的从头预测的数量。
2. 它们不识别存储在公共数据库(如RefSeq和UniRef100)中的已知蛋白质序列,因此不能分配数据库交叉引用(dbxref),即稳定的公共数据库标识符,方便与更详细的数据库互连。
3. 对于跨越人工序列边的CDS结构注释,没有考虑附加的序列信息,即完整性和拓扑结构。
为了解决这些问题,团队们开发了Bakta。它为编码和非编码基因提供了全面的注释工作流程,并加之CRISPR阵列、gaps、oriC和oriT特征的预测。与其他轻量级注释管道不同,Bakta能够通过自定义的sORF提取和过滤步骤来检测和注释小分子蛋白。而CDS注释流程通过一种基于哈希的、无需比对的蛋白质序列识别方法。
注:CRISPR,clustered regularly interspaced short palindromic repeats
此外,这种新的方法便于通过稳定的标识符交叉引用公共数据库来标注CDS。
软件工作流程如下图。
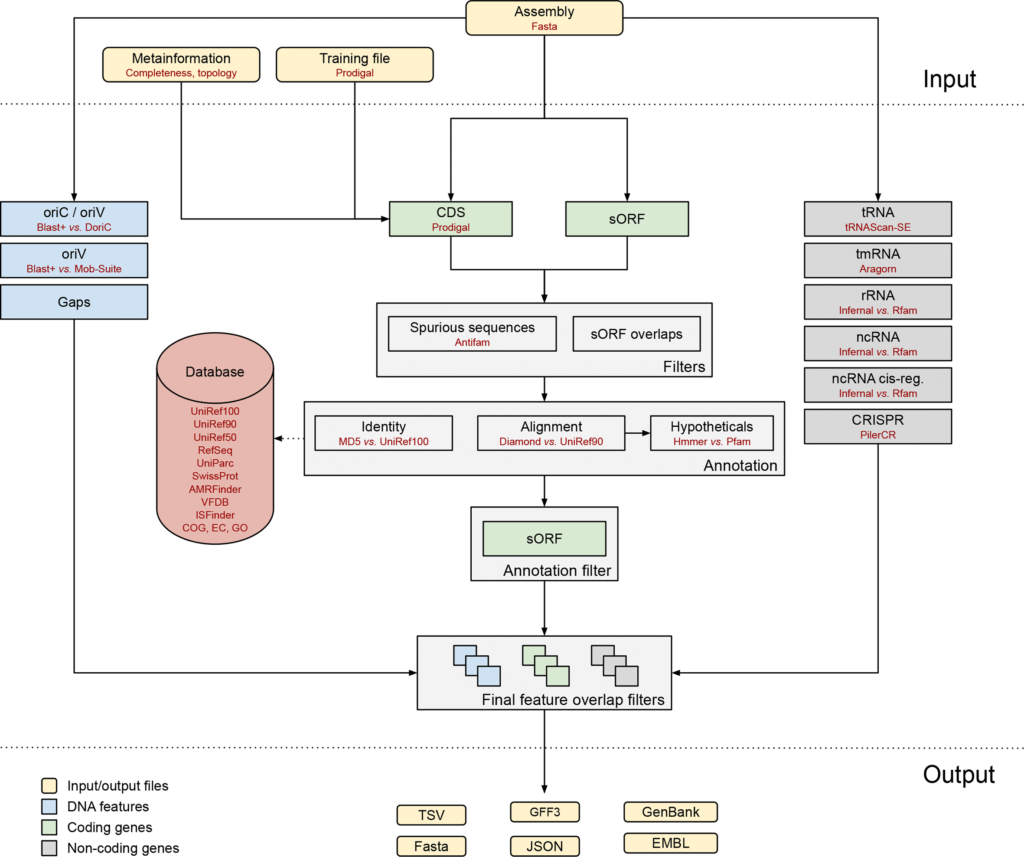
输入文件为fasta格式的基因组组装序列。
可选择输入序列元数据文件或Prodigal软件提供的training文件。非编码基因,比如tRNA使用tRNAscan-SE预测和注释。
gaps、oriC和oriT等特征的预测用BLAST+工具。利用Prodigal预测CDS,使用BioPython提取短于30个氨基酸的小蛋白的sORF。
HMMER和AntiFam分别过滤假阳性序列和重复的sORF。为了加快对CDS和sORF的注释,使用无比对序列鉴定(AFSI),即通过全长蛋白序列MD5哈希算法和相关蛋白质序列长度检查进行鉴定的组合过程。使用Diamond和UniRef90比对识别剩余的未识别蛋白质序列。
Bakta有自建的SQLite数据库,用于识别查找UniRef100, UniRef90、UniRef50、RefSeq、COG、EC 、GO、耐药基因、VFDB等。对于还是没有明确注释的CDS标记为假设蛋白(Hypotheticals),通过HMMER使用Pfam HMM图谱筛选蛋白质结构域。
通过与其它软件工具进行基准测试,评估Bakta的性能。
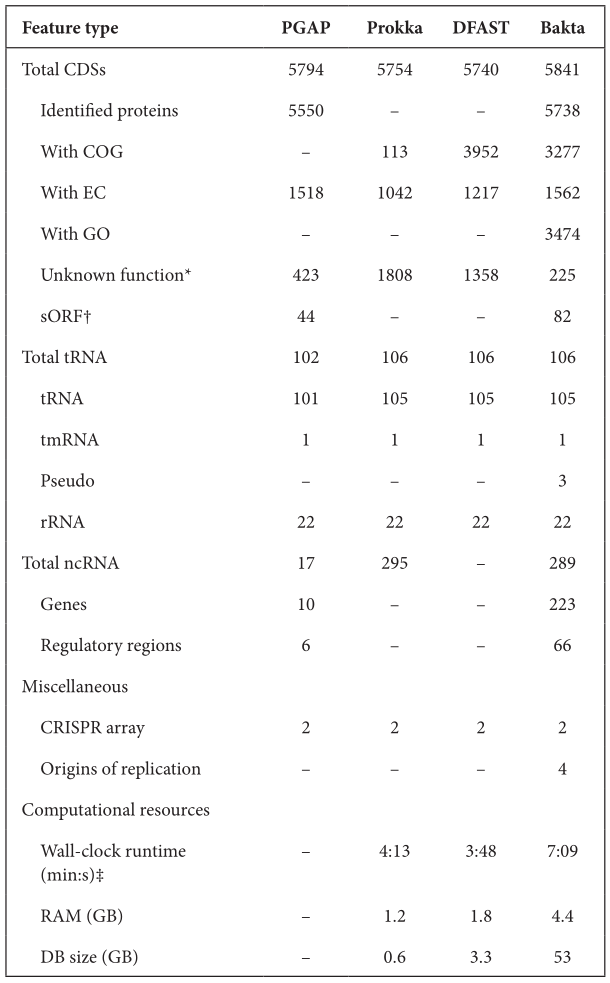
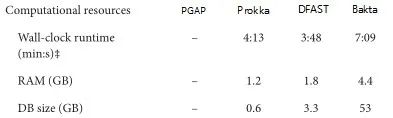
首先是注释得到的特征结果之间的比较。作者选择E. coli O26 : H11 strain 11368菌株的基因组,分别使用Prokka、DFAST、PGAP与Bakta比较,如下表。对于CDS,PGAP和Bakta预测到更多的基因。在CDS序列的功能注释方面,PGAP和Bakta表现最好,且Bakta是唯一一个分配到GO术语的工具。

其次是功能注释的性能基准测试。选择来自RefSeq的35个不同分类的细菌基因组进行注释。统计其假设蛋白占总CDS的比例,如小提琴图。
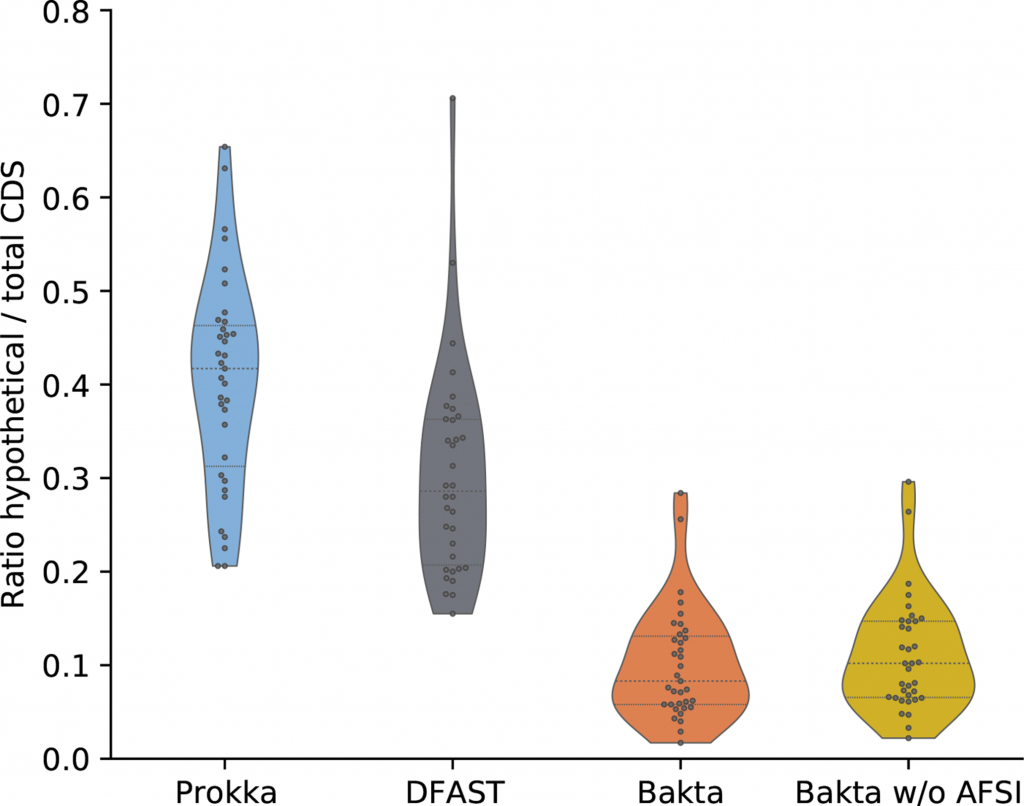

同时统计了在没有用AFSI(Bakta w/o AFSI,只是用Diamond比对序列)和使用AFSI的假设蛋白占总CDS的比例,两者之间的差异只有0.9%。
由此得出,AFSI对RefSeq中检测到的小蛋白的功能注释贡献很小。表格中展示了Bakta检测到的小蛋白参与的一系列与致病性高度相关的重要过程,以及更一般的细胞内务管理过程。
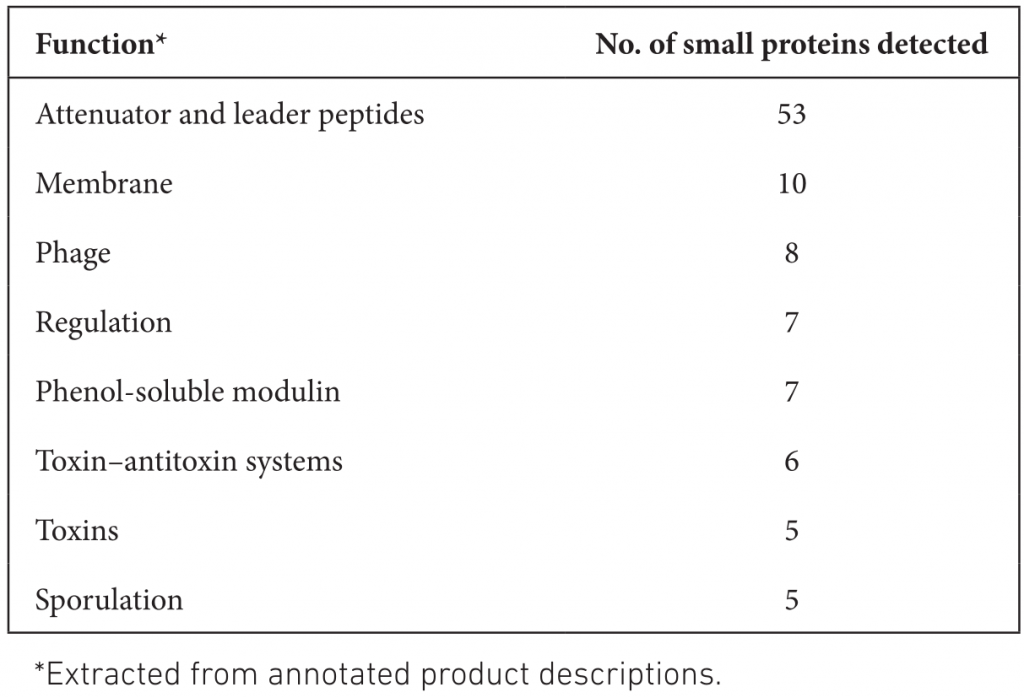

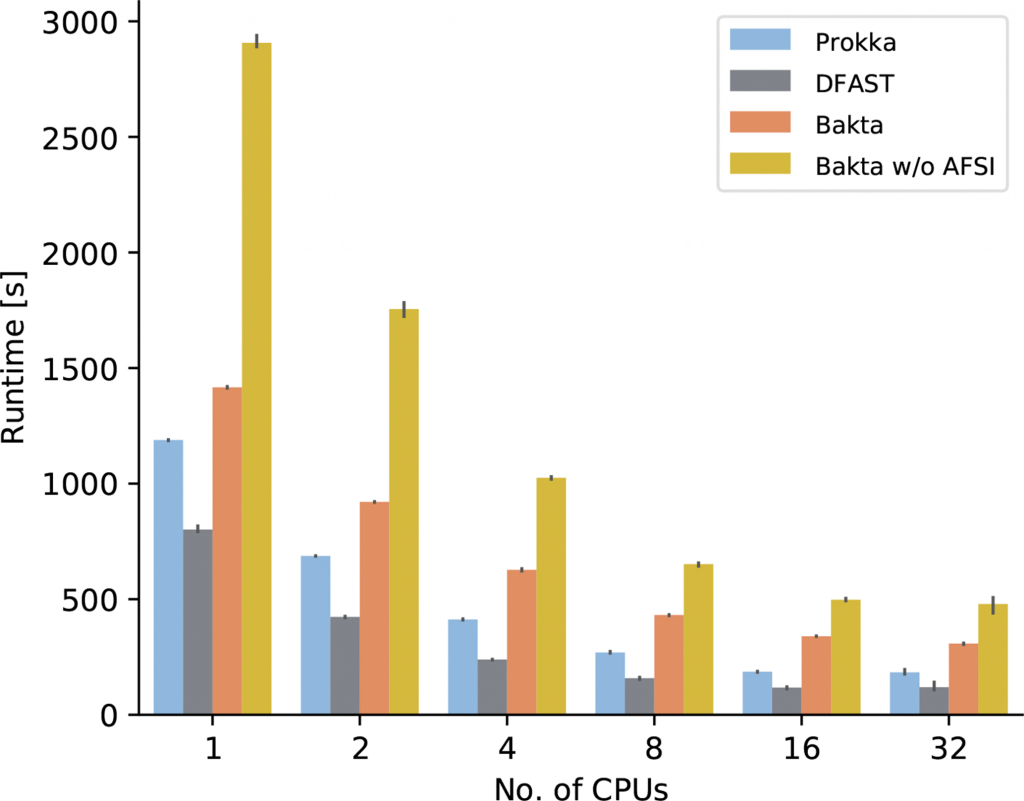
最后比较了Bakta的运行时间、内存消耗和存储需求。在具有4个Intel Xeon E5-4627CPU和总共40个核的服务器机器上,使用不同数量的CPU连续三次测量注释E.coli O26:H11 strain11368的PROKKA、DFAST、Bakta和不使用AFSI的Bakta的运行时间。
结果如下图和表格。Bakta虽然运行时间时最慢的,但它所包含的数据库内容是最多的,其分析深度也有很大提高。对比没有使用AFSI的Bakta,在同等条件下,使用AFSI大大提高了序列注释的速度。

Bakta可以对宏基因组的MAG进行注释,在与DFAST和Prokka的比较中,Bakta依旧是假设蛋白占总CDS比例最低的;注释结果格式符合INSDC标准;在线版Bakta应用程序,提供交互式GUI向导,可输入数据与命令行工具一样,适合不太熟悉命令行操作的研究员,地址:bakta-web-ui (computational.bio)
通过以上的工作流程介绍和性能评估,该软件有如下优势:
1 Bakta在已知和未知物种的分类范围内对CDS序列的功能注释方面优于现有工具
2 Bakta能够检测和注释当代预测工具无法预测的小蛋白,比如在使用Prodigal和MetaGeneAnnotator工具预测时
3 Bakta能够精确识别已知的蛋白质序列,并分配RefSeq和UniProt数据库标识符
4 新的AFSI方法加速了Bakta的功能注释工作流程
5 Bakta利用序列元数据改进了CDS的结构预测
6 Bakta以功能类别(COG、EC和GO)为CDS提供了同等或更全面的注释。
目前看来,较为明显的缺点就是运行时间长,虽然提供了Web版本,但如果样本数量较大,还是需要在linux上运行。
参考文献:
Schwengers O, Jelonek L, Dieckmann MA, Beyvers S, Blom J, Goesmann A. Bakta: rapid and standardized annotation of bacterial genomes via alignment-free sequence identification. Microb Genom. 2021 Nov;7(11). doi: 10.1099/mgen.0.000685. PMID: 34739369.
谷禾健康
人类微生物组的变化与许多疾病和健康状况有关。然而,看懂人类微生物组研究结果的报告具有挑战性,因为它通常涉及微生物学、基因组学、生物医学、生物信息学、统计学、流行病学等领域的方法。
人类微生物组的研究与其他类型的分子流行病学研究具有许多共同特征,但它们也需要独特的考虑因素,具有自己的方法学最佳实践和报告标准。除了流行病学研究设计的标准要素外,独立于培养的微生物组研究还涉及生物标本的收集、处理和保存;不断发展的实验处理方法,具有更高的批次效应潜力;生物信息学处理;稀疏、异常分布、高维数据的统计分析;报告可能的数千种微生物特征的结果等。
由于微生物组研究没有公认的金标准方法,而且该领域还没有就这些方面形成共识,但是各个领域的人员共同努力逐渐形成适用于广泛的人类微生物组研究报告的规范流程是微生物领域快速发展的基础。
参 与 者
对研究参与者的描述中,应描述入组的人群,以及如何从人群中抽取参与者。
参与者的特征,例如环境、生活方式行为、饮食、生物医学干预、人口统计和地理都可能引起微生物组的显着差异,因此应该包含这些基本描述。
时间背景也很重要,因此应说明招聘、跟进和数据收集的开始和结束日期。
此外,还应包括用于评估潜在参与者是否符合研究资格的具体标准,包括纳入标准和排除标准的详细信息。纳入和排除标准是用于选择研究参与者的预先确定的特征,描述这些标准对于了解研究的目标人群至关重要。
应描述收集的有关可能影响微生物组的抗生素或其他治疗的任何信息,以及是否有任何排除标准包括最近使用抗生素或其他药物。
应说明最终的分析样本量,以及在招募、随访或实验室过程的任何步骤中排除参与者的原因。
建议使用流程图来显示参与者被排除在研究之外的时间和原因。例如如下流程说明。

Mirzayi C, et al., Nat Med. 2021
如果参与者在纵向研究中失访或未完成所有评估,则应说明如何进行随访的详细信息,并应报告特定时间点的样本量。
此外,将病例与对照进行匹配的研究应描述在匹配中使用了哪些变量。
实 验 方 法
应描述实验室样品的处理,包括样品采集、运输和储存的程序。
由于 DNA 提取可能是跨研究技术差异的主要来源,因此应描述 DNA 提取方法。如果进行了人类 DNA 去除和微生物 DNA 富集的描述,也应包括在内。同样,如果使用阳性对照、阴性对照或污染物减轻方法,则应对其进行识别和描述。
应描述报告与测序相关的方法。这包括引物选择和 DNA 扩增(包括16S rRNA 基因可变区,如果适用)。测序完成的主要单位(公司或者检测机构),例如鸟枪法或扩增子测序。最后,应解释用于确定相对丰度的方法。
批次效应应作为潜在的混杂来源进行讨论,包括为确保批次效应不与暴露或感兴趣的结果重叠而采取的步骤。如果进行宏转录组学、宏蛋白质组学或代谢组学,应提供这些方法的详细信息。
数 据 源 / 测 量
对于非微生物组数据(例如,健康结果、参与者的社会经济、行为、饮食和生物医学特征,包括疾病位置和活动以及环境变量),应描述每个变量的测量和定义。例如,参与者的性别和年龄可以从电子病历或分发给参与者的问卷中获得,那么应该清楚描述这个数据源的获得方式。还可以讨论测量的局限性,包括由于错误分类或丢失数据导致的潜在偏差,以及为解决这些测量问题所做的任何尝试。
因果推断的研究设计注意事项
在没有直接观察到假设的因果关系的情况下,观察数据通常用于测试旨在进行因果推断的关联。
方法包括,例如,使用多变量分析或匹配来调整假设暴露(例如微生物分类群的丰度)与研究中的疾病或病症之间的混杂变量。混杂因素可以被认为是暴露和研究结果的常见原因,可以导致暴露和结果之间的虚假关联。例如,年龄可能是一个常见的混杂因素,因为它会影响微生物组和大多数健康结果的风险。
如果不采取措施避免批次间条件的不平衡,实验室批次效应也可能混淆微生物组与感兴趣条件之间的关系。试图控制测量混杂的常用方法是调整或分层混杂。应为因果推断的回归模型中包含或排除的变量提供理由,因为对非混杂变量进行调整或分层会引入偏差。作为这一理论论证的一部分,作者应考虑包括一个有向无环图,显示假设的感兴趣的因果关系。
除了考虑本研究的理论动机外,还应讨论可能会扭曲微生物组与感兴趣变量之间观察到的关系的选择或生存偏差的可能性。例如,这种偏倚可能是由于失访(在纵向研究中)或由于疾病本身而没有将参与者纳入研究(例如,死于侵袭性结直肠癌和还没有幸存下来,无法参与结肠直肠癌微生物组的假设研究)。检查表中其他地方的其他项目可能与因果推断问题直接相关,包括假设、研究设计、匹配、偏倚和普遍性。鼓励调查因果问题的作者在因果推断的背景下考虑他们对这些项目的报告。
生物信息学和统计方法
对生物信息学和统计方法的充分描述对于生成严谨且可重复的研究报告至关重要。
应描述数据转换(例如标准化、稀疏和百分比)。应充分披露质量控制方法,包括过滤或删除读数或样本的标准。应说明用于分析数据的所有统计方法,包括如何选择感兴趣的结果(例如,使用P值、q值或其他阈值)。
应详细描述分类、功能分析或其他序列分析方法。为了重现性,所有用于数据预处理和分析的软件、软件包、数据库和库都应该被描述和引用,包括版本号。
可重复的研究
可重复的研究实践作为出版过程中的质量检查以及进一步的透明度和知识共享,如 Schloss 提出的标题中所详述。期刊越来越多地实施可重复的研究标准,包括数据和代码的发布,并且在可能的情况下应遵循这些指南。
如果可能,原始数据和处理过的数据,应存放在独立维护的公共存储库中,这些存储库可提供长期可用性,例如由 NCBI 或 EMBL-EBI 维护的公共存储库。Zenodo ( https://zenodo.org/ ) 或 Publisso (https://www.publisso.de/en/ ) 可用于为处理后的数据集提供 DOI。
如果数据或代码不公开或不能公开,即使在提供限制访问选项的存储库中,也应提供感兴趣的读者如何访问数据的描述。至少应描述任何受保护的信息,以及如何访问此类数据。
描 述 性 数 据
应报告关于研究人群的描述性统计数据。至少,应描述研究人群的年龄和性别,共享数据文件中应包括每位参与者的年龄和性别,但应尽可能报告其他重要的参与者特征,包括药物使用或生活方式因素,例如饮食。
作者应考虑在描述性统计表中如何报告这些数据。例如R 软件中的 table1 包等包,使创建这样的表不那么复杂。
结 果 数 据
研究的主要结果应该是详细的,包括描述性信息、感兴趣的发现和任何额外分析的结果。
应为每个组和每个时间点报告描述性微生物组分析(例如,降维如主坐标分析、多样性测量和总分类组成)。
这应为读者提供了差异丰度分析的结果。当报告差异丰度测试结果时,应明确说明每个可识别的标准化分类单元的差异丰度的大小和方向。其他类型分析的结果,如代谢功能、功能潜力、MAG 组装和 RNA-seq,也应在结果中描述。
附加结果(例如,非显著结果或完全差异丰度结果)可以包含在补充中,不应完全排除。
虽然这个问题已经存在了几十年,许多领域的期刊都认识到发表偏倚的问题,但在出版物中包含此类结果将有助于降低这种偏倚的严重程度,并改进未来的系统评价和荟萃分析。
讨论应包括对本研究和相关方法的局限性的讨论。应讨论偏差的可能性以及它们将如何影响研究结果。
许多形式的偏倚,例如残差/未测量混杂、与成分分析相关的偏倚、测量偏倚或选择偏倚,都可能影响对研究结果的解释,在讨论中承认潜在的偏倚来源很重要。
还应考虑研究发现的普遍性,以及这些发现是否适用于目标人群或其他人群。如果不同形式的偏见没有被评估或假设,可以忽略不计,但应说明这一点。
主要参考文献
Mirzayi C, Renson A; Genomic Standards Consortium et al. Reporting guidelines for human microbiome research: the STORMS checklist. Nat Med. 2021 Nov;27(11):1885-1892. doi: 10.1038/s41591-021-01552-x.
Wirbel, J. et al. Meta-analysis of fecal metagenomes reveals global microbial signatures that are specific for colorectal cancer. Nat. Med. 25, 679–689 (2019).
Simoneau, J., Dumontier, S., Gosselin, R. & Scott, M. S. Current RNA-seq methodology reporting limits reproducibility. Brief. Bioinform. 22, 140–145 (2021).
Ten Hoopen, P. et al. The metagenomic data life-cycle: standards and best practices. Gigascience 6, 1–11 (2017). – PubMed – PMC
Yilmaz, P. et al. Minimum information about a marker gene sequence (MIMARKS) and minimum information about any (x) sequence (MIxS) specifications. Nat. Biotechnol. 29, 415–420 (2011). – PubMed – PMC

谷禾健康

编辑
微生物组和组学数据集,由于其生物学性质,通常是高维的,特征常以各种成分,如基因、OTU、RNA转录本等的计数为特征。这些数据统称为成分数据。
这类数据分析的中心概念是对数转换,而其中最简单的策略是ALR(Additive log ratio)方法。对于高维数据,ALR方法有一下几个特点:
(a) 次要成分都是相干的
(b)可以解释100%的总对数方差
(c)测量结果非常接近于等距。
最近,来自西班牙科学团队的一篇题为“Compositional Data Analysis of Microbiome and Any-Omics Datasets: A Validation of the Additive Logratio Transformation” 的文章指出:
ALR对数转换可以有效提供一组简单的变量来表示整个成分数据集,其关键节点在于选择哪个成分为参考,并使用三个高维组学数据集进行验证。
通过ALR方法的理论和推导公式(这里不详述,推荐看原文),分别计算总对数方差(The total logratio variance 总结了采样点在多维空间中的分散程度),Logratio Geometry和Procrustes分析,以此找到有效的参照特征。再与其它对数转换方法对比,如CLR对数转换。
1. 兔子数据集
数据集为非零数据集,89个样本,3937个特征。
总对数方差为0.1601,Procrstes相关系数最高为0.9991,对应的基因数为856。该基因在3937个基因中的相对丰度排名第201位。
图一为所有3937个特征的Procrstes相关性直方图。为了直观地显示ALR变量接近等距的程度。
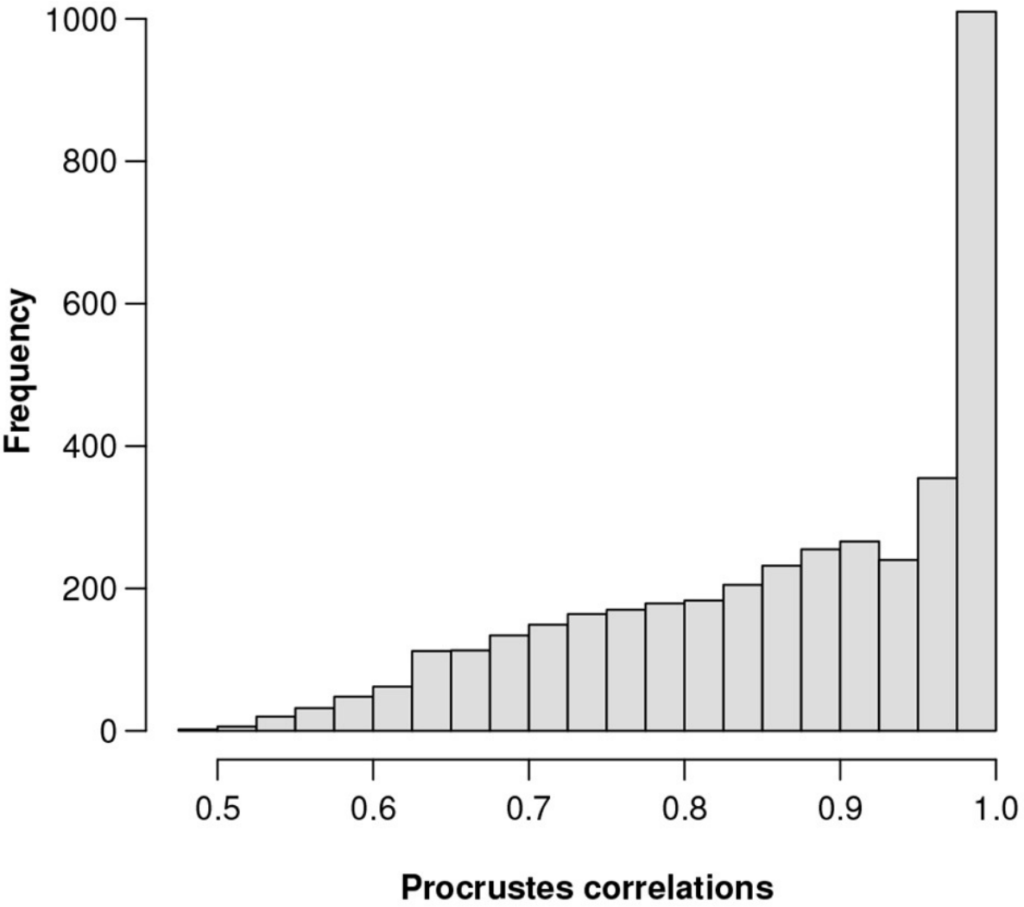
图一
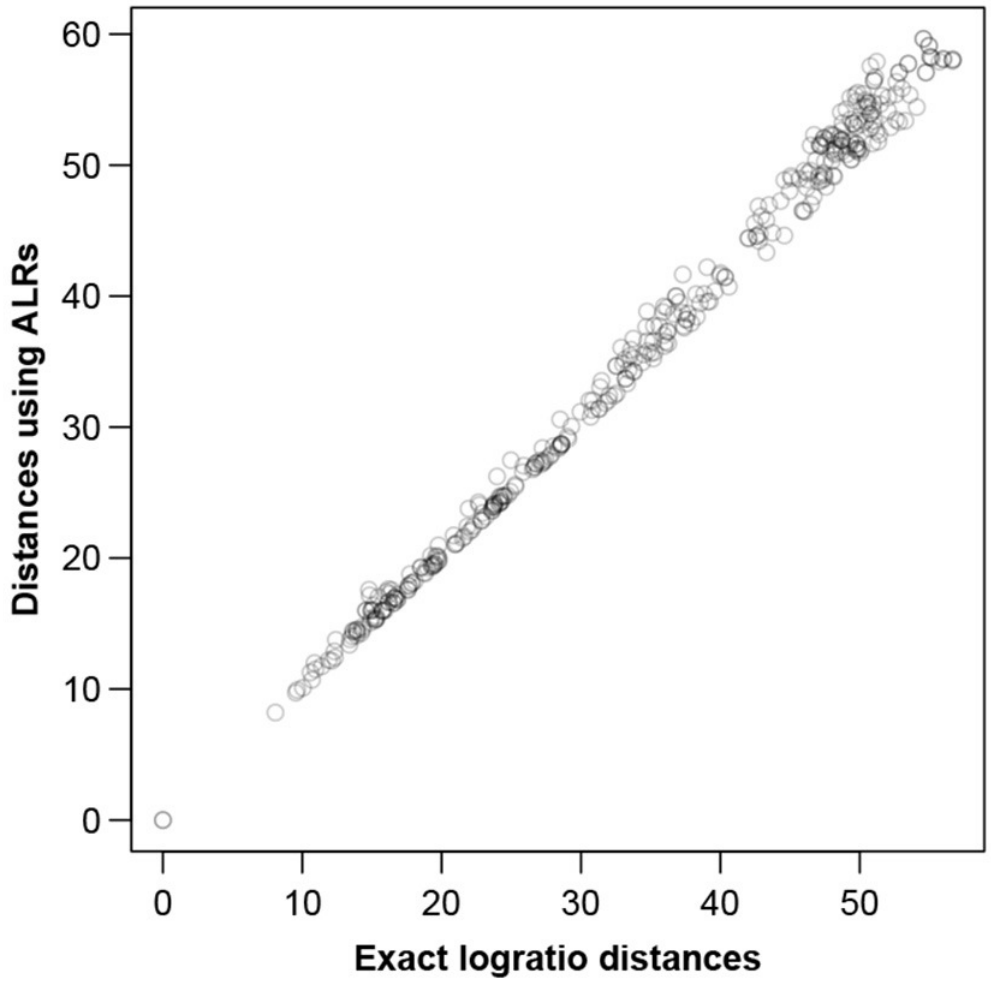
图二显示了在ALR上计算的所有样本间距离,基于所有成对对数的对数距离或同等情况下的所有CLR绘制相应的精确对数距离。
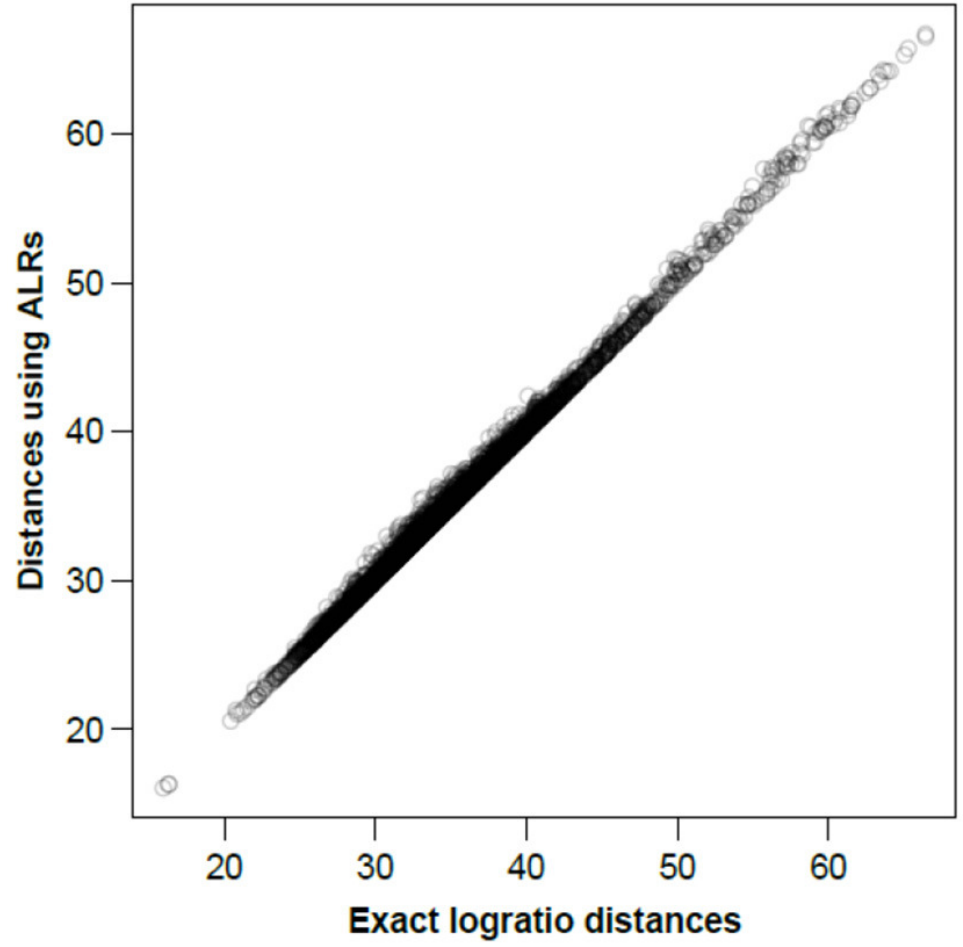
图二
图三为对于数据集的89个样本,参考基因编号856的计数与计数总和之间成正比。
图三
下图四展示了整个数据集的LRA(是所有成对对数的主成分分析(PCA),相当于所有CLR的主成分分析,以加权或非加权的形式)。
而图五中展示了具有参考基因856的ALR的对应PCA。主成分分析与参考成分微生物基因编号为856时,其几何形状实际上与确切的直线几何形状相同(Procrstes相关=0.9991)。字母S和F代表进行测序的两个实验室,显示出明显的分离。
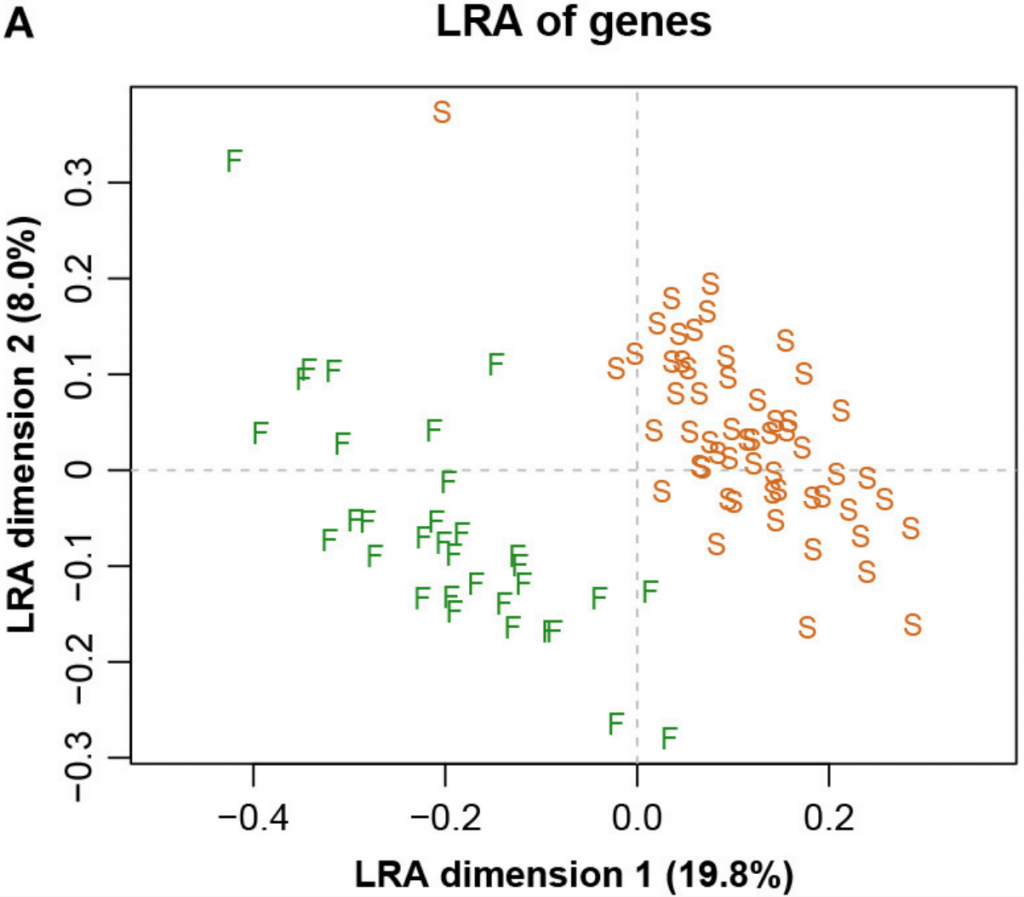

图四
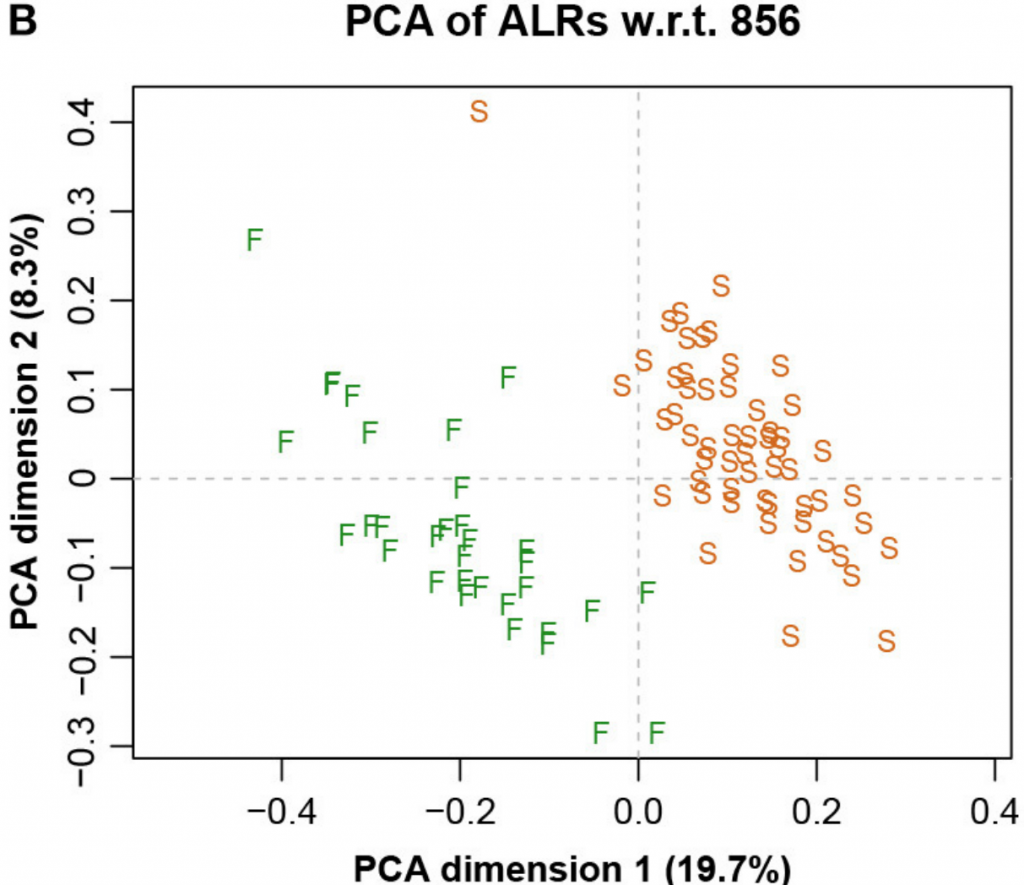

图五
2. 小鼠数据集
数据集大小,28个样本,3147个特征。此数据集中有34个零,使用R包zComposition中的函数cmultReplin替换。
总对数方差 0.2099,Procrustes相关系数最高为0.9977,对应转录本编号1318,其中转录本编号1179的Procrustes相关系数也与其相似。
图六
图七
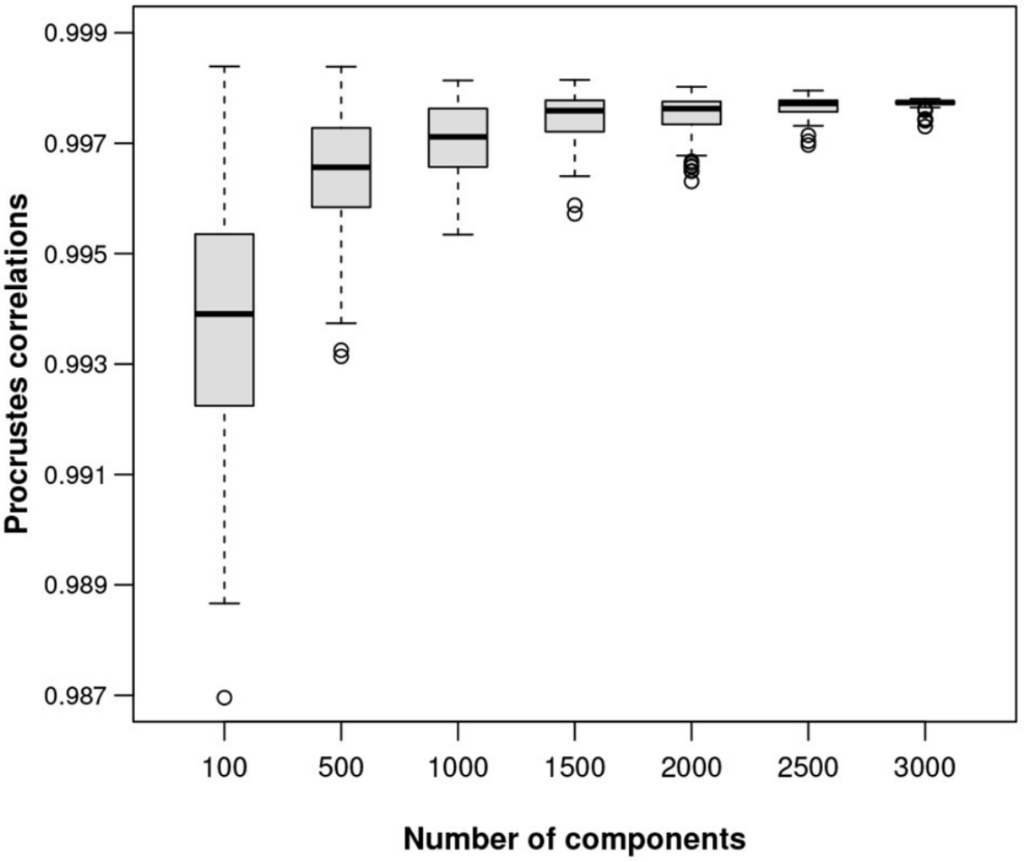
图六显示了在ALR上计算的样本间距离。为了显示任意大小数据集的ALR变换的质量,对MICE数据进行了模拟研究,从数据中随机抽取不同大小的样本,将每个样本作为独好的立的样本,并为该特定数据集的ALR变换找到最佳参考。
对于100、500、1,000、1,500、2,000、2,500、3,000和3,500个转录本的子集,以及每个子集的100个随机样本,绘制最佳的Procrstes相关性,如图七展示。ALR变换的等距质量随着可能的参考成分特征数量的增加而提高。
图八展示完整数据集的LRA,图九展示了参考转录本编号1179的ALR的PCA。它们实际上是相同的,只是有很小的差异,而在这之前的Procrstes相关系数结果就已经指示出了。标签代表两种不同的处理(L和M)和7种不同的时间(0、1、2、4、6、9和12h)。
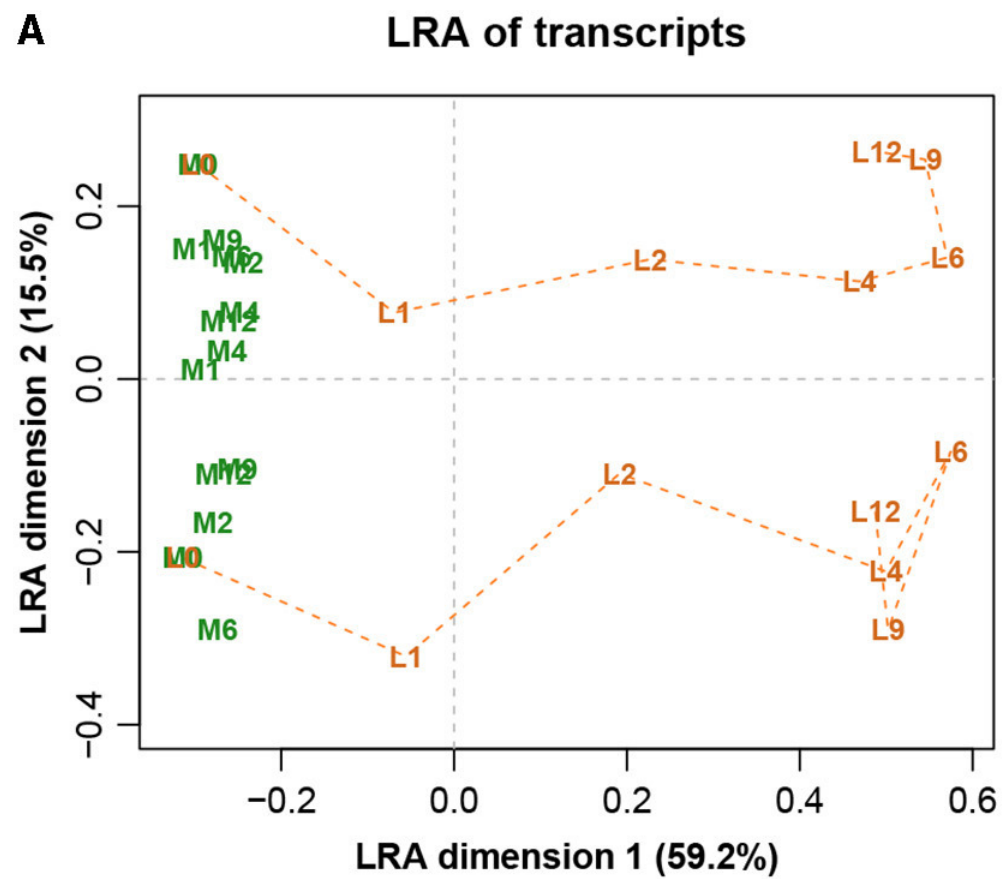
图八
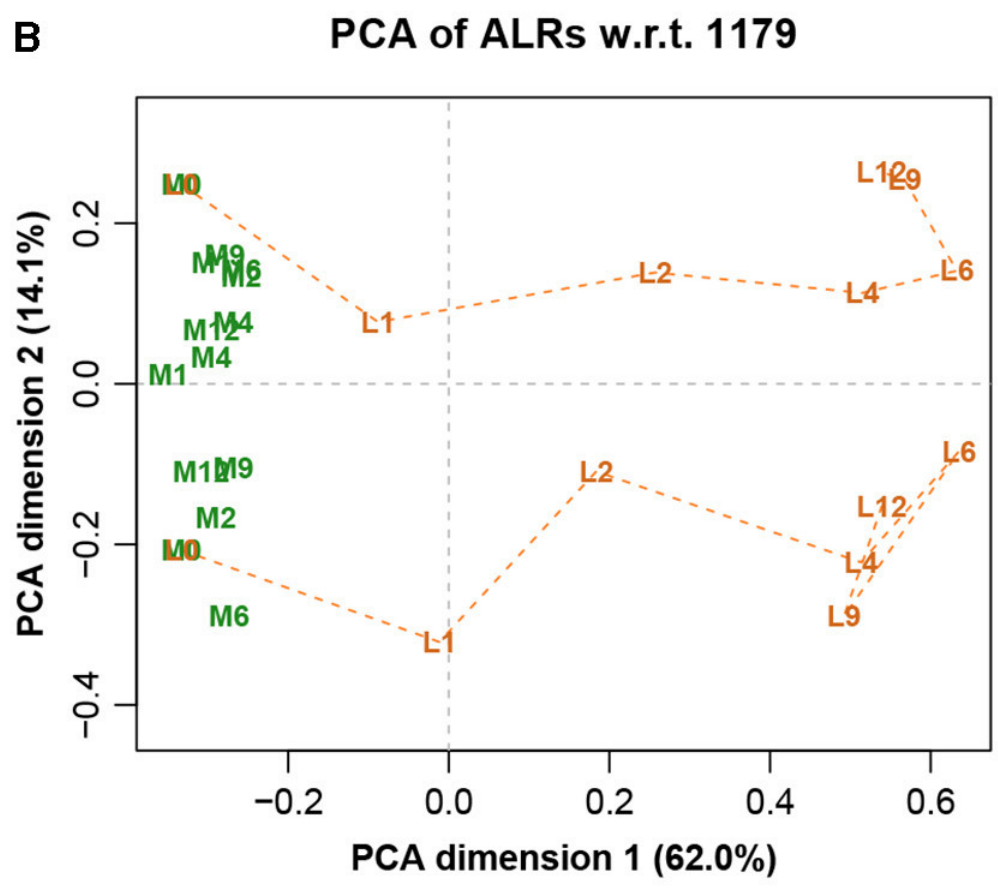
图九
3. 奶牛数据集
这是一个大小为211个样,127个特征的核磁共振强度数据集。样本被分成三个饮食组:精料组、混合组和饲草组,还测量了甲烷产量。

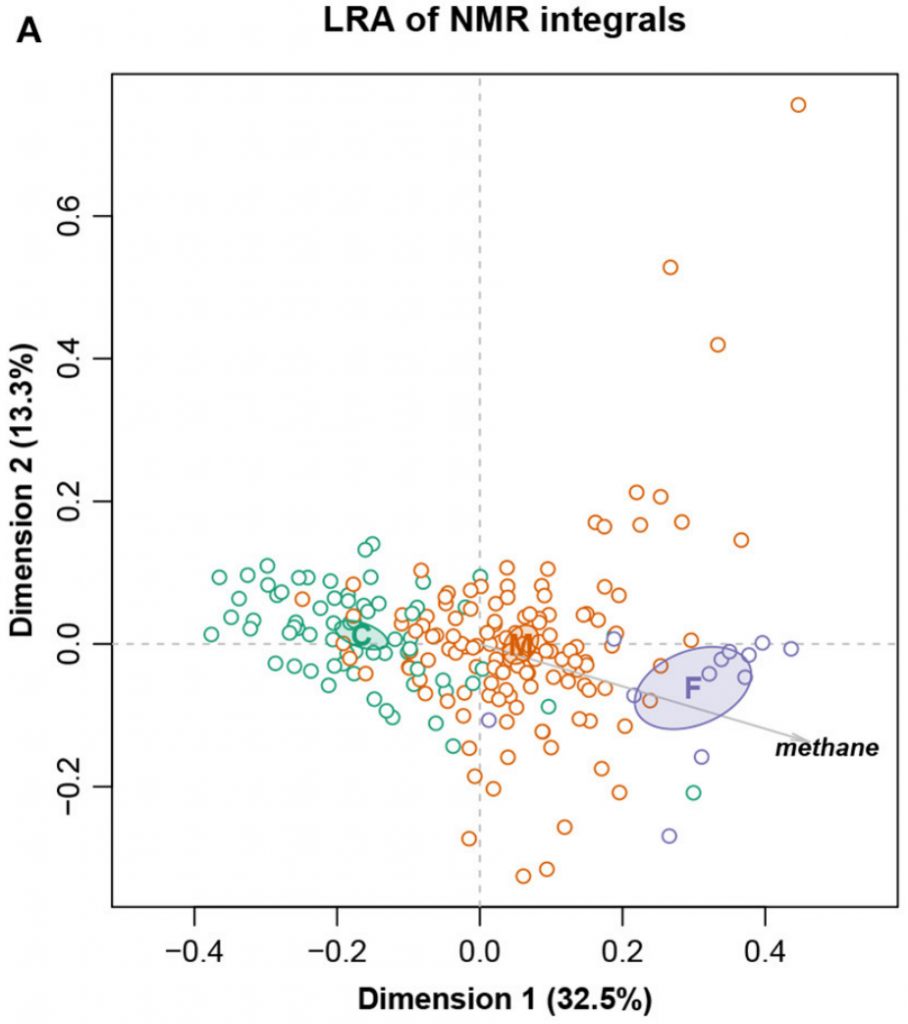
图十

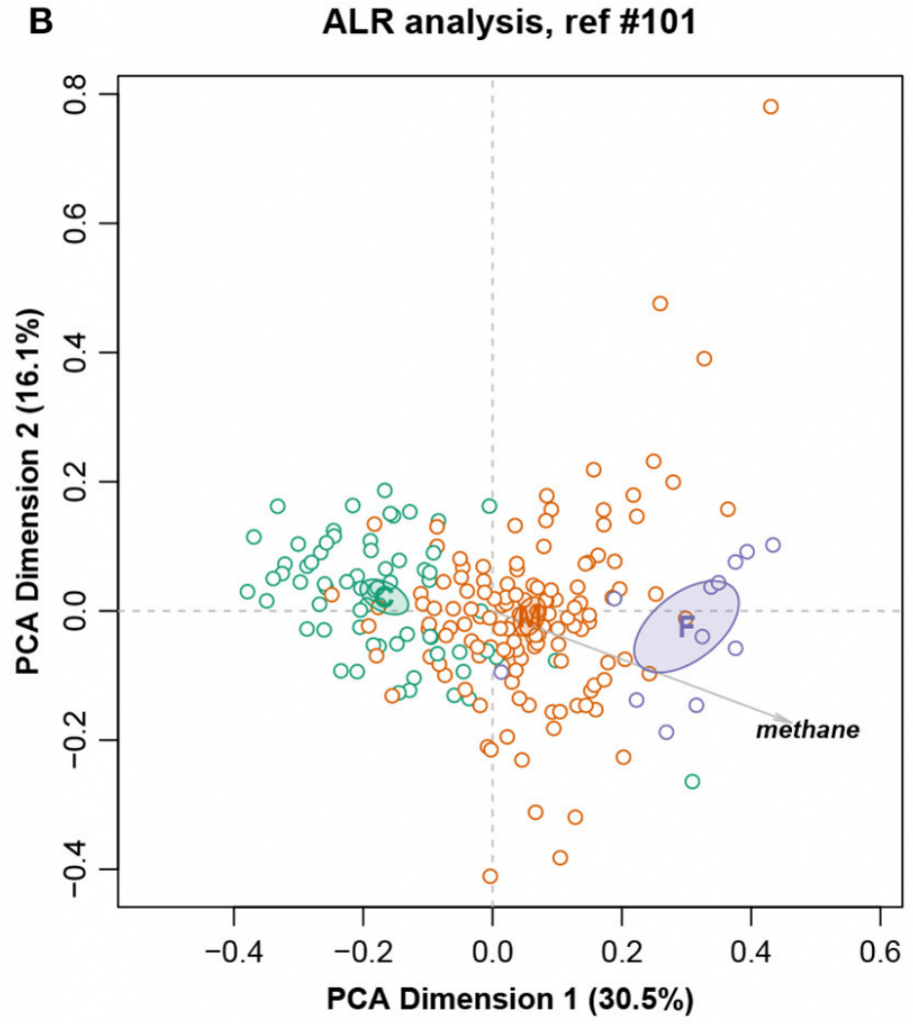
图十一
总对数方差0.09128,Procrustes相关系数最高为0.9902,对应于编号101。图十展示完整数据集的LRA,图十一展示了编号101的ALR的PCA。标签C(精料)、M(混合)和F(饲料)。
从以上三个数据集的验证分析不难看出,对于高维数据,使用ALR对数转换也能得到对全部特征使用CLR对数转换方法的结果,关键在于找到有效的参考特征(成分)。
文章中作者建议将其作为此类高维数据成分数据分析的第一步。作者公开了部分数据集的存放地址,以及用于数据处理的部分代码。可以自己尝试看看是否适用。
扩展:数据集位置及实用脚本
兔子数据集: https://www.ebi.ac.uk/ena/browser/view/PRJEB46755
小鼠数据集:http://doi.org/10.5281/zenodo.3270954
其它数据集及脚本:https://github.com/michaelgreenacre/CODAinPractice
在这个github中有详细列出文中所使用的用于数据处理的各个R源码,以及目前这些数据处理的相关函数。
而这些脚本现已被整合为R包,easyCODA,可以从CRAN中直接下载。在Rstudio中调用“install.packages(“easyCODA“)”。
Tips
在对成分数据(composition data)进行分析时,通常会对原始数据进行矫正,也可以理解为一种标准化方法。比较常用的对数转换方法是CLR(Centered Log-Ratio),其次是ALR(Additive Log-Ratio,也就是文章主要推荐的方法)和ILR(Isometric Log-Ratio)。
每种方法都有优缺点,对于后续统计分析的适用程度,CLR>ALR>ILR。个人建议先使用CLR和ALR对数据进行转换,然后使用PCA或其他降维分析方法查看其类群分布,搭配adonis查看其统计显著性水平。只要能达到预期结果就都能使用。如果CLR和ALR数据转换后结果差异不大,那推荐使用CLR。
参考文献:
Greenacre M, Martínez-Álvaro M, Blasco A. Compositional Data Analysis of Microbiome and Any-Omics Datasets: A Validation of the Additive Logratio Transformation. Front Microbiol. 2021 Oct 11;12:727398. doi: 10.3389/fmicb.2021.727398.

谷禾健康
ResistoXplorer基于Web的耐药基因组数据可视化,统计和探索性新分析工具。
对宏基因组测序后的数据进行抗生素耐药性基因组的注释与分析,逐渐成为一条必经之路。过去,人们需要自己下载相关数据库再用比对工具进行比对,然后去冗余,再进行下游分析。这通常需要学习编程并熟练应用,对于一些临床医生或科研人员是一个很大的挑战。
最近有一款新的工具,用于对耐药基因组数据的成分分析,功能分析和比较分析。
ResistoXplorer,一款Web程序,地址:http://www.resistoxplorer.no
ResistoXplorer的主要功能包括:
1.支持多种常用和先进的方法,用于成分分析、可视化和探索性数据分析
2.全面支持各种数据归一化方法,包括标准的和最新的统计和机器学习算法
3.支持对配对数据集进行垂直数据综合分析的多种方法
4. ARG功能注释及其微生物和表型关联,基于10多个参考数据库的对比结果
5.功能强大且齐全的网络可视化,直观展现ARG于微生物的关联
打开网址后的界面:

由三个主要分析模块组成(上图红色箭头所指框内):
“ARG List”:探索给定的ARG信息的功能和微生物宿主的关联,可视化网络。
“ARG Table”:对从宏基因组组学研究中获得的耐药基因组丰度文件进行功能分析,α多样性分析,排序分析,差异丰度分析等。
“Intergration”:综合分析,进一步探索潜在的联系,并结合新的生物学见解和假说,相似性分析,成对微生物-ARG相关分析等
上图绿色箭头所指框内:
“DataFormat”和“About”: 提供了关于注释表的格式、结构和数据库统计信息的详细描述
“FAQs”:提供了一些问题的答疑
“Resources”:分为“Manuals”和“Downloads”两个模块
Manuals是使用手册,对用户进行操作指导,建议仔细阅读。

Downloads,提供了示例上传文件和单个数据库的下载
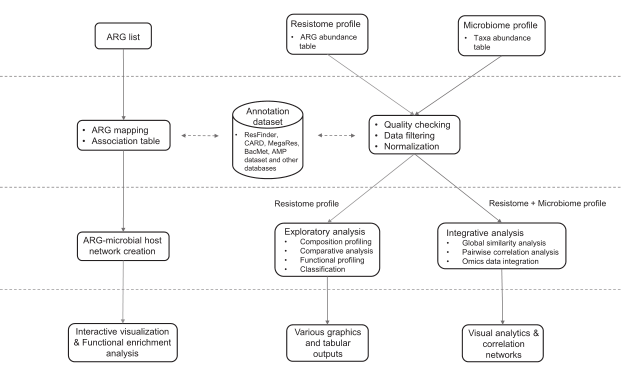
分析流程
ResistoXplorer接受抗性基因列表和ARG/taxa丰度表作为输入数据。然后是数据处理、数据分析和结果输出三个步骤。数据处理包括数据过滤和标准化,数据分析包括成分分析,比较分析和综合分析。结果输出以可视化图形,表格或html格式输出。
ResistoXplorer的功能注释使用的参考数据库来自9个通用的AMR数据库,CARD、ResFinder、MEGARes、AMRFinder、SARG、DeepARG-DB、ARGminer、ARDB和ARG-ANNOT。
此外,研究人员还从BacMet数据库和抗菌肽(AMP)耐药基因数据集中手动构建了功能注释信息,使用户能够对抗菌药物/金属和AMP抗性基因进行功能分析和下游分析。
数据处理、分析及结果
数据过滤和标准化
默认情况下,低质量的特征会根据样本流行度及其丰度水平进行过滤。默认值是其他工具所使用的值,大多数在文献中可以找到。用户可以根据分位数间范围、标准差或变异系数排除这些低变异特征。
除alpha多样性和稀疏性分析外,过滤后的数据大多数用于下游分析。在综合分析的情况下,用户还可以对分类注释和耐药基因组丰度数据选择不同的数据筛选标准。
过滤后的数据还需要normalization(归一化)。ResistoXplorer提供了三种数据归一化方法,rarefying, scaling和transformation(稀疏、缩放和转换)。此外还支持其他归一化方法,如中心对数(CLR)和加性对数比(ALR)变换,以便于成分数据分析。方法的选择取决于要执行的分析类型。归一化后的数据用于探索性数据分析,包括排序、聚类和综合分析。用户可以自行探索适合的参数。
成分分析
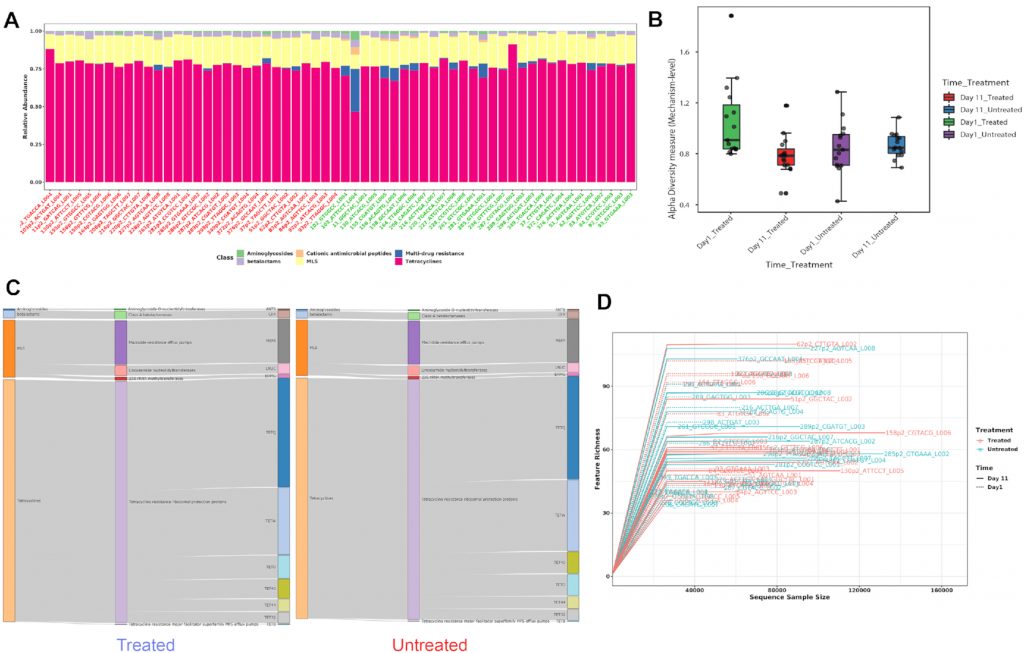
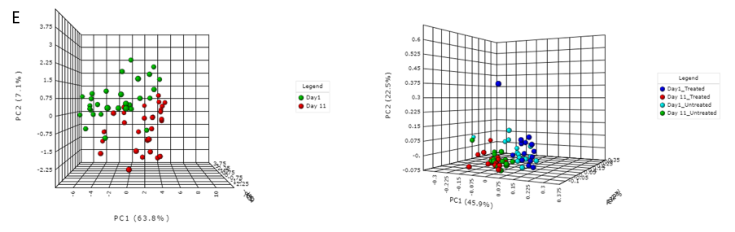


A) 显示各样本在不同分类水平下的ARG丰度。
B) Shannon多样性指数
C) 桑基图。显示了各组内的包括类别,机制和分组的ARG丰度分布。
D) 稀疏曲线。评估样本中估计的多样性的可靠性,在稀疏曲线中,识别的唯一特征(ARG)的数量与序列样本大小相对应。
E) 排序分析。左边是基于时间点的带有样本颜色的3D PCA图。右边是根据不同的治疗组和时间点绘制3D PCoA图。目前,支持三种通用的排序方法, PCoA、NMDS和 PCA。结果表示为2D和3D样本图。
比较分析
差异丰度分析
使用DESeq2、Edger、metagenomeSeq、Lefse,以及单变量分析方法,比如ALDEx2和ANCOM。DESeq2和Edger说明计数数据的特征,相比之下metagenomeSeq使用推荐的CSS规范化,在更大的分组规模下具有更高的性能。
Lefse使用标准的非参数检验统计显著性,结合线性判别分析来评估差异丰富特征的效应大小。
ALDEx2对来自数据的模型化概率分布的对数比值执行参数或非参数统计测试,并返回统计测试的期望值以及效应大小估计。
ANCOM使用非参数统计检验来检验所有特征对的对数比丰度,以找出均值差异。结果以表格样式展现。
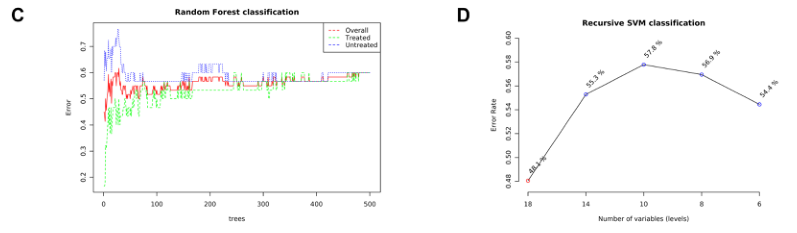
基于机器学习的分类
提供了两种功能强大的监督分类方法–随机森林和支持向量机(SVM),以识别潜在的生物标志物。
C)随机森林
D)展示了SVM在特征(变量)数量减少的情况下的分类性能
其他的一些可视化分析

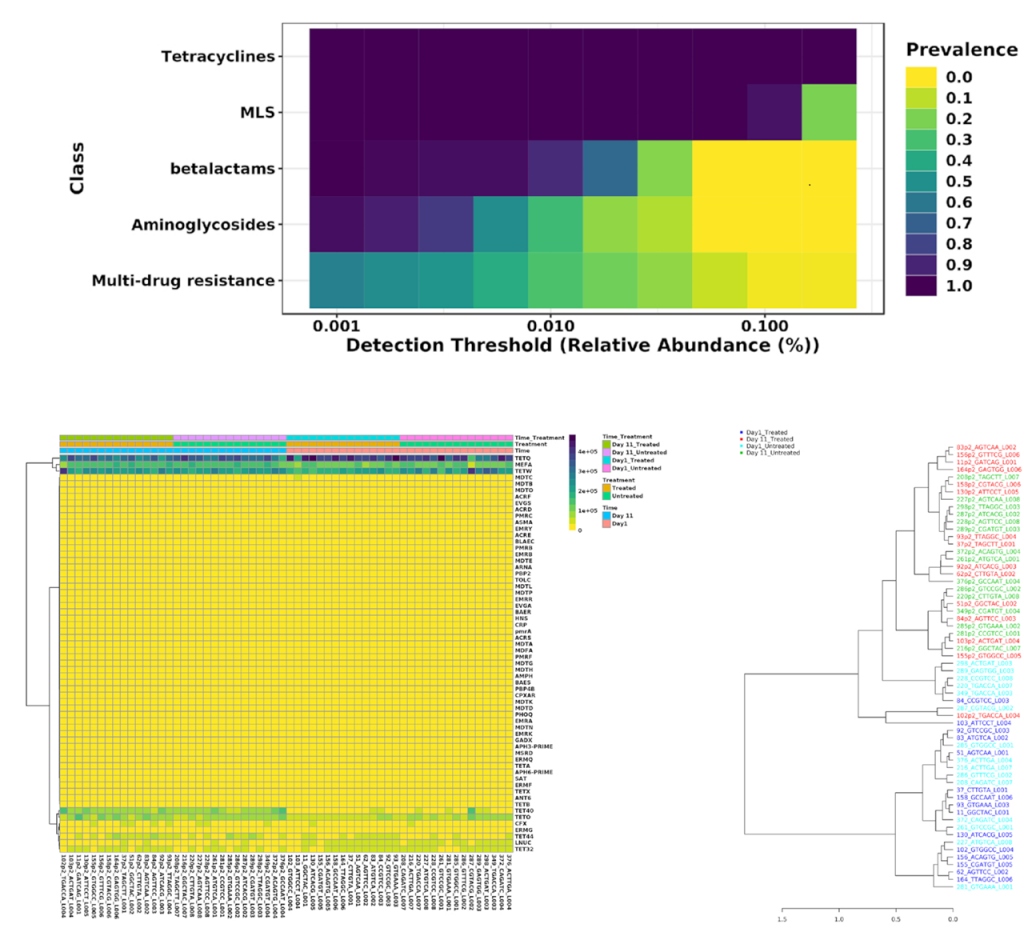
用户可以根据样本的丰度和流行程度,执行核心抗性分析来检测样本或样本组中存在的核心特征集,以热图的形式展现;以及关联分析和层次聚类,使用热图或者树状图可视化。
综合分析
使用各种综合数据分析方法来探索和揭示微生物群和抗性群之间潜在的潜在关联,这种分析大多用于探索不同环境中细菌和ARGs之间的联系。目前,为数据集成和相关分析提供了几种领先的、常用的单变量和多变量统计方法。所有这些分析都是在过滤和归一化数据集上执行的。
全局相似性分析
用两种基于多变量相关性的方法来确定微生物组和AMR数据集之间的总体相似性,分别为普鲁克分析(PA)和协惯量分析(CIA),在各种功能和分类级别上执行分析。相似系数和P值用于评估两个数据集之间的关联的强度和显著性,相似性系数在0到1之间,0表示两个数据集之间的完全相似,而1表示两个数据集之间的完全不相似。可视化结果用2D和3D排序图表示,如下图

A) 来自普鲁克分析的3D NMDS图,包含与数据集相关的样本、形状和颜色。
B) 来自协惯量分析的3D PCoA图,其中连接两点的线的长度表示两个数据集之间的样本的相似性。
组学数据集成方法
基于多变量投影的探索性方法,如正则化典型相关分析(RCCA)和稀疏偏最小二乘法(SPLS),用于微生物组和AMR数据的集成。这些方法旨在突出高维“组学”数据集之间的相关性。
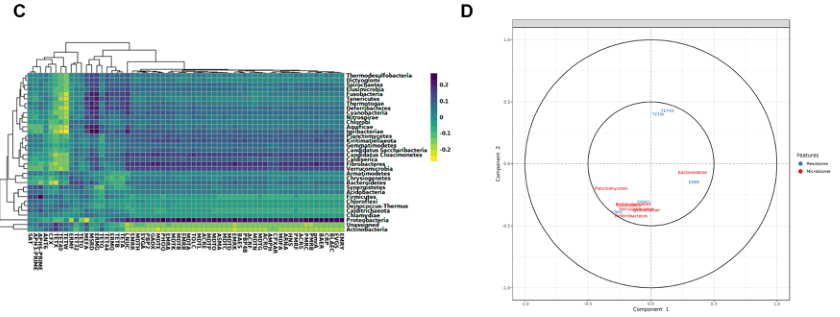
A 门水平微生物群落与ARGs(组水平)之间的聚类图像热图
B 显示存在于两个数据集中的特征(分类群/参数)的相关结构的相关圆图
成对微生物-ARG相关分析
使用单变量相关分析来确定单个菌群和ARGs(耐药基因组)之间是否存在强相关。使用Spearman、Pearson、CCLasso和最大信息系数(Maximal Information Coefficient)四种方法。用户可以使用绝对相关系数和调整p值的组合来选择强且显著的成对相关性。结果如下图,每个节点表示一个菌或ARG。用户可以双击一个节点,以突出显示网络中相应的相关节点。边缘的宽度和颜色表示两个节点之间相关性的强度和方向。
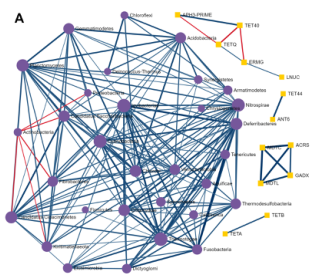
探索ARGs-微生物宿主网络
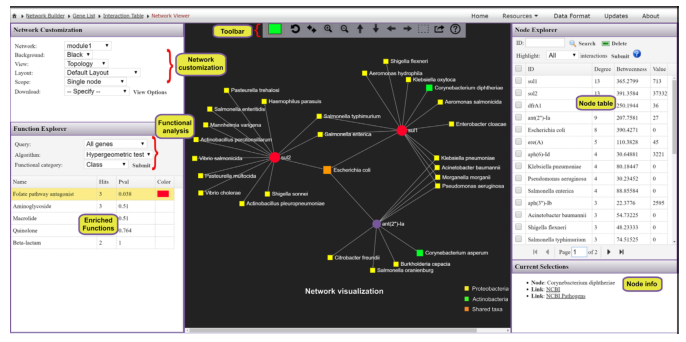
基于网络的可视化分析系统,提供了解ARGs和微生物宿主之间复杂的“多对多”关系的可能性。例如,通过查找在多个微生物中发现的ARGs或通过识别同时包含多个感兴趣的ARGs的微生物,可以直接从网络的角度找到承上启下的关键点。
从ResistoXplorer程序中涵盖的数据库中搜集ARGs-微生物宿主信息,构建的关联表用于网络可视化和功能分析。如下图,它由三个主要组件组成:中央网络可视化区、左侧的网络定制和功能分析面板,包含节点表的右侧面板。
用户可以使用带滚轮的鼠标直观地查看和操作中心区域的网络。例如,可以滚动滚轮来放大和缩小网络,将鼠标悬停在任何节点上以查看其名称,单击节点以在右下角显示其详细信息,或双击节点以将其选中。
顶部的水平工具栏显示了操纵网络的基本功能。第一个是颜色选择器,能够为下一次选择选择高亮颜色。还可以使用工具栏中的虚线方形图标选择并拖动多个节点。对当前网络中存在的ARGs进行功能富集分析,使用超几何测试方法,这种方法与网络可视化系统相结合,在解释AMR耐药机制和提供ARGs的可能传播路径信息可能会有更好的效果。
文章中为了展示该工具的可用性,在已发表的一些研究中,选择了1个研究进行抗性分析,“利用商业饲养牛检验图拉霉素(抗菌药物)对肠道微生物组和耐药性的影响”,分析的内容就如同上面展示的那样,这里就不多加赘述。
与其他工具的比较,文章中也列举了一个表格,分别与AMR++Shiny、resistomeAnalusis、WHAM!在分析模块上进行了比较。实际上大同小异,主要的分析模块以及使用的数据库都是相似的,只是谁的数据库更强大,搭载的分析模块更多的区别。
哪款软件的算法和统计分析匹配你的实验数据,或者它能为你提供更多的数据信息,就是适合你的。
这款在线分析抗生素耐药性基因组的程序值得探索一下,统计分析方法和数据库内容都挺强大的,交互式的使用也免去了对编程语言的探索,并且开发人员也表示会持续更新和精选数据库以达到更准确的下游分析。
参考文献
Dhariwal A, Junges R, Chen T, Petersen FC. ResistoXplorer: a web-based tool for visual, statistical and exploratory data analysis of resistome data. NAR Genom Bioinform. 2021 Mar 24;3(1): lqab018.
Interagency Coordination Group on Antimicrobial Resistance No time to wait–securing the future from drug-resistant infections. Rep. Secret. Gen. Nations. 2019.
Simonsen G.S., Tapsall J.W., Allegranzi B., Talbot E.A., Lazzari S. The antimicrobial resistance containment and surveillance approach-a public health tool. Bull. World Health Organ. 2004; 82:928–934.
Cecchini M., Langer J., Slawomirski L. Antimicrobial Resistance in G7 Countries and Beyond: Economic Issues, Policies and Options for Action. Paris: Organization for Economic Co-operation and Development. 2015; 1–75.
Xia Y., Zhu Y., Li Q., Lu J. Human gut resistome can be country-specific. PeerJ. 2019; 7:e6389.
Forslund K., Sunagawa S., Kultima J.R., Mende D.R., Arumugam M., Typas A., Bork P. Country-specific antibiotic use practices impact the human gut resistome. Genome Res. 2013; 23:1163–1169.