 国家高新企业 | ISO9001认证
国家高新企业 | ISO9001认证 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康

尽管地球上微生物类群的繁多,但只有一小部分得到了培养和有效命名。因为大多数菌无法在非常特定的条件下培养分离鉴定。
在过去十年中,宏基因组研究的重要性已经凸显,因为它能够评估细菌基因库并发现当前实验室培养技术无法掌握的新细菌基因组。这些数据对于扩大我们对地球上微生物多样性的理解至关重要。
由于宏基因组测序数据由来自多个物种和菌株的 DNA 序列片段组成,通常有数千个来自不同生命领域,因此此类分析的主要挑战是正确确定每个 DNA 序列片段的真实来源。不幸的是,这些步骤容易出错,因此必须对结果进行严格审查,以避免发布不完整和低质量的基因组。
最近,比利时研究人员新开发MAGISTA,这是一种评估宏基因组基因组组装质量的新方法,基于随机森林的方法估计MAGs的完整性和污染度,解决了当前基于参考基因的方法经常被忽视的一些缺陷。
MAGISTA是基于宏基因组bins内的contig片段之间的无对齐距离分布,而不是一组参考基因。该方法利用了来自整个 bin 的信息。为了正确评估此方法,并说明基于参考的工具的缺点,最近,比利时研究人员构建了一个高度复杂的 DNA 模拟群落,由 227 个细菌菌株组成,并且具有不同程度的相似性。

方 法
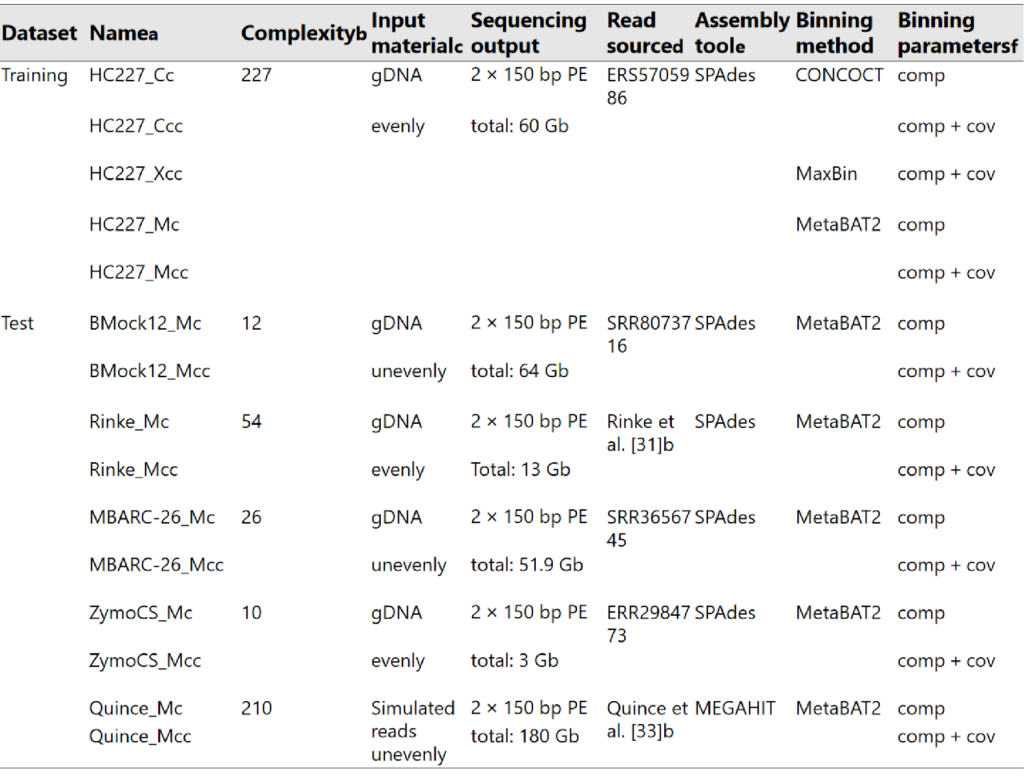
训练集来(HC227)自 227 个细菌菌株,测试数据集由五个公开可用的短读(short reads)子集构成,其中四个含有来自复杂度相对较低的基因组 DNA 模拟群落的reads。具体情况如下图所示。
Complexity列指示菌株数;Assembly tool列表示所使用的用于组装的软件;Binning method列表示所使用的用于分箱的工具;Binning parameters列表示所使用的用于评估分箱质量的指标,comp为完整度,cov为覆盖率。
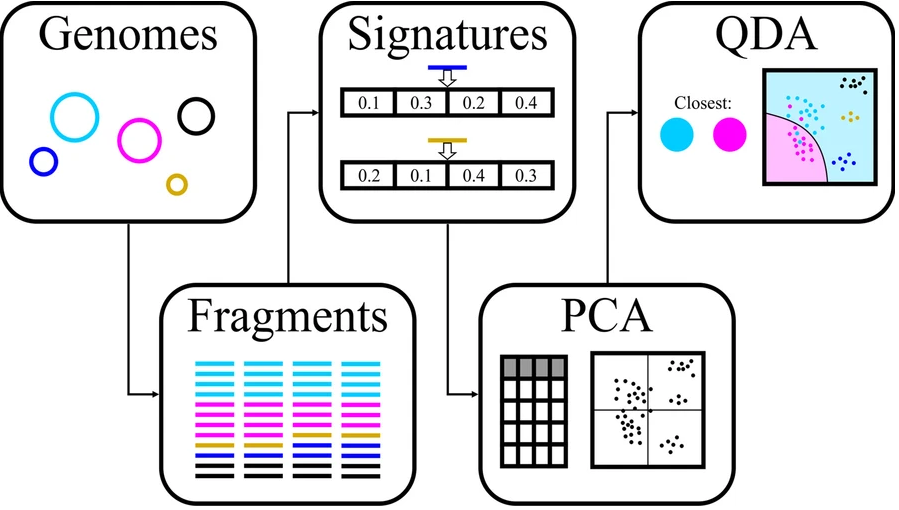
MAGISTA计算步骤:
输入binning后的每个bins
-●-
第 1 步:选择适合的片段大小与距离计算方法
-●-
首先将每个 bin 中的每个 contig 拆分为固定长度的片段,然后使用四种不同的方法(即 PaSiT4、MMZ3、MMZ4 和 Freq4)计算一个 bin 中的片段之间的所有距离。对于每种方法,都选择了特定的片段长度,以便为不同的生物产生不同的特征分布。
每种方法的最终片段长度的选择是通过不同方法分析整合决定的,方法如下图所示。每组的设计中至少两个基因组来自同一个家族,两个基因组来自相同的顺序但来自不同的家族。这些基因组被人为地分成所需长度的片段,并为每个片段计算目标特征。
对于每组五个基因组,混合所有片段并根据它们的特征进行主成分分析(PCA),然后进行二次判别分析,用于生成分类器,旨在区分每组中重叠最多的两个基因组。对该分类器的准确度取平均值,结果用于选择方法和片段长度的最终组合。
-●-
第 2 步:模型中特征变量的选择
-●-
为每种方法选择片段长度后,使用平均值、标准差、偏度、峰度和中位数以及 2.5%、5%、10%、90%、95% 和 97.5% 百分位数计算距离分布。此外,还计算了 1 kb 片段的 GC含量分布。以及每个bin的大小,共计66个特征变量。
-●-
第3步:模型构建
-●-
使用 R (v 4.0.3) 包“RandomForest”中的“RandomForest”函数和默认参数训练随机森林模型。同时使用R包lm再建立一个线性模型执行线性回归,输入经对数转换后的特征变量值,用于交叉验证分析。
主 要 结 果
一个高度复杂的基因组DNA模拟群落
由来自 227 个细菌菌株的基因组 DNA 组成,这些菌株属于8 个门(Actinobacteria, Bacteroidetes,Deinococcus-Thermus, Firmicutes,Fusobacteria,Planctomycetes, Proteobacteria和Verrucomicrobia),18 类,47目,85科,175属,197种。
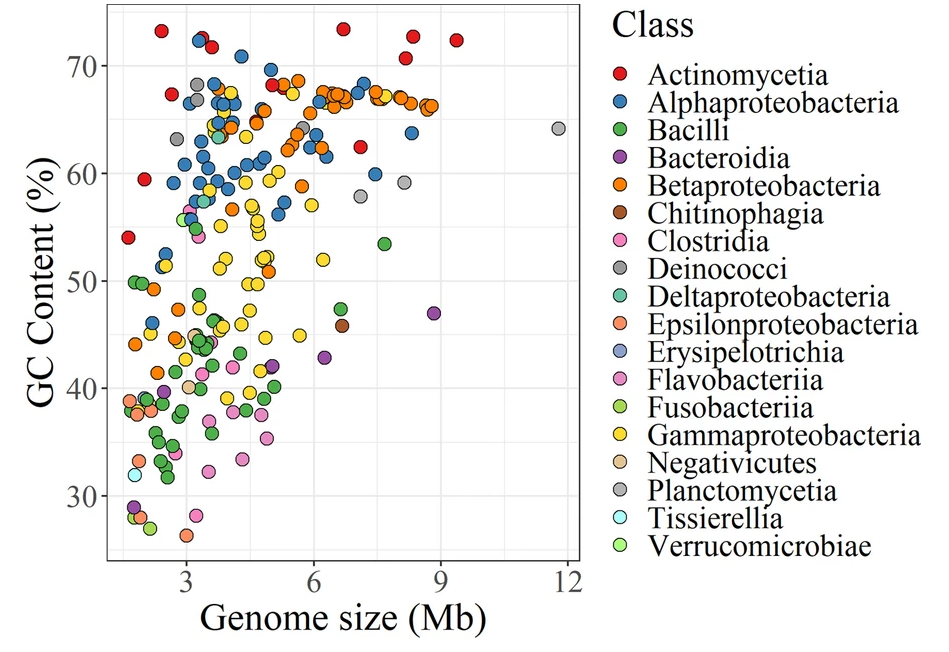
编辑
上图为模拟群落中的细菌菌株的基因组大小和GC含量(从26.3%到73.4%)散点图;
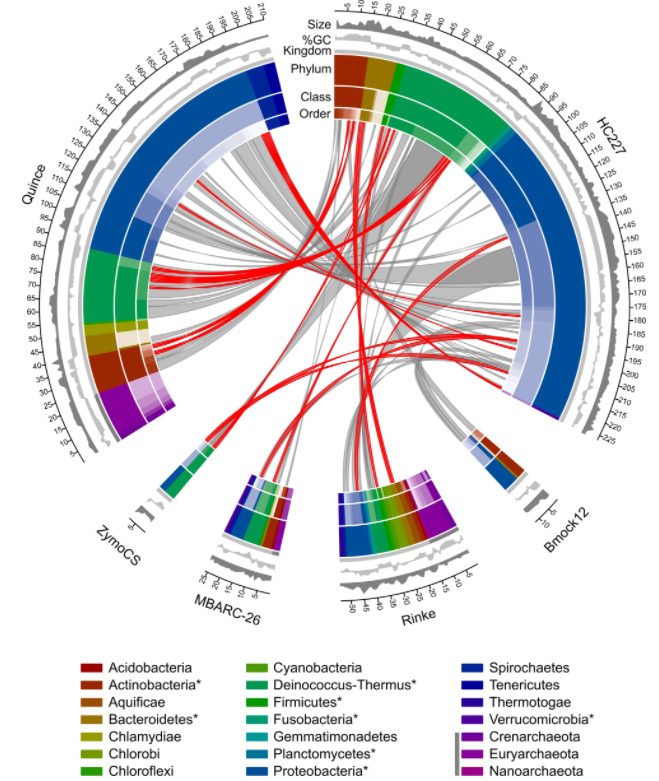
编辑
图为训练集与测试集中物种之间的关系图。红色线条表示在训练集中存在的菌种,灰色线条表示在训练集中存在的菌属。环状图中的不同颜色代表不同分类水平。图例中存在于训练集中的菌门用*标记,存在于古生菌的菌门用深灰色色带标记。
CheckM中基于单拷贝标记基因(SCMG)来评估 bin 质量的存在的缺陷
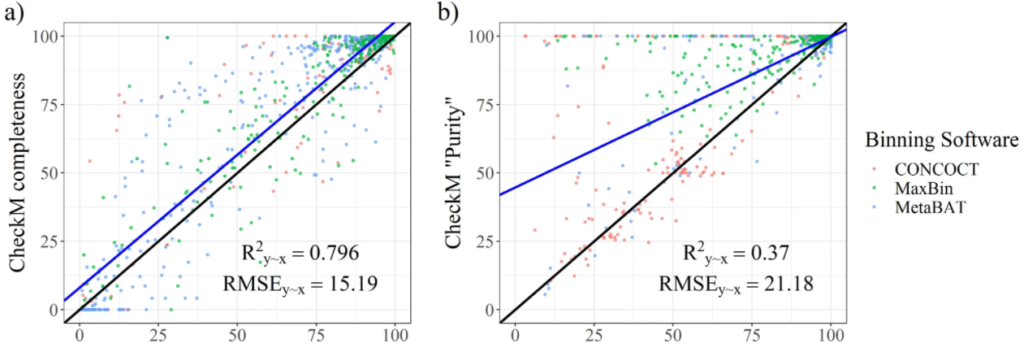
图a和b分别为从CheckM中输出的完整性指标和污染度。使用R^2y∼x(解释方差的百分比),RMSE(相对于实际值的均方根误差)两个参数评估结果。结果表示CheckM高估了bin的质量。许多受污染的bins被预测为接近未受污染。
使用MAGISTA分析模拟群落中的bins
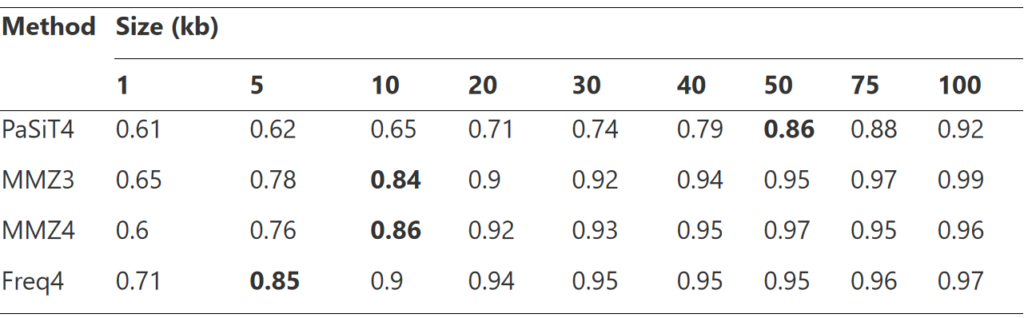
首先选择最佳片段大小用于计算距离分布,如上图所示,考虑了 1、5、10、20、30、40、50、75 和 100 kb 的片段,最终选择了粗体所示的片段大小。
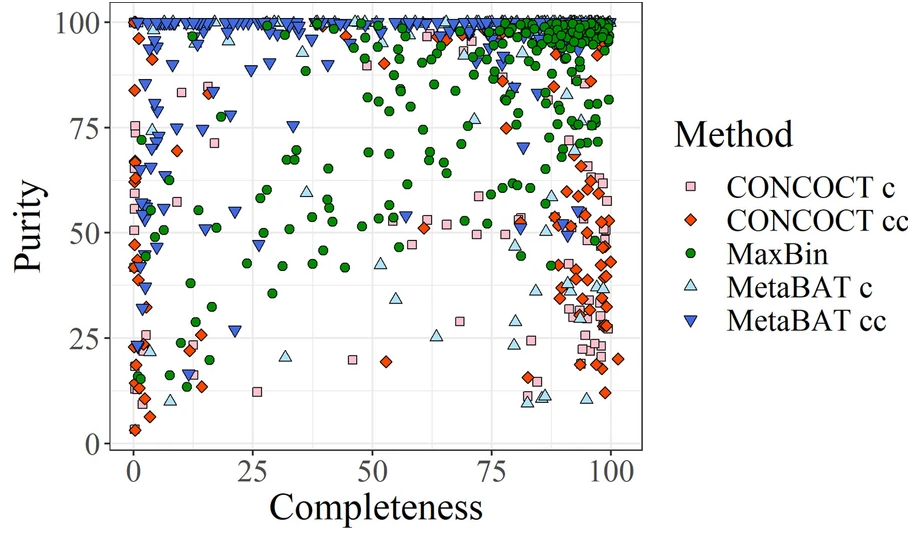
图为concont、MetaBAT和MaxBin产生的bins的完整性和污染度信息。
由于通过模拟生成这样的数据集并不能准确地表示真实的结果,所以使用了binning软件的结果,提供了一组不同质量的真实的bins。训练数据集的完整性和未污染度均在90%以上。
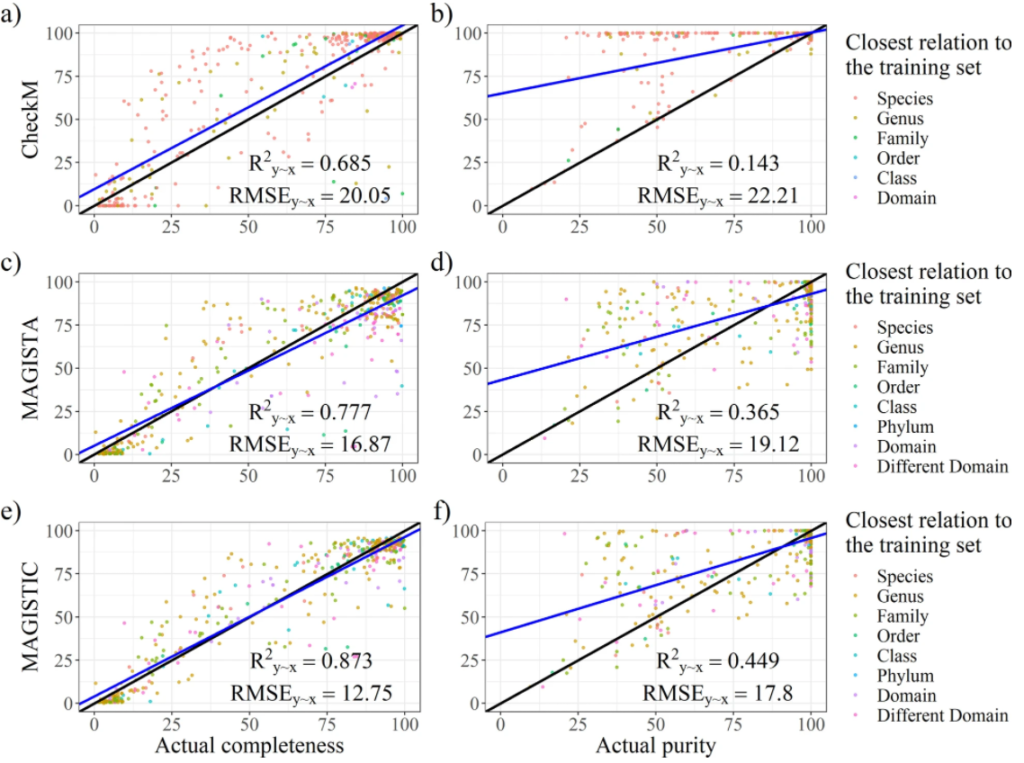
最后是模型构建,建立完整性和污染度的预测模型。并进行了模型评估,如图所示。分别对CheckM、MAGISTA 和 MAGISTIC测试了其性能。CheckM是现在主流的一款评估bin质量的工具。MAGISTIC是一款结合了CheckM和MAGISTA 的工具。使用解释方差的分数(R2y∼x)和均方根误差(RMSE)作为评估性能的指标。对于完整性的预测,MAGISTA 优于 CheckM。对于污染度的预测,MAGISTA 的表现优于 CheckM。
结 论
研究人员开发了一种新的用于预测高度复杂的宏基因组组装基因组bin的质量的方法,MAGISTA。是基于 SCMG 的低复杂性宏基因组方法的一个同样好的替代方法。除了MAGISTA之外,还通过结合CheckM的结果,使用MAGISTIC生成了一个更准确的预测。
研究人员在文章中指出MAGISTA 和 CheckM 都没有达到足够的准确度来被认为是可靠的。MAGISTIC 产生了比 MAGISTA 更好的结果。
在附加分析中,将测试集分为了两个子集,从真实和模拟reads中获得的bins,对此再进行分析,结果表示,CheckM 对于“真实”子集表现良好(但相比MAGISTA 和 MAGISTIC还是较差),对于“模拟”子集部分表现较差。而MAGISTIC相比MAGISTA会更准确些。但是文章中并没有详细说明MAGISTIC的工作流程。
查看作者在github上公开的软件说明,地址如下。但是没有说明和给出输出文件的内容。个人认为还不太成熟。
https://github.com/LM-UGent/MAGISTA
参考文献:
Goussarov G, Claesen J, Mysara M, Cleenwerck I, Leys N, Vandamme P, Van Houdt R. Accurate prediction of metagenome-assembled genome completeness by MAGISTA, a random forest model built on alignment-free intra-bin statistics. Environ Microbiome. 2022 Mar 5;17(1):9. doi: 10.1186/s40793-022-00403-7. PMID: 35248155; PMCID: PMC8898458.