-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康

为什么一种药物对一位患者反应良好,但对另一位患者反应不佳,甚至在某些情况下会导致严重的药物不良反应?
研究表明,一些药物的反应率在50%-75%,也就是说很多人服用药物后其实没有得到实际的改善,这并不代表该药物没用,可能只是对一些人效果不大,这是什么原因导致的呢?
这其实与药物反应个体差异(简称IVDR)相关。这种差异归因于多种因素,包括遗传、年龄、性别、生活方式、疾病状态、周围环境、肠道微生物群和药物相互作用等。
在精准医学正在加速来临的时代,根据个人的遗传、环境和生活方式因素制定量身定制的医疗干预措施成为可能,然而,药物反应的个体差异是实施精准医疗的主要障碍。因此,需要更好地了解药物反应的个体差异,以提高药物疗效或预防不良反应。
研究表明,在肠道中,药物和微生物群之间存在复杂的相互作用:
▸ 一方面,药物可以导致肠道微生物群的组成和功能的改变,导致微生物代谢和免疫反应的变化。
例如,二甲双胍可以减少肠道中的脆弱拟杆菌,降低该菌的胆盐水解酶活性,使GUDCA水平升高,以不依赖于肠道AMPK的方式,抑制肠道FXR信号,发挥改善代谢的作用。
▸ 另一方面,肠道微生物群可能改变药物,包括产生催化或降解药物分子的酶、改变药物在宿主体内的代谢水平以及与药物分子竞争代谢酶等方式,直接或间接地改变药物的吸收、分布、代谢和排泄,从而影响药物疗效。
例如,同样使用华法林的心血管疾病患者,有些人对该药物反应较低,这部分患者的肠道中拟杆菌、志贺氏菌、克雷伯氏菌较多,华法林是一种抗凝血药物,通过抑制维生素K的活性来减少血液凝块的形成。志贺氏菌有助于产生维生素K。因此,如果肠道中的志贺氏菌数量较多,它们可能会提供足够的维生素K,从而减少华法林的抗凝血效果。
本文我们来了解一下,常用药物和肠道微生物组之间复杂的双向相互作用,通过人体各个系统中的不同例子来阐述具体的机制。对这方面的逐步了解,有助于改善药物的效果,同时最大程度地减少不良反应,也有助于推动精准医疗的发展。
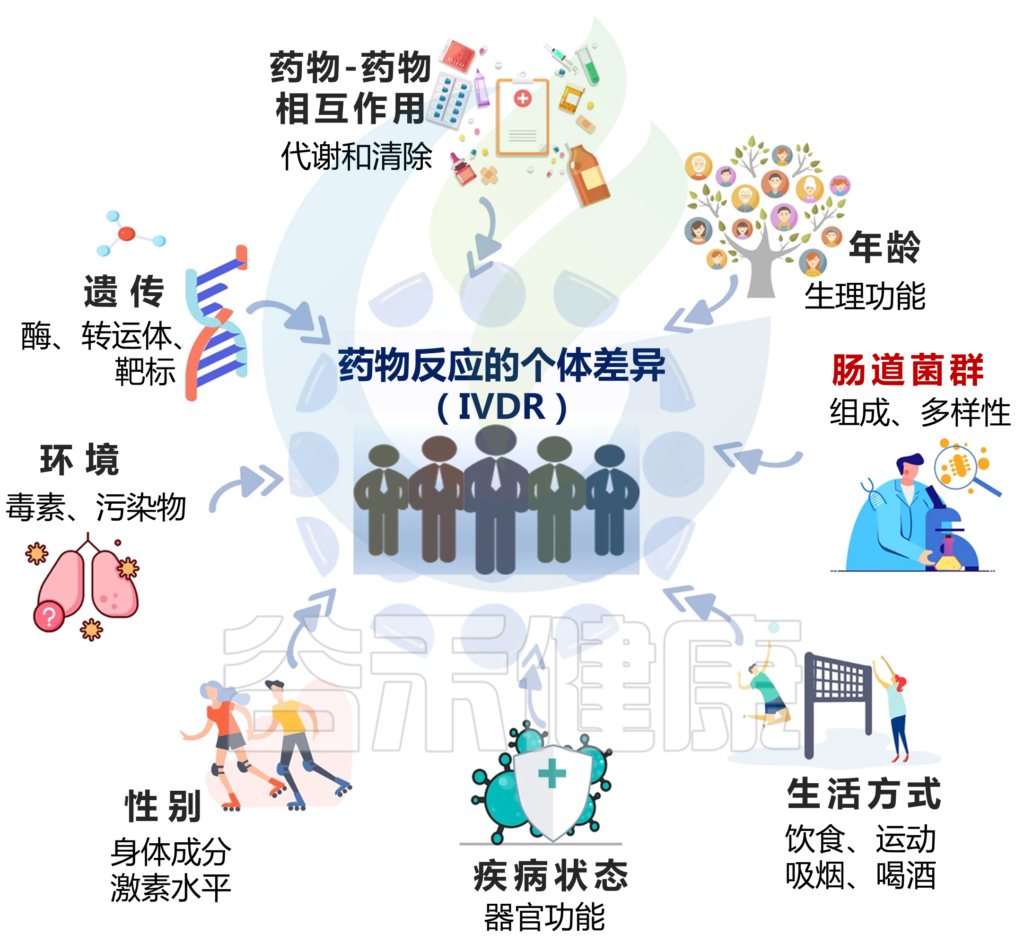
药物反应的个体差异(IVDR)是指不同个体在使用相同药物时表现出的不同反应情况。这种差异可以涉及药物的吸收、分布、代谢、排泄等方面。
了解个体差异可以帮助医生更好地调整药物剂量和选择合适的药物,以最大程度地提高治疗效果并减少不良反应的发生。
Zhao Q, et al., Signal Transduct Target Ther. 2023
IVDR是多种因素共同作用的结果,包括:
1)遗传学
编码药物代谢酶、转运蛋白和靶标的单个基因的变异会影响其代谢和对药物的反应,导致IVDR。
2)年龄
由于生理功能的差异,如肝肾功能、身体成分和激素水平的变化,年轻和老年患者可能会经历不同的药物反应。例如,老年患者可能肾功能和肝代谢降低,导致PK改变。
3)性别
男性和女性之间的生物差异会影响药物反应。由于身体成分和激素水平的差异,女性可能有更高的药物浓度,这将影响药物代谢和清除。
4)生活方式
饮食、运动、吸烟等因素,酒精摄入可以通过改变药物代谢和从体内清除的方式来影响药物反应。
5)疾病状态
患有某些疾病或病症的患者可能会因器官功能变化、药物代谢改变或药物受体表达改变而经历IVDR。例如,肝病患者的药物代谢可能会降低,肾病患者的药物清除率可能会降低。
6)药物-药物相互作用
同时使用多种药物可能会导致药物-药物的相互作用,从而改变药物的吸收、分布、代谢、排泄。
7)环境因素
暴露于环境毒素和污染物会影响药物代谢和清除,可能导致IVDR.
8)肠道微生物群
肠道微生物群也可能影响药物药代动力学(PK)和药效学(PD),个体之间肠道微生物群组成的变化会导致IVDR。
肠道微生物群与药物治疗之间的关联
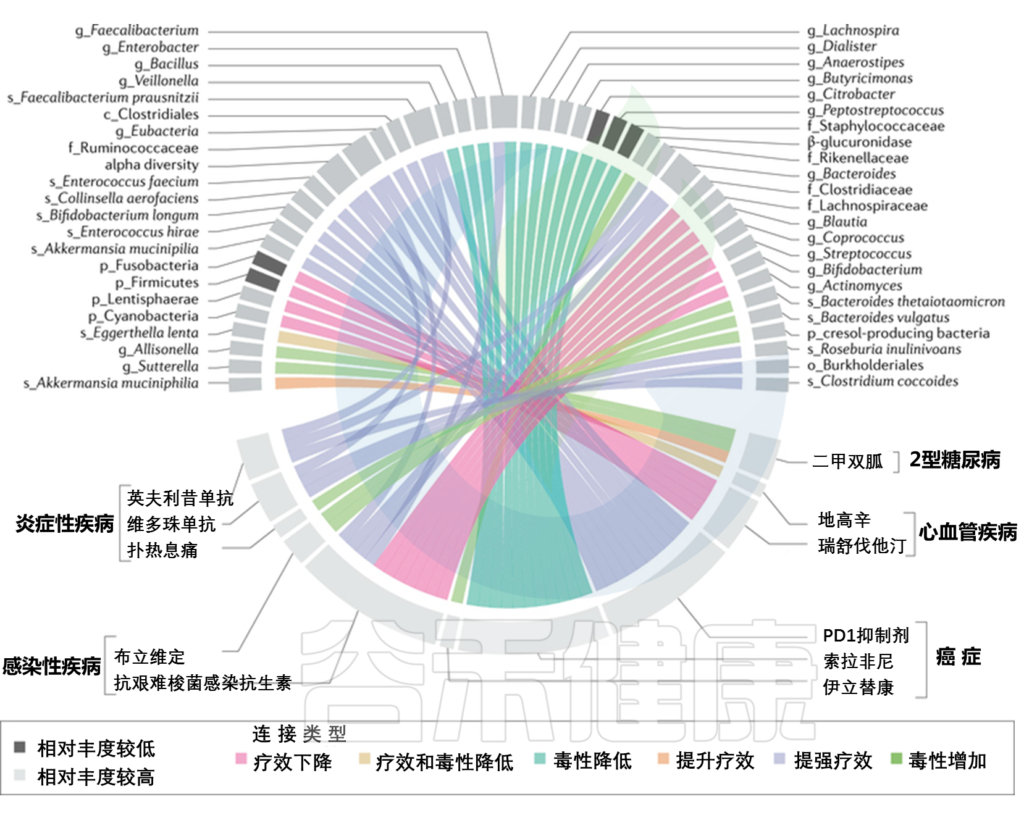
doi: 10.1038/s41575-021-00499-1
总体而言,了解这些因素对于优化药物治疗和将药物不良反应风险降至最低至关重要。
药物遗传学专注于识别影响药物代谢、转运、药效学 (PD) 等的遗传变异。
药物基因组学是一个更广泛的领域,考虑整个基因组及其对药物反应的影响。
尽管药物遗传学和药物基因组学取得了进展,人类基因组贡献的变异性可能在20%-95%,因此,仅遗传因素不足以解释药物反应的个体差异,其他因素,例如肠道微生物群也很重要。
肠道微生物群可以通过直接转化药物或调节宿主的代谢或免疫系统,来改变药物的药效学和药代动力学。
药物微生物组学一词,用来描述肠道微生物群和药物反应之间的相互作用,这些相互作用可以改变药效学(即药物吸收、分布、代谢、排泄及其血浆药物浓度动态的变化)或药代动力学(即药物靶点或生物途径的变化,导致生物体对药物作用的不同易感性)。
接下来,我们就从人体八大系统的具体疾病中来一一了解,微生物群与药物之间的相互作用。了解这种相互作用有助于开发针对微生物群的方法,以提高药物疗效并减少不良反应。
现代医疗体系下的药物种类很多,我们按照以下几大类来介绍常见的药物:心血管系统、内分泌系统、消化系统、神经系统、运动系统、泌尿生殖系统、呼吸系统、癌症。
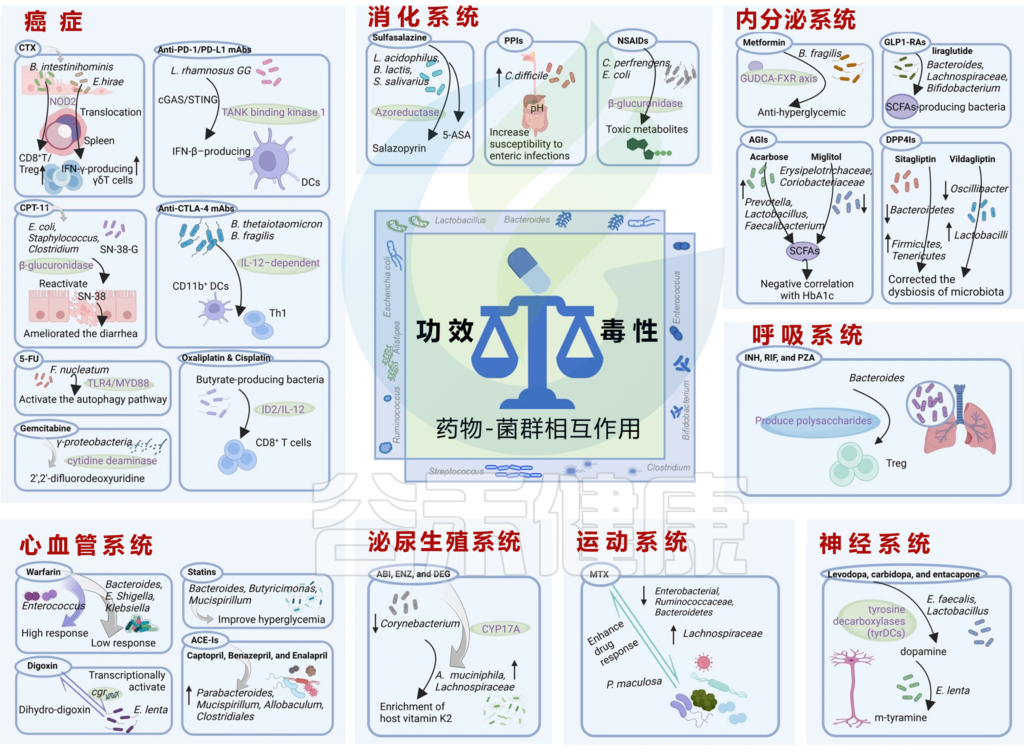
Zhao Q, et al., Signal Transduct Target Ther. 2023
华 法 林
华法林:防治血栓,但治疗窗口有限
华法林是一种维生素K拮抗剂,用于预防和治疗血栓形成和栓塞的疾病,然而,其治疗窗口有限,IVDR明显,促使许多研究确定影响治疗结果的遗传因素。
对华法林低反应者,志贺氏菌等菌群较多
已知肠道微生物群和维生素K代谢之间有联系。一项研究首次建立了肠道微生物群和IVDR与华法林之间的联系。针对200名对华法林有不同程度反应的患者的试验表明,拟杆菌、志贺氏菌、克雷伯氏菌在低反应者中表现突出。
华法林是通过抑制维生素K的作用,来防止血液凝结,维生素K参与合成凝血因子,在血液凝固过程中起着重要的角色,而志贺氏菌具有产生维生素K所必需的酶,可能出现一边抑制一边合成的情况,这可能是患者对华法林反应较弱的原因。相比之下,肠球菌与对华法林的反应增加有关。
▸
还需要更多的研究来证实和完善结果。
他汀类药物
他汀类药物:降胆固醇、抗炎、抗血栓
他汀类药物通常用于降低“坏”胆固醇,治疗心血管疾病。通过抑制HMG-CoA,减少肝脏中胆固醇的从头合成。
注:“坏”胆固醇,是指低密度脂蛋白胆固醇,简称LDL-C。
HMG-CoA,3-羟基-3-甲基戊二酰辅酶A,是一种参与胆固醇和同化代谢的重要酶。
近几十年的研究表明,他汀类药物具有许多多效性作用,包括抗炎、抗血栓形成和抗氧化,这可能与其心血管保护特性有关。他汀类药物可抑制Rho激酶和Rac1蛋白的功能,这两种蛋白与动脉粥样硬化的进展有关,动脉粥样硬化是心血管疾病的主要危险因素。他汀类药物还激活PPAR-γ.
注:PPAR-γ,过氧化物酶体增殖物激活受体γ,这是一种有助于调节炎症、脂肪代谢和糖代谢的核受体。
他汀类药物可以改变肠道微生物群的结构
一项研究发现,他汀类药物治疗显著增加了某些抗炎相关菌(如拟杆菌、Butyricimonas、 Mucispirillum),这可能有助于改善高血糖。
另一项研究表明,他汀类药可能通过PXR(孕酮X受体)介导的机制改变胆汁酸的复杂性,从而导致肠道失调,带来不良影响。
他汀类药物→改变胆汁酸→菌群失调
胆汁酸是在肝脏中从胆固醇合成的,对膳食脂肪的消化和吸收至关重要。他汀类药物通过直接抑制新生胆固醇的合成,可以抑制肝脏中的胆汁酸合成,从而改变人体肠道中的胆汁酸。肠道中胆汁酸的数量和结构的改变,可能通过改变肠道菌群直接或间接诱导出复杂而显著的生理反应。
▸
要更多的研究了解他汀类药物治疗对肠道微生物组的长期影响,以及这些变化是否始终对心血管疾病有益。此外,评估他汀类药物治疗和IVDR的可能风险和益处至关重要。
地高辛
地高辛:提高心脏泵送效率,但对10%患者无效
地高辛,是一种可以提高受损或虚弱心脏泵送效率的药物,从而提高临床稳定性和运动能力。然而,在低流量充血性心力衰竭中,由于特定菌株的失活,地高辛可能对10%的患者无效,这突出了IVDR对人类和微生物群的重要性。地高辛的失活是通过Eggerthella lenta将地高辛转化为失活状态二氢地高辛介导的。
E. lenta:可能成为地高辛失活的微生物标志物
在另一项开创性研究中,发现一个编码E. lenta细胞色素、心脏甙还原酶(cgr)的操纵子,被鉴定为地高辛转录激活,表明这可能是地高辛失活的可预测的微生物组生物标志物。
精氨酸增加→调节E.lenta→抑制地高辛失活
研究人员还探索了内源性地高辛相关物质可能被选择失活的可能性,并确定了合理的饮食干预是否可以调节体内地高辛的灭活。如前所述,E.lenta的增殖与精氨酸有关,精氨酸不仅能改善增殖,还能防止地高辛失活。因此,无论是饮食还是微生物群产生的精氨酸浓度的增加,都可能抑制这种不需要的微生物群功能。
饮食中增加蛋白质→提高地高辛水平
为了验证这一假设,研究人员使用两种不同饮食的小鼠进行了一项体内实验:一种不含任何蛋白质,另一种含有蛋白质总热量摄入的20%。结果表明,蛋白质摄入的增加显著提高了血液和尿液中的地高辛水平,但仅在携带地高辛减少菌株的小鼠中。
▸
总之,这些发现对受益于地高辛治疗的心力衰竭患者,具有重要的临床应用价值。此外,鉴定药物失活的预测性微生物群标志物,可能有助于制定个体微生物群落为基础的个性化治疗指南。
血管紧张素转换酶抑制剂(ACE-Is)
一代ACE-Is药:卡托普利,降压
卡托普利是第一代ACE-Is药物,通过抑制中枢和外周部位的肾素-血管紧张素系统(RAS)用于治疗高血压。除了其抗高血压作用外,卡托普利也会影响肠道微生物群的组成。研究表明,卡托普利在停药后保持其抗高血压活性。
一项用卡托普利治疗大鼠的实验中,下列菌群丰度增加:
此外,卡托普利可减少自身区域的神经元炎症,并减少交感神经驱动,它平衡了肠道微生物群。
最近的研究结果还表明,接受卡托普利治疗的母亲通过重建肠道微生物群、改善肠道病理和通透性以及恢复雄性后代失调的肠脑轴,具有持续的降压作用。
卡托普利治疗的怀孕大鼠中,梭菌属和梭菌目的存在率较高
与没有治疗的怀孕大鼠相比,用卡托普利治疗的怀孕大鼠在肠道微生物群中下列梭菌目平均丰度更高,例如:
因此,卡托普利可能会影响人类肠道微生物群的生长和组成,从而改变药物的有效性。
二代ACE-Is药:贝那普利和依那普利,治疗心血管疾病
贝那普利和依那普利是第二代血管紧张素转换酶抑制剂,主要用于治疗心血管疾病,如动脉高血压、充血性心力衰竭。这些药物通过抑制ACE的活性发挥作用,导致血管紧张素II生成减少。
注:ACE,血管紧张素转化酶,参与调节血管张力、血压以及体液平衡等生理过程。
血管紧张素Ⅱ,是一种使血管变窄并升高压力的激素。
最近研究表明,贝那普利和依那普利都有能力对肠道微生物群产生积极影响。
贝那普利治疗:促进肠道微生物群结构的恢复
贝那普利主要在肝脏中代谢,并转化为二酸贝那普利的形式,这会影响肠道微生物群。
依那普利治疗:改变肠道菌群,降低TMAO
依那普利可以降低血液中TMAO的水平。高浓度的TMAO与心血管疾病的高发病率有关。依那普利通过改变肠道微生物群的组成和控制尿液中甲胺的排泄,来影响血浆TMAO水平。
注:TMAO,三甲胺N-氧化物,一种由肠道微生物群代谢某些营养物质产生的化合物
心血管系统:药物-微生物相互作用

Zhao Q, et al., Signal Transduct Target Ther. 2023
▸
这些发现表明,ACE-Is可能在改善肠道微生物群组成和心血管健康方面有效。通过促进肠道微生物群结构的恢复和降低TMAO水平,贝那普利和依那普利可能有助于降低心血管疾病的风险。
其他降血压药物:药物-微生物相互作用
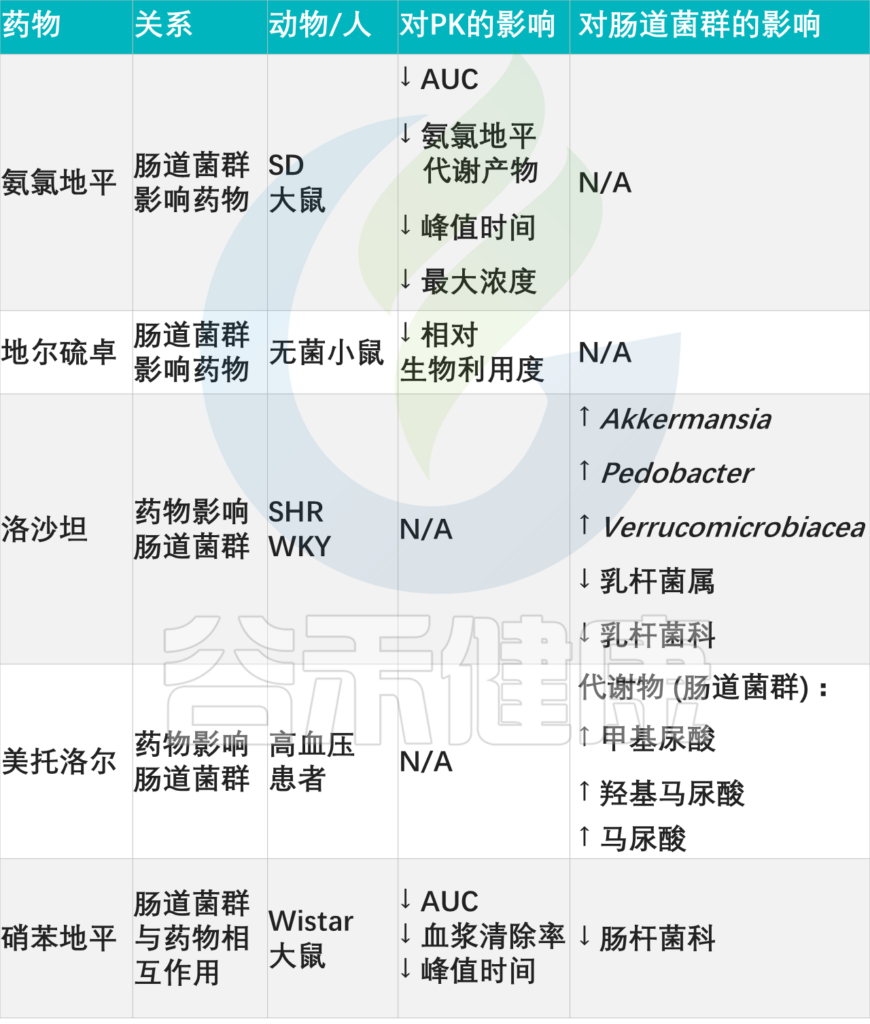
doi: 10.3389/fmed.2021.742394
内分泌系统是一个复杂的腺体系统,产生激素,负责调节各种身体功能,目前的证据表明,微生物群在内分泌系统的调节中发挥着重要作用,并可影响内分泌疾病的IVDR。
下面列出抗糖尿病药物和肠道微生物群之间相互作用的几个案例。
二甲双胍
二甲双胍是一种常见的抗糖尿病药物,也可以减少肥胖、降低心血管疾病和癌症的发病率等。
研究逐步发现,肠道微生物群调节葡萄糖代谢和能量平衡的机制,特别是可能参与IVDR的机制。
肠道菌群如何影响二甲双胍的抗高血糖作用
将未经治疗的2型糖尿病患者的粪便,移植到无菌小鼠身上,与对照小鼠相比,葡萄糖耐量显著改善。确定A.muciniphila及其金属结合蛋白的表达,是二甲双胍-微生物群相互作用的关键因素。
另一项研究表明,二甲双胍的抗高血糖作用涉及一种新的机制——脆弱拟杆菌-GUDCA-FXR轴。该途径依赖于肠道微生物群,并涉及肠道FXR信号的激活,以改善代谢功能障碍。发现二甲双胍可减少脆弱拟杆菌,防止GUDCA降解。
注:GUDCA,甘氨熊脱氧胆酸,一种FXR的拮抗剂
二甲双胍→抗炎→对心血管疾病的保护作用
包括激活AMPK途径和抑制NF-κB途径。越来越多的证据表明,微生物群介导的途径可能与二甲双胍的治疗作用有关。例如,二甲双胍治疗引起的肠道微生物群结构变化可能导致葡萄糖、激素、胆汁酸、短链脂肪酸和免疫的改变。
二甲双胍→调节肠道菌群→改善认知功能
二甲双胍不仅具有抗炎和抗氧化作用,还与神经保护有关,有研究表明二甲双胍的使用与2型糖尿病患者患痴呆症的风险降低有关,轻度认知障碍得到改善。这其中部分有益作用通过肠道微生物群的组成和功能来调节,从而增加短链脂肪酸和胆汁酸的产生。
二甲双胍对肠道菌群的影响

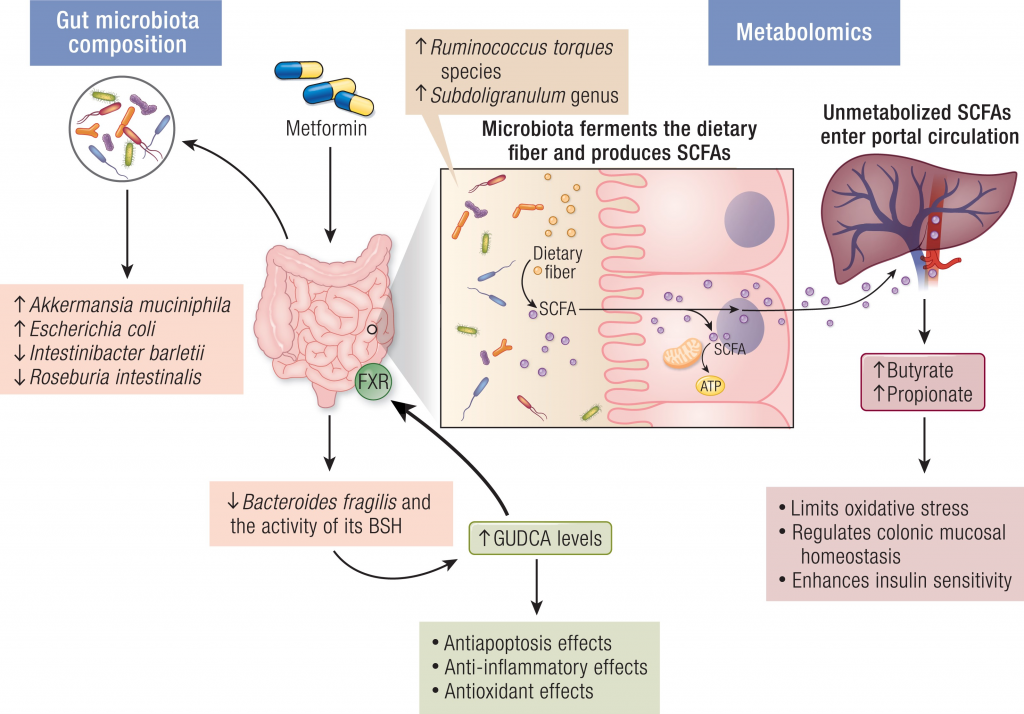
doi.org/10.1210/endrev/bnad029
▸
总的来说,新出现的证据表明,微生物群介导的肠道微生物群结构变化机制,可能是其多效性作用的原因。这对开发针对肠道微生物群及其与IVDR相互作用的新疗法具有重要意义。
α-葡萄糖苷酶抑制剂(AGI)
AGI是一类改变小肠中碳水化合物吸收和代谢的药物。这些药物会显著影响肠道微生物群的结构和变异性,这可能对管理代谢疾病(如2型糖尿病)有重要意义。阿卡波糖和米格列醇是两种常用的AGI。
阿卡波糖:产短链脂肪酸菌,乳杆菌 ↑↑
拟杆菌科、肠杆菌科、部分梭菌属 ↓↓
在体外,实验表明,阿卡波糖通过抑制麦芽糖导入物选择性地抑制大肠杆菌的生长,而临床试验表明,阿卡波糖增强了产短链脂肪酸菌的丰度,例如普雷沃氏菌属、乳杆菌属、粪杆菌属(Faecalibacterium)。短链脂肪酸对代谢健康有益,用乳杆菌治疗可以降低血糖。
此外,一项研究表明,在肠道微生物群中发现的Dialister在阿卡波糖治疗后增强,并与HbA1c呈负相关,这表明它可能在控制葡萄糖代谢中发挥作用。阿卡波糖也被证明可以增加双歧杆菌和乳杆菌,并减少高脂血症或2型糖尿病患者粪便中有害的拟杆菌科、肠杆菌科和卵磷脂酶阳性梭菌。
注:HbA1c在医学中指糖化血红蛋白,以反映最近两到三个月内的平均血糖水平
米格列醇:抗炎,逆转高能量饮食引起的菌群变化
米格列醇在小鼠中具有抗炎特性,可以抑制炎症反应的组织学和分子指标,并减少高脂肪高糖饮食产生的肠道转运时间。摄入高能量饮食会增加Erysipelotrichaceae和Coriobacteriaceae,但米格列醇可以逆转这种作用。据推测,肠道炎症的减少与肠道微生物群的这些变化有关。
▸
总的来说,AGI特别是阿卡波糖,可能有助于重建2型糖尿病患者的肠道微生物群平衡。通过增加产短链脂肪酸菌和减少潜在的有害菌,这些药物可能会对代谢健康产生积极影响。需要更多的研究来充分阐明其影响。
胰高血糖素样肽-1受体激动剂
GLP-1(胰高血糖素样肽-1)是肠内分泌细胞通过饮食产生的一种肠内激素,GLP-1在增强葡萄糖诱导的胰岛素分泌、抑制胰高血糖素分泌、抑制食欲和胃排空方面的作用,使其成为治疗糖尿病和超重的有力靶点。
最近的研究强调了肠道微生物群通过诱导GLP-1分泌来调节饱腹感和葡萄糖稳态的功能,GLP-1受体激动剂(如利拉鲁肽)作为一类新兴的抗糖尿病药物,已被证明可以调节肠道微环境,并改变肠道微生物群组成。
利拉鲁肽:改变厚壁菌门/拟杆菌门比例,有争议
通常,厚壁菌门与拟杆菌门的比例是肠道微生物群结构的重要指标。这在我们之前的一篇文章有讲述这方面内容,详见:
研究表明,利拉鲁肽可以使正常体重的糖尿病小鼠的肠道菌群比例向瘦体比例转变,从而使厚壁菌门与拟杆菌门的比例更高。
有趣的是,另一项研究表明,利拉鲁肽降低了厚壁菌门与拟杆菌门的比例,以降低单纯超重和糖尿病超重个体的体重,而与血糖状况无关。
这种差异可能归因于不同水平的高血糖和使用的不同模型系统。
GLP1受体激动剂已被证明可极大改变糖尿病雄性大鼠的肠道微生物群结构。这些变化包括选择性增强几种产短链脂肪酸菌,如拟杆菌、毛螺菌科和双歧杆菌。此外,GLP1受体激动剂可至少部分重建肠道微生物群的稳态。
二肽基肽酶-4抑制剂(DPP4Is)
DPP4Is(例如西格列汀和维达格利汀)是用于2型糖尿病的一线抗2型糖尿病药物。DPP4Is主要通过抑制GLP-1降解来降低血糖。先前的研究表明,肠道微生物群与DPP-4相关功能可能是DPP4Is的目标,可能为DPP4Is调节肠道微生物群不一致性开辟新的治疗应用。
研究表明,DPP4Is可以通过增加拟杆菌门来改善血糖控制,从而大大恢复高脂饮食诱导的肠道微生物群改变。
西格列汀:产短链脂肪酸菌↑↑适度恢复菌群失调
西格列汀对肠道微生物群影响的研究表明,拟杆菌门减少,厚壁菌门和软壁菌门(Tenericutes)增加。然而,西格列汀适度恢复了2型糖尿病大鼠的微生物群失衡,并改变了产短链脂肪酸菌的丰度。
维达利汀:乳杆菌↑↑ Oscillibacter↓↓
类似地,维达利汀给药与糖尿病大鼠的拟杆菌门增加和厚壁菌门减少,厚壁菌与拟杆菌门比例降低有关。此外,维达利汀通过调节肠道微生物群产生益处,导致乳杆菌增加和Oscillibacter减少。为了阐明变异机制,研究人员通过实验证实,维达利汀抑制盲肠内容物中的TLR配体,并恢复抗菌肽水平和回肠隐窝深度。研究还表明,DPP4Is可能通过对肠道微生物群的影响,间接减少肝脏中促炎细胞因子的分泌。
▸
总的来说,这些研究强调了DPP4Is对肠道微生物群的重要性,并揭示了改善葡萄糖稳态和IVDR的有希望的策略。
内分泌系统:药物-微生物相互作用

Zhao Q, et al., Signal Transduct Target Ther. 2023
消化系统是药物微生物组学最广泛的领域之一,对药物代谢和疗效至关重要。肠道微生物群会影响用于治疗各种消化系统疾病的药物的结果,例如,炎症性肠病(IBD)和肠易激综合征(IBS)。IBD患者的肠道微生物群组成和功能发生了改变,这可能会影响IVDR。
柳氮磺吡啶(SAS)
SAS是一种最初开发的药物,旨在治疗细菌感染引起的炎症。后来发现它在治疗溃疡性结肠炎方面有效。该药物由氨基水杨酸盐和磺胺吡啶组成,它们通过偶氮键连接在一起。SAS的一个独特特性是它不会在上游胃肠道中快速吸收。相反,它被结肠中的肠道微生物群分解为其各个组成部分。
随后,磺胺吡啶被吸收到血液中,而美沙拉秦(5-氨基水杨酸,5-ASA)可以在结肠中失活。
SAS+益生菌:5-ASA和磺胺嘧啶恢复
一项针对健康受试者的PK研究表明,肠道微生物群对激活SAS至关重要。这一发现或许可以解释为何该药物在治疗溃疡性结肠炎(UC)方面比克罗恩病(CD)更为有效。
通过给予益生菌菌株(例如嗜酸乳杆菌L10、乳酸双歧杆菌B94、唾液链球菌K12),可以增强肠道菌群SAS的功能。
在一项体外试验中,研究人员在厌氧条件下将大鼠结肠的内容物与SAS单独或SAS与益生菌联合孵育。发现益生菌具有偶氮还原酶活性,使其能够代谢SAS。经与SAS和益生菌共同培养后,样本中的5-ASA和磺胺嘧啶含量得到了更大程度的恢复。
尽管SAS是一种有效且廉价的溃疡性结肠炎治疗方法,但一些患者在使用时报告了不良反应,如恶心、皮疹和厌食症。这可能是由SAS通过肠道微生物群代谢引起的。因此,SAS不如其他溃疡性结肠炎治疗方法受欢迎。
质子泵抑制剂
PPIs是广泛用于治疗酸性疾病的药物,包括消化性溃疡、胃食管反流、消化不良、胃十二指肠病、出血。
PPI 有效,但长期使用可能存在不良反应
基于其疗效和安全性特点,质子泵抑制剂治疗在近几十年快速扩大。然而,与长期使用PPIs相关的安全性和可能的不良反应仍存在争议。据估计,高达70%的PPIs处方被认为是不必要的。一旦开始使用,很少重新评估PPIs的原始适应症,试图停药后不必要的长期使用也随之产生。
PPI:肠道菌群多样性↓↓ 部分口腔菌群增加↑↑
荷兰一项基于患者的大规模临床试验表明,PPI与肠道微生物群多样性减少和分类学变化之间存在很大相关性。
分析来自不同队列的16S数据发现,PPI的使用导致部分肠道微生物群丰度下降,口腔微生物群增加。

例如,肠杆菌科、肠球菌科和乳杆菌科增加,瘤胃球菌科和双歧杆菌科减少。而典型口腔微生物群的改变表现为下列菌群增加:
此外,观察到的变化似乎是PPI的一类功效,剂量的增加与微生物群的更大变化有关。
PPI的使用与可预测的功能变化
最近的研究表明,PPI的使用与24个分类群和133个途径高度相关,具有可预测的功能变化,包括脂肪酸生物合成、脂质生物合成和L-精氨酸的生物合成增加,嘌呤脱氧核糖核酸的降解。
PPI→胃酸↓↓→增加肠道感染的易感性
另一项受流行药物直接影响肠道微生物群的体外实验表明,生长速度发生了显著变化,这意味着PPIs与细菌H+/K+ATP酶的结合可能介导直接影响。 PPIs引起的胃酸减少,使口腔微生物群能够定植肠道微生物群,导致分类稳态改变,并可能增加对肠道感染的易感性,例如艰难梭菌、弯曲杆菌和沙门氏菌。
儿童期使用PPI,可能与日后肥胖相关
此外,PPI的启动和停用可能导致肠道微生物群的改变,可能加剧肝硬化。长期使用PPI,特别是在儿童时期,会导致肠道微生物群的永久性改变,可能导致日后的肥胖。
▸
虽然PPI被广泛认为是无害和有效的,但临床上应该重新评估其广泛、长期的影响和OTC的可及性。PPI使用的快速而广泛的转变导致了肠道微生物群的变化,影响了多达五分之一的人群。仔细评估PPIs诱导的肠道微生物群改变带来的长期影响,特别是在早期发育过程中,以及对晚年健康和疾病的潜在影响,这一点很重要。
非甾体抗炎药
非甾体抗炎药通常用于缓解疼痛和炎症,但它们的使用会导致不良反应,如胃溃疡和小肠粘膜损伤。
这是由于细菌葡萄糖醛酸酶在肠道中代谢非甾体类抗炎药,类似于CPT-11的情况,它将药物转化为损害肠道粘膜的有毒代谢产物。肝脏中产生的非甾体抗炎药的葡萄糖醛酸苷通过胆汁到达肠道,在胆汁中细菌GUSs将其水解为苷元。这些苷元随后被细胞色素P450重吸收并转化为潜在的细胞毒性中间体,从而引起肠道毒性。
注:CPT-11属于盐酸伊立替康注射液
与CPT-11一样,最近的一项研究表明,Inh1(抑制剂1)是一种新型的微生物特异性GUSs抑制剂,可以减少双氯芬酸的肠道药物不良反应。这表明抑制GUSs是减少某些药物不良反应的一种很有前途的方法。
虽然肠道微生物群产生对生物体具有有益功能的GUS,但条件致病菌或肠道致病菌,如产气荚膜梭菌和大肠杆菌,负责对药物进行去葡萄糖醛酸化反应,产生有害代谢产物。这是由于GUSs的细胞内变化,包括构象、疏水性和迁移率的变化。细菌GUSs诱导的ADR的增强潜力,尤其是肠道损伤,并非NSAIDs和CPT-11独有。
其他药物,包括抗肿瘤酪氨酸激酶抑制剂,瑞格菲尼和静脉补剂类黄酮,是GUSs 的底物。这些证据强调了了解细菌酶在IVDR中的重要性,以制定减少药物不良反应的策略的价值。
消化系统:药物-微生物相互作用
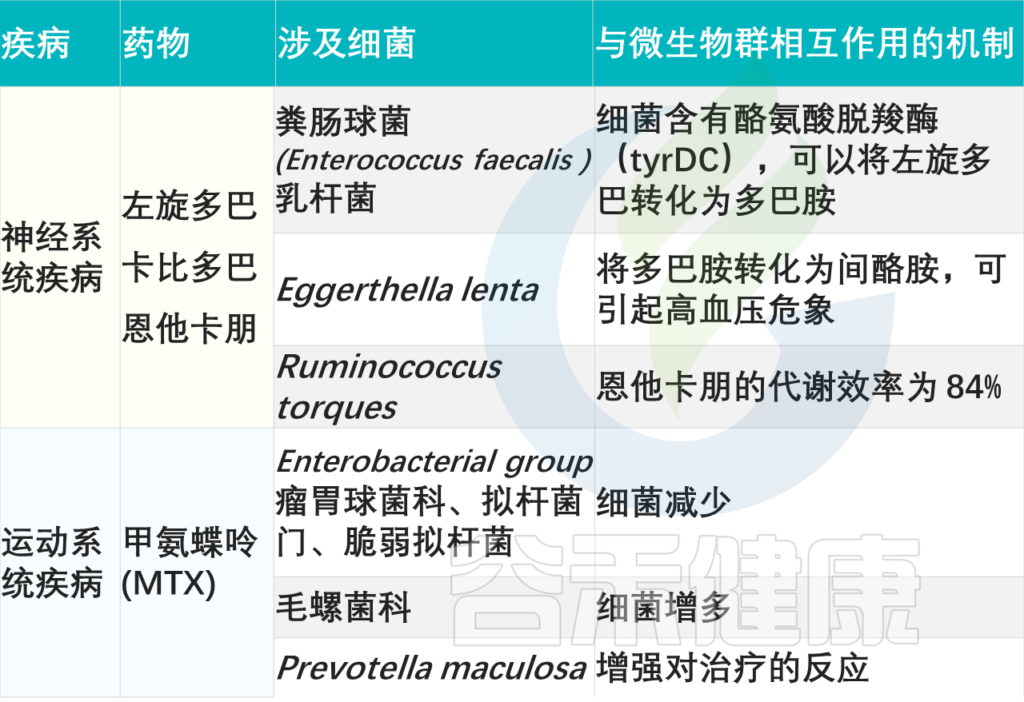
Zhao Q, et al., Signal Transduct Target Ther. 2023
初步研究表明,微生物群可能在调节神经系统IVDR中发挥关键作用。其中一个例子是使用益生菌来提高抗抑郁药物的有效性。多项研究表明,某些肠道微生物群菌株可以产生神经递质,如血清素和γ-氨基丁酸(GABA),可以调节情绪和焦虑。
通过补充益生菌,研究人员希望增加这些神经递质的可用性,改善对抗抑郁药治疗的反应。另一个领域是微生物群对神经退行性疾病的影响,例如帕金森病,常用的治疗药物是左旋多巴。
左旋多巴、卡比多巴和恩他卡朋
左旋多巴是一种治疗帕金森病的药物,帕金森病是一种神经系统疾病,表现为震颤、僵硬和行动困难。左旋多巴口服,必须通过小肠重新吸收才能到达大脑,在那里它被酪氨酸脱羧酶(tyrDC)转化为多巴胺。该药物的有效性取决于大脑的生物利用度,该药物通常与儿茶酚胺代谢抑制剂(如卡比多巴和戊卡彭)一起使用,以抑制脑外代谢。
E.lenta 代谢左旋多巴,可能与高血压风险相关
新出现的证据表明,肠道微生物群可以代谢左旋多巴。某些物种,如粪肠球菌(E.faecalis)和乳杆菌含有酪氨酸脱羧酶。然而,这种微生物群代谢减少了大脑中可获得的左旋多巴的量,导致药效降低。此外,左旋多巴的微生物群代谢也会导致药物不良反应,因为E.lenta和其他十个物种可以进一步将多巴胺转化为间酪胺,从而导致高血压危机。
残留左旋多巴也可以与微生物群相互作用
绝大多数左旋多巴通过肠道氨基酸转运载体输送到循环系统,还有10%左右残留,也可以与微生物群相互作用,通过其代谢产物影响肠道稳态。Clostridium sporogenes 能够将L-多巴胺脱氨化为3-(3,4-二羟基苯基)丙酸,后者可以体外抑制回肠运动。
粪肠球菌:98.9%效率代谢左旋多巴和恩他卡朋
肠道微生物群也可以直接代谢卡比多巴和恩他卡朋。例如,粪肠球菌能以98.9%的效率代谢左旋多巴和恩他卡朋。反过来,恩他卡朋会减少一些物种,例如Ruminococcus torques,后者仍能以84%的效率代谢恩他卡朋。
▸
这些发现说明了肠道微生物群和IVDR之间的复杂相互作用,这可能会影响药物的疗效和安全性。这些有利于拓宽我们对药物-微生物群相互作用的了解,并探索神经和精神疾病的新治疗策略。
运动系统的健康可能受到各种疾病的影响,例如骨关节炎、类风湿性关节炎和骨质疏松症,这些疾病会导致疼痛、僵硬和活动能力下降。近年来,人们越来越关注肠道微生物群在这些疾病的病理学和治疗中的影响,如甲氨蝶呤、非甾体抗炎药等。
甲氨蝶呤(MTX)
甲氨蝶呤是一种用于治疗自身免疫性疾病(如类风湿关节炎)的强效细胞毒性药物。其潜在作用基于抑制二氢叶酸还原酶和胸苷酸合成酶,从而防止嘧啶和嘌呤的从头合成。虽然甲氨蝶呤是有效的,但它也有很高的不良反应发生率,包括胃肠道、血液学、肾毒性和肝毒性。
甲氨蝶呤:E.faecium↓↓ Lachnospiraceae↑↑
可能影响对甲氨蝶呤反应的一个潜在因素是肠道微生物群。研究表明,甲氨蝶呤治疗可以改变肠道微生物群的组成,降低肠道细菌的丰度,尤其是粪肠球菌(E.faecium)的丰度,并增加Lachnospiraceae的丰度。然而,部分有益菌(如瘤胃球菌科、拟杆菌科、脆弱拟杆菌)的减少可能会加剧胃肠道药物不良反应,特别是肠道粘膜炎。
甲氨蝶呤和肠道微生物群之间的联系是双向的
肠道微生物群的变异性可能会影响甲氨蝶呤的治疗反应。研究发现,肠道微生物群多样性较高的患者,具有丰富的Prevotella maculosa,对甲氨蝶呤的反应更好。尽管看起来不错,但甲氨蝶呤的应答率极其不稳定,介于10%-80%之间,只有40%的患者能够达到治疗所需的药物浓度,并且不良反应显著。
▸
已经探索了遗传因素来预测甲氨蝶呤的IVDR,但尚未达到临床相关性。需要更多的研究来评估。肠道微生物群可能在甲氨蝶呤的PD中发挥重要作用,进一步的探索可能有助于患者个性化治疗。
神经、运动系统:药物-微生物相互作用

Zhao Q, et al., Signal Transduct Target Ther. 2023
泌尿生殖系统的微生物群会影响几种药物的疗效和毒性,导致该系统出现了药物微生物组学领域。特别是,泌尿生殖微生物群会影响雄激素剥夺治疗(ADT)的有效性,这是癌症的常见治疗方法。ADT通常涉及使用雄激素合成抑制剂(如醋酸阿比特龙),或雄激素受体拮抗剂(如恩扎鲁胺、醋酸德格雷)。
醋酸阿比特龙、恩扎鲁胺、醋酸地加瑞克
最近的研究揭示了ADT对免疫系统和肠道微生物群的影响,以及这如何影响前列腺癌症的进展。
醋酸阿比特龙是ADT中广泛使用的药物,研究强调了胸腺依赖性T细胞在调节前列腺癌进展中的关键作用,由于CD4+和CD8+T细胞的消耗,导致IVDR控制的肿瘤发展的部分减少。
醋酸阿比特龙→ A.muciniphila ↑↑ → 维生素K2 ↑↑
前列腺癌患者接受醋酸阿比特龙治疗后,重塑肠道微生物群并促进抗炎菌A.muciniphila的增殖,从而提高微生物群产生维生素K2。
此外,研究表明,具有较高α-多样性的微生物群和特定的肠道微生物群,如A.muciniphila,与更好的疗效相关。
进一步研究发现,接受口服雄激素靶向治疗(ATT)的男性的泌尿生殖微生物群组成,与接受促性腺激素释放激素(GnRH)激动剂或拮抗剂单一治疗或不接受ADT的男性有所不同。例如,A. muciniphila、瘤胃菌科等能够合成类固醇的菌,在接受口服ATT治疗的男性的微生物群中丰富,这可能会影响疾病进展和免疫疗法。
肠道菌群→调节循环性激素→促进ADT耐药性
另一项研究表明,肠道微生物群可以通过对人体细胞的影响,也可以通过直接的生物转化或合成来调节循环性激素的水平,从而促进ADT耐药性。前列腺癌患者表现出更高水平的菌株,如A. muciniphila、瘤胃菌科,它们能够通过CYP17A1样细菌酶合成类固醇激素。使用CYP17A1抑制剂醋酸阿比特龙培养细菌可抑制雄激素生物合成。然而,微生物群对雄激素水平降低的反应导致能够合成雄激素的菌株的扩增,尚待阐明。
▸
总之,微生物群调节循环性激素的能力和能够合成雄激素的细菌的扩张,可以驱动对ADT的耐药性。未来的研究有必要澄清肠道微生物群、前列腺癌和治疗之间的复杂相互作用。
药物可能会影响呼吸微生物群的组成和功能,相反,微生物群可能会影响药物代谢和反应。例如,抗生素可以减少微生物群的多样性,并促进可能导致慢性呼吸道疾病的机会性病原体的生长。吸入皮质类固醇可以增加微生物群的多样性,降低致病菌的丰度,这可能有助于其治疗效果。
此外,呼吸道微生物群会影响药物向肺部的输送。最著名的例子之一是使用抗生素异烟肼(INH)、利福平(RIF)和吡嗪酰胺(PZA)进行抗结核治疗。
异烟肼、利福平、吡嗪酰胺
研究抗结核抗生素对结核分枝杆菌感染小鼠肠道微生物群结构的影响,发现在小鼠模型中,异烟肼、利福平、吡嗪酰胺治疗三个月会导致显著和持久的生态失调。肠道微生物群的组成发生了改变:
研究进一步表明,单剂量利福平降低了微生物群的多样性,而与未用抗生物治疗的小鼠的肠道微生物群相比,单独使用异烟肼或吡嗪酰胺会导致微生物群组成的改变。
具体而言,异烟肼富集Gordonibacter,而吡嗪酰胺富集Marvinbryanti。 结核病感染不会显著改变肠道微生物群组成,但它被发现可以调节粘膜免疫反应。
在人类中,一线抗结核药物治疗似乎对肠道微生物群的影响很小。然而,一些菌株可以调节宿主的免疫反应,如:
研究还发现,抗结核病药物可以减少肠道稳态所需的微生物群,增加复发结核病的风险。与无结核病患者相比,先前治疗的患者的T细胞对2型表位的识别能力也较差。人类肠道中的2型表位与分枝杆菌(mycobacteria)有关。
▸
抗结核病治疗的多年试验有助于更深入地了解微生物群在结核病进展中的重要性。抗结核抗生素和宿主调节剂的组合,可以缩短结核病的治疗时间,减轻严重程度,减少再次感染的风险。对宿主-病原体相关性、宿主免疫学和宿主靶向治疗的研究表明,这种方法可能对结核病的治疗有益。
呼吸系统:药物-微生物相互作用

Zhao Q, et al., Signal Transduct Target Ther. 2023
在癌症方面,肠道微生物群对于几种癌症的发病机制、进展和 IVDR 至关重要。这里介绍几种代表性药物和肠道微生物群在癌症患者中相互作用的机制,见下表。
Zhao Q, et al., Signal Transduct Target Ther. 2023
关于癌症药物与微生物群之间的相互作用,在我们之前的文章也有介绍,详见:
肠道微生物群通过酶修饰结构和生物活性或毒性的改变来影响药物的有效性和可接受性。肠道微生物群可以通过多种方式影响IVDR,包括以下:
1) 药物代谢的改变
微生物群可以影响体内代谢药物的酶的活性,从而影响药物的疗效和副作用。
2) 产生耐药性
随着时间的推移,微生物群会对药物产生耐药性,使其在治疗感染方面的效力降低。这种耐药性可能是由于微生物群DNA突变或从他人那里获得耐药性基因所致。
3) 药物靶点的修饰
微生物群可以修饰体内的药物靶点,使其不易受药物影响。如微生物群可以修饰氟喹诺酮类抗生素的靶位点,从而降低其结合和抑制酶的能力。
4) 与药代动力学相互作用
微生物群影响药代动力学,影响药物的疗效和安全性。例如,肠道微生物群可以改变药物的吸收、分布、代谢、排泄,从而改变其治疗效果和毒性。
5) 宿主微生物群的破坏
使用抗生素会干扰宿主微生物群中的自然生态系统,导致致病菌(如艰难梭菌)过度生长。这会导致感染和其他不良反应,从而影响药物安全。
6) 宿主免疫反应的调节
肠道微生物群可以调节宿主的免疫功能,这可能会影响靶向免疫系统的药物的活性和毒性。例如,肠道微生物群可以通过调节宿主免疫反应来调节免疫疗法在肿瘤治疗中的有效性。
7) 肠道屏障功能的调节
肠道微生物群可能影响肠道屏障的完整性,从而影响药物吸收和肠道-宿主相互作用。例如,肠道微生物群可以调节肠道中紧密连接蛋白的表达水平,从而影响药物的吸收。
肠道菌群编码的一些酶及其对药物的影响

Zhao Q, et al., Signal Transduct Target Ther. 2023
在一项重要全面的研究中,作者评估了1000多种药物的抗微生物特性,其中835种是影响人体细胞的靶向药物。其中,24%表现出抗菌作用,影响了至少50%的受试菌株的增殖。
药物影响肠道微生物群的机制有几个:
1) 直接抗生素效应
抗生素可以杀死包括有害菌和有益菌,可能导致肠道微生物群失衡,对人类健康产生负面影响。
2) 肠道运动能力改变
食物通过肠道的速度,会影响肠道微生物群的增殖和功能。某些药物,如阿片类止痛药,会减缓肠道运动,从而导致有害细菌过度生长。这可能会导致便秘和腹胀等胃肠道症状,在某些情况下还会导致感染(如艰难梭菌)。
3) 免疫功能的调节
肠道微生物群在调节肠道免疫方面具有关键功能。几种药物,如免疫检查点抑制剂,会影响肠道免疫,进而影响肠道微生物群。
4) pH值的变化
肠道的pH值会影响不同类型细菌的生长和存活。一些药物,如用于治疗胃酸倒流的质子泵抑制剂,可以改变肠道的pH值,从而影响不同微生物群的增殖,从而影响肠道微生物群的整体组成。
5) 干扰微生物代谢
肠道微生物群在代谢肠道中的化合物方面具有重要功能。一些药物,如非甾体抗炎药,会干扰微生物代谢,这可能会对肠道微生物群产生影响。特别是,非甾体抗炎药会干扰胆汁酸的肠道微生物群代谢,导致肠道微生物群的变化。
6) 饮食变化
某些药物,如泻药或抗腹泻药,会改变肠道的饮食环境。这可能会通过改变肠道微生物群生长和生存所需的营养素和其他化合物的可用性来影响肠道微生物群。
▸
药物对肠道微生物群的影响可能是积极的,也可能是消极的。例如,某些益生菌或益生元能够调节肠道微生物群,这可能对个体的健康产生积极影响。然而,服用对肠道微生物群产生负面影响的抗生素或其他药物可能导致微生态失调,对人类健康产生负面影响。
因此,必须仔细评估药物对肠道微生物群的潜在影响,并开发考虑个体肠道微生物群组成和功能的个性化药物治疗方法。
以上我们了解到微生物群和常见药物之间复杂的相互作用对药物效果的影响,肠道微生物群在药物治疗中的重要性意味着,肠道微生物群驱动的精准医疗,明确靶向肠道微生物群或将提高临床疗效。
未来多元化应用
通过测定肠道微生物群的组成、数量及代谢产物相对丰度,可以预测药物在体内的疗效和毒性。肠道微生物特征可作为非侵入性辅助诊断方法或提供预后评估。因此,开发能够预测药物疗效和毒性的改进生物标志物,对于成功实施精准医疗至关重要。
设计小分子抑制体内与药物生物转化为有毒代谢物相关的微生物代谢活性。
可以通过引入益生菌、益生元、粪菌移植等方式来调节微生物群的组成和比例,提高药物的生物利用度,减少不良反应。
药物微生物组学领域正在兴起
整合多个高通量多组学数据集,提供药物反应的个体差异的全面概况。这些将辅助全面揭示药物与微生物之间相互作用背后的决定因素,可以有效地满足对患者进行聚类和预测以及发现新生物标志物的需求。
多组学策略促进药物微生物组学走向人类精准医学

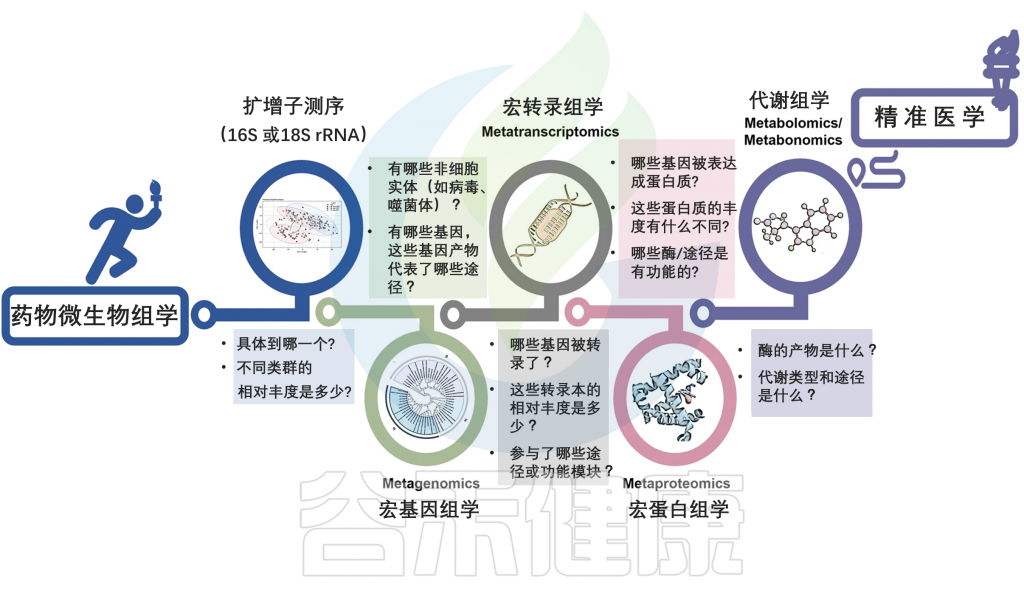
Zhao Q, et al., Signal Transduct Target Ther. 2023
高通量数据需要使用先进的计算技术通过人工智能算法进行建模。开发模拟药物与微生物相互作用的预测模型和软件对于减少研究时间和成本至关重要。
仍然存在一些挑战
——研究还需更加深入
对药物-微生物之间相互作用背后的机制仍然缺乏全面的了解。肠道菌群是一个复杂且不断变化的生态系统,受到饮食、年龄、性别、生活方式、宿主遗传学等多种因素的影响。很难将这些因素的影响与药物引起的微生物群变化分开。
——临床转化需要克服的障碍
例如,开发基于微生物群的药物反应生物标志物将需要大规模的验证研究和监管部门的批准。
许多药物微生物组学研究的样本量通常有限,可能限制其统计能力和概括研究结果的能力。需要更大规模的研究来确认和扩展初步结果。
应对这些挑战需要研究人员、临床医生和行业合作伙伴之间的合作,加深我们对药物-微生物相互作用机制的理解,并确保研究结果转化为安全有效的临床实践,为患者提供更好的治疗策略,共同推动精准医学领域的进步。
主要参考文献
Zhao Q, Chen Y, Huang W, Zhou H, Zhang W. Drug-microbiota interactions: an emerging priority for precision medicine. Signal Transduct Target Ther. 2023 Oct 9;8(1):386.
Rosell-Díaz M, Fernández-Real JM. Metformin, cognitive function, and changes in the gut microbiome. Endocr Rev. 2023 Aug 21:bnad029.
Weersma RK, Zhernakova A, Fu J. Interaction between drugs and the gut microbiome. Gut. 2020 Aug;69(8):1510-1519.
Schupack DA, Mars RAT, Voelker DH, Abeykoon JP, Kashyap PC. The promise of the gut microbiome as part of individualized treatment strategies. Nat Rev Gastroenterol Hepatol. 2022 Jan;19(1):7-25.
Chen HQ, Gong JY, Xing K, Liu MZ, Ren H, Luo JQ. Pharmacomicrobiomics: Exploiting the Drug-Microbiota Interactions in Antihypertensive Treatment. Front Med (Lausanne). 2022 Jan 19;8:742394.
Daisley BA, Chanyi RM, Abdur-Rashid K, Al KF, Gibbons S, Chmiel JA, Wilcox H, Reid G, Anderson A, Dewar M, Nair SM, Chin J, Burton JP. Abiraterone acetate preferentially enriches for the gut commensal Akkermansia muciniphila in castrate-resistant prostate cancer patients. Nat Commun. 2020 Sep 24;11(1):4822.
Jia L, Huang S, Sun B, Shang Y, Zhu C. Pharmacomicrobiomics and type 2 diabetes mellitus: A novel perspective towards possible treatment. Front Endocrinol (Lausanne). 2023 Mar 23;14:1149256.
Khan I. Drugs and gut microbiome interactions-an emerging field of tailored medicine. BMC Pharmacol Toxicol. 2023 Aug 30;24(1):43.
Pant A, Maiti TK, Mahajan D, Das B. Human Gut Microbiota and Drug Metabolism. Microb Ecol. 2023 Jul;86(1):97-111.
谷禾健康
为了使研究更系统更全面,越来越多的研究人员追求在多组学背景下解释分子数据。
OmicsNet (www.omicsnet.ca) 顺应而生,这是一个在线平台,允许用户轻松地构建、可视化和分析多组学网络。
OmicsNet 2.0在原有基础上进行了升级,主要改进了三个方面:
(1) 可视化分析中2D图形布局选项增加了11个,以及一个新颖的3D模块。
(2) 支持三种新的组学类型:
(3) 分析时将同时输出使用的R包OmicsNetR和历史命令,并生成共享链接可在网页查看和交互操作,从而提高研究的可重复性。
研究中使用OmicsNet 2.0对炎症性肠病(IBD)的数据进行多组学分析。
OmicsNet 2.0工作流程
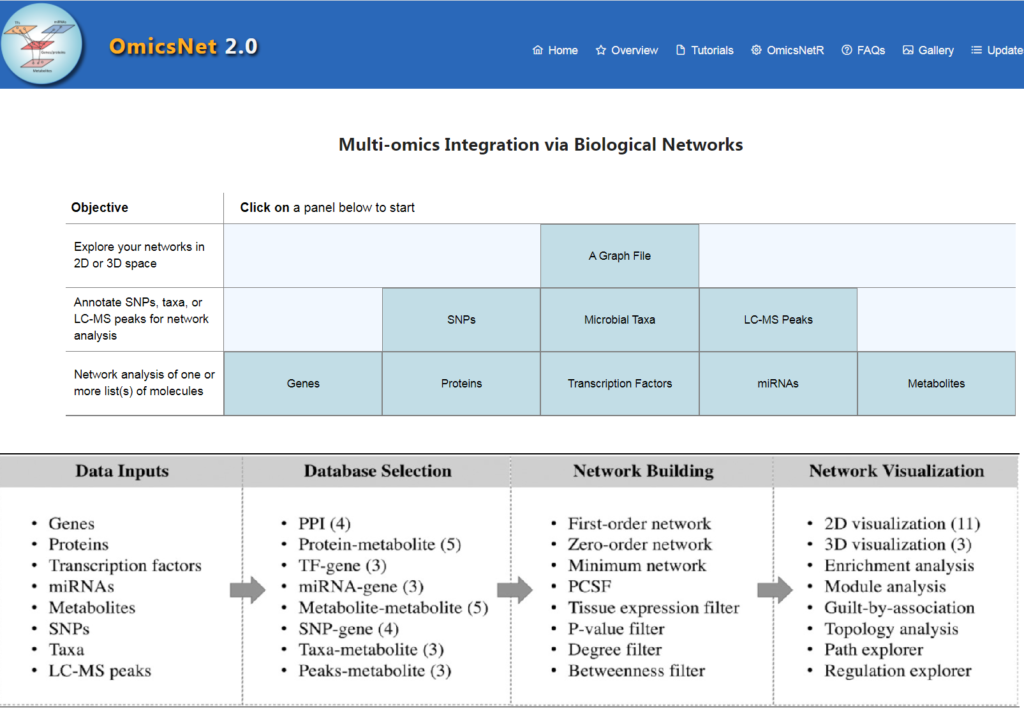

如图,主要为4个步骤:
OmicsNet 2.0 的改进
1、更新了分子相互作用数据库
包括PPI数据库(STRING,InnateDB和IntAct),TF-target数据库(TRRUST和JASPAR), miRNA-target数据库(TarBase和miRTarBase),代谢数据库(KEGG, Recon3和AGORA)。
2、支持三种新的组学类型
可支持来自遗传变异研究的SNP矩阵、来自非靶向代谢组学的 LC-MS 峰和来自微生物组分析的物种丰度矩阵数据的输入及互作分析。
对于那些对影响基因调控的变异感兴趣的用户,可以分别基于ADmiRE和SNP2TFBS将SNPs映射到miRNAs或TF结合位点。由此产生的网络可以通过proteins、miRNAs或TFs进一步扩展,以了解潜在的影响。
对于代谢组学分析,使用最近发布的NetID算法对LC-MS峰(m/z, retention time, intensity和p-value)进行数据处理,可选三种数据库(KEGG, PubChemLite_BioPathway和HMDB)进行注释。
使用Rcpp/C++ 引擎重新编写了核心算法,使其速度提高了10 倍以上。使用 lpsymphony 包进一步优化了整数线性规划。
直接上传物种丰度矩阵,可以使用贝叶斯逻辑模型预测潜在代谢物,该模型使用超过 6000 个高质量基因组规模的代谢模型进行训练。
3、代码开源
开放R包OmicsNetR(https://github.com/xia-lab/OmicsNetR),显示分析期间所执行的R命令。结果可输出为网页链接,有效期一个月。
4、减少假阳性
对于来自人类和小鼠的数据,提供了一个基于ENCODE、基因型-组织表达(GTEx)或人类蛋白图谱(HPA)的基因表达数据的过滤器,这些过滤器帮助研究人员专注于与生物相关的相互作用,并减少假阳性。
5、支持 2D 和 3D 网络可视化
2D模块可支持11种图形布局,3D模块可支持交互。图形化主要基于 igraph 包和 graphlayouts 包。
使用OmicsNet 2.0进行的IBD多组学研究
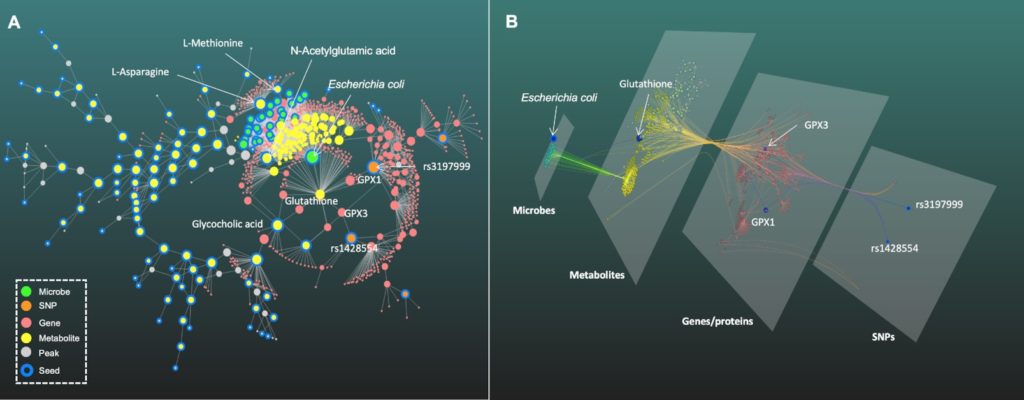
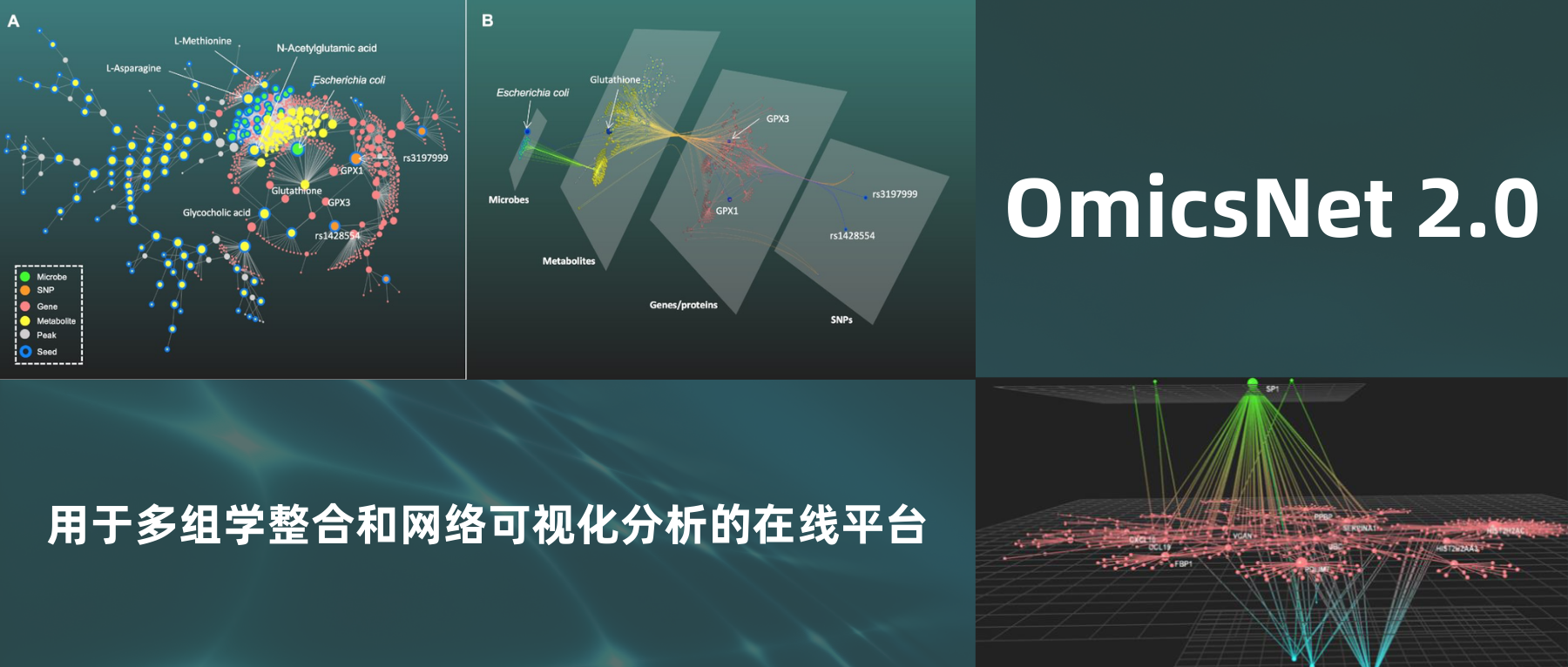
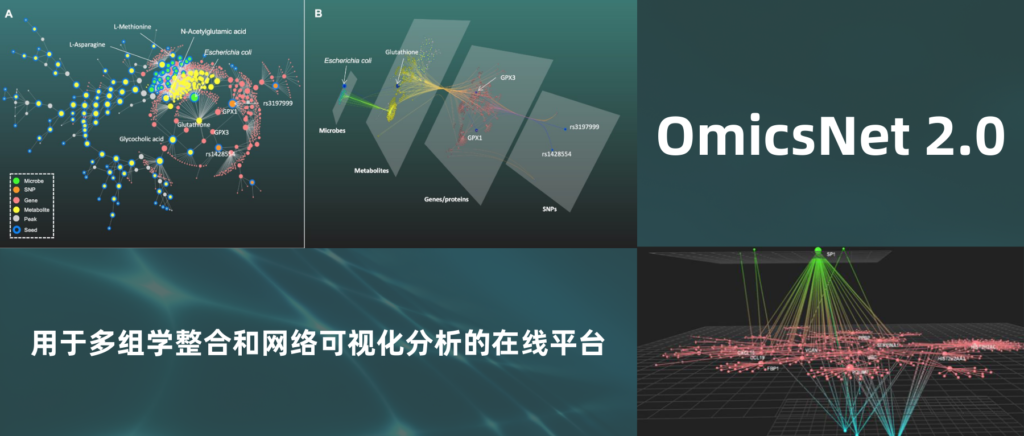
图A为2D视图,图B为3D视图
将获得的物种丰度矩阵,SNP矩阵和LC-MS峰数据上传至OmicsNet 2.0平台。
利用AGORA数据库预测潜在的微生物代谢物(potential score:0.9)。
利用PhenoScanner对基于eQTLs的基因进行SNP定位。
利用KEGG数据库对LC-MS峰进行注释。
通过添加代谢物-蛋白质相互作用,从SNPs和LC-MS峰生成的个体网络进一步扩大,这样三个网络可以在代谢组学层合并。
p-value过滤:(cut-off: 0.2)来排除p值较大的LC-MS峰值所贡献的节点。其网络连接基于其相关代谢物、基因或蛋白质。
如图A,生成的子网络1包含六种类型的节点(物种、SNPs、代谢物、SNPs或代谢物相关的基因/蛋白质),结果显示出glutathione是宿主-微生物相互作用中的重要交互作用点。
由此推测包括大肠杆菌在内的几种微生物会产生glutathione,两个SNPs(rs3197999和rs1428554)通过编码谷胱甘肽过氧化物酶(GPx)的基因GPX1和GPX3与代谢产物相关。
与此呼应的是,在以往的研究中证明大肠杆菌和相关物种在IBD中的过度表达可能是由于它们能更好地产生glutathione以抵抗氧化应激。
图B的3D分层网络提供了一个直观的多组学整合视角,突出显示了连接微生物组和宿主遗传的glutathione的流动路径。
OmicsNet 2.0与其它工具的比较
如下表,比较OmicsNet 2.0与其他基于web的多组学集成工具的关键特性。
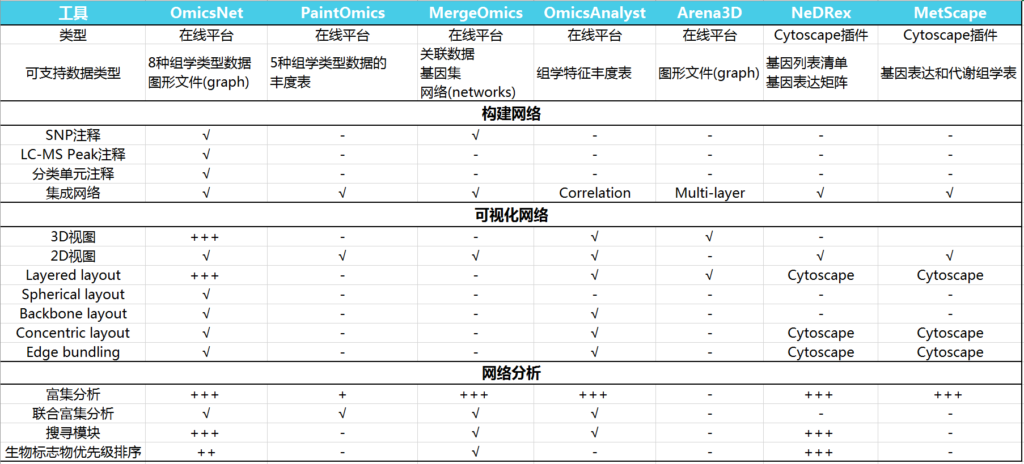
‘√’表示支持,‘-’ 表示缺失,“+”表示定量评估(“+”越多表示更好的支持)
附带各工具的官网及主要特点描述:
PaintOmics
(http://www.paintomics.org)
专注于在视觉上呈现多组学数据的探索分析,包括转录组学,代谢组学,表观基因组学,miRNA和转录因子,将其映射到KEGG通路联合分析。
MergeOmics
(http://mergeomics.research.idre.ucla.edu)
整合了来自单个组学层面的关联研究的汇总统计数据,以及不同功能基因组学数据,以获得机理上的见解,最近还加入了多组学信息的药物重新定位(drug repositioning)。
OmicsAnalyst
(https://www.omicsanalyst.ca)
基于输入的数据,利用多元统计、相关性分析和聚类方法,结合网络、热图和散点图进行多组学分析。
Arena3D
(https://www.arena3d.org)
擅长使用基于3D的分层布局对多层网络进行交互式可视化,适用于多组学网络数据。
NedRex
(https://nedrex.net)
是一个Cytoscape插件,专注于疾病模块识别和药物再利用,使用各种模块识别和优先排序算法。
MetScape
(http://metscape.ncibi.org)
是一个Cytoscape插件,通过构建和分析不同类型的含有酶、代谢物和/或反应的网络,专注于基因表达和代谢组学数据的集成和可视化。
OmicsNet
(https://www.omicsnet.ca)
通过将多个分子互作数据库与强大的2D/3D可视化网络分析相结合,对于多组学集成分析和结果解释有更好的理论基础,对以上工具的不足做了补充。
结 语
OmicsNet 2.0是一个基于网络的多组学分析平台,支持2D和3D网络可视化探索。
在1.0版本中强调基于web的3D网络可视化。
在2.0版本中,进一步改进了其可视化分析系统,添加了一个功能完整的2D网络可视化系统,并支持三种新的组学数据输入(SNPs、微生物分类单元和LC-MS峰),目前的生物信息学工具无法很好地支持这些数据。
除了富集分析、搜寻模块和最短路径分析,用户还可以使用重启随机游走算法(Random Walk with Restart)搜索候选疾病标记物。最后,2.0版本通过发布底层的R代码和共享链接,改进了工具的可再现性和透明度。
使用IBD多组学数据的案例研究表明,OmicsNet 2.0可以揭示与原始出版物和后续出版物以及IBD文献一致的有意义的模式、关系和功能。总之,OmicsNet 2.0解决了对多组学数据进行分析的一些需求。
参考文献:
Zhou G, Pang Z, Lu Y, Ewald J, Xia J. OmicsNet 2.0: a web-based platform for multi-omics integration and network visual analytics. Nucleic Acids Res. 2022 May 26:gkac376. doi: 10.1093/nar/gkac376. Epub ahead of print. PMID: 35639733.

谷禾健康
生物系统——组成
生物系统很复杂,具有许多调节功能,例如DNA,mRNA,蛋白质,代谢物,以及表观遗传功能(例如DNA甲基化和组蛋白翻译后修饰(PTM))。 这些特征中的每一个都可能受到疾病的影响,并引起细胞信号传导级联和表型的改变。 除了宿主对疾病的反应调节机制外,微生物组还可以改变宿主特征的表达,例如它们的基因,蛋白质和/或PTM。
生物系统——疾病
为了深入了解疾病的机制,我们需要研究这些特征及其相互作用。例如,黑色素瘤、肺癌和甲状腺癌等癌症是由BRAF癌基因驱动的。然而,当患者接受抑制BRAF的治疗时,往往会产生耐药性。最近的多组学研究揭示了肿瘤特征的异质性和复杂性,如基因突变、转录组、蛋白质和信号通路。现在人们认识到肿瘤可以绕开治疗而产生耐药性。
生物系统——技术
随着下一代测序和质谱技术的发展,人们越来越需要融合生物特征的能力来研究整个系统。转录组、甲基组、蛋白质组、组蛋白翻译后修饰和微生物组等特征都影响宿主对各种疾病和癌症的反应。由于样品制备步骤、测序所需的材料量和测序深度要求,每个平台都有技术限制。近年来,数据集成方法的发展受到了推动。每种方法都使用诸如概念整合、统计整合、基于模型的整合、网络和路径数据整合等方法来具体整合组学数据的子集。
生物系统——多组学
多组学方法的整合使得对疾病病因学有了更深入的了解,例如:揭示微生物组在减轻或增加疾病风险方面发挥作用的各种方式。双酚A(BPA)是一种大规模生产的化学品,广泛应用于食品包装、塑料和树脂中,双酚A的不完全分解就是一个例子。由于双酚a是一种内分泌干扰物,双酚A已成为日益增长的公共卫生问题。因此,利用微生物手段快速、完全降解双酚A等化合物的研究具有重要意义。
本文讨论每个数据特征的研究设计考虑,基因和蛋白质丰度及其表达率的限制,当前的数据整合方法,以及微生物对基因和蛋白质表达的影响。在开发整合多组学数据的新算法时应考虑的因素。
不同生物基因数量
生物系统是具有多种调控功能的复杂生物。 例如,人类基因组由大约32亿个核苷酸组成,可产生20 000至25 000个蛋白质编码基因,并且通过选择性剪接事件可产生超过100万种蛋白质(下图)。
不同的生物有不同数量的基因和蛋白质。例如,在大肠杆菌、酿酒酵母和智人基因组中分别有大约4300、6000和25 000个基因。这导致大肠杆菌、酿酒酵母和智人的每个细胞中分别有大约2400到7800、15 000和300 000个mRNA分子。线粒体转录物约占多聚腺苷酸化RNA的20%。其他高丰度的转录物包括编码核糖体蛋白质和参与能量代谢的蛋白质的转录物。下图概述了人类DNA、DNA甲基化、组蛋白翻译后修饰、mRNA和蛋白质的复杂性。

Graw et al., 2020 Molecular Omics
染色质结构和基因/蛋白质调控的概述。 DNA通路受DNA甲基化和组蛋白翻译后修饰(PTM)的调控。调节的每一层也可以通过环境和宿主生物中存在的微生物进行修饰。 可以通过使用各种核苷酸和蛋白质/肽测序技术对生物调节的每个水平进行测序。
细胞中蛋白质含量
一个细胞中蛋白质的估计数量约为2.36×106(在大肠杆菌中),约为2.3×109(在晚期智人细胞中)。在一个细胞的全部蛋白质总数中,最丰富的蛋白质可占蛋白质含量的5-10%,由核糖体蛋白、酰基载体蛋白(ACP)(在脂肪酸生物合成中的功能)组成,分子伴侣和折叠催化剂、糖酵解蛋白质(能量和碳代谢的主干)和肌动蛋白等结构蛋白质。
转录因子是一种低丰度的蛋白质,在细菌中每个细胞的拷贝数为1-103,在哺乳动物细胞中为103-106。
最丰富的蛋白质通常在细菌中有数千个拷贝,在哺乳动物细胞中有数百万个拷贝。由转录因子调控的基因数量取决于其浓度。蛋白质含量取决于生长条件和基因诱导。最后,考虑到微生物与宿主细胞数量的比例(取决于宿主细胞类型)和其他因素,这可能会变得更加复杂
mRNA 和蛋白质寿命以及差异
由于仪器检测,动态范围和分子寿命表达的限制,用于各种组学平台的测序技术只能捕获某一时刻某个细胞群体中发生的情况的快照。 例如,mRNA转录本和蛋白质的终生表达差异很大。 在大肠杆菌中,mRNA的中位寿命为5分钟,在发芽酵母中为20分钟,而对于人参则为600分钟。然而,蛋白质的寿命约为1-2天。
转录和翻译的速率因生物体的不同而不同(大肠杆菌:每秒10-100个核苷酸(nt)和10-20个氨基酸(aa)/ s。智人:6-70 nt / s和2 aa per s;分别为转录和翻译速率)。
对于大肠杆菌来说,一个单一的mRNA转录本在被降解之前可以产生10-100个蛋白质。鉴于这一信息,我们可以看到,将我们对组学平台的选择和由此产生的对细胞过程的解释相结合,检测具有更长寿命的蛋白质的机会将增加。
在考虑数据整合研究设计、开发新算法和解释结果时,认识生物体的生物复杂性、分子的动态范围、测序限制以及这些分子的表达寿命非常重要。
近年来,微生物组学在宿主健康中的重要性已得到公认。全生物和全基因组的概念对我们如何看待微生物组有着深远的影响,尤其是在治疗方面。这种微生物-宿主相互作用的密切关系可以更明确地称为“微生物群-营养代谢-宿主表观遗传轴”。微生物与宿主相互作用的紧密关系可以更明确地称为“微生物群-营养代谢-宿主表观遗传轴”。
微生物群及其代谢产物可以通过直接修饰组蛋白,改变DNA甲基化谱图和影响而影响宿主表观遗传。 非编码RNA的性质(上图)。 例如,可以通过改变组蛋白修饰酶的活性和酶底物的水平,通过微生物群来修饰组蛋白。
微生物影响药效
微生物群也可以影响药物的治疗性质。 许多前药,即必须进行代谢转化才能在药理上有用的药物,可能会保持无活性(即不存在介导前药向其活性形式转化的微生物群),或者该药物/前药可能无法生物利用。此外,服用NSAIDs(非甾体类抗炎药)的患者可能会促进抗生素耐药菌的优势,因为24%的非处方非处方NSAIDs被抑制。
这些代谢组学效应引起人们对旨在用于人类和农业系统的治疗药物或其他饮食和治疗方案的潜在副作用的担忧。 例如,抗生素可以消除产生组蛋白脱乙酰基酶(HDAC)抑制剂的微生物。 这些微生物(如果存在)可以增强调节性T(Treg)细胞,从而有助于抗炎过程。
微生物代谢途径的多样性及其对药物药代动力学和药效学的影响可能部分解释了个体和人群之间药物反应的变化。 因此,涉及微生物组的治疗方法可能必须因地制宜。组蛋白可以同时进行变体置换和翻译后修饰(PTM),这些共同构成了“组蛋白密码”。 这些局部排列可以影响染色质结构,从而导致转录活性的激活或抑制。
通过饮食,微生物有能力改变宿主的甲基化和PTM谱,并且还可以通过膳食碳水化合物的发酵影响短链脂肪酸(SCFA)的生成。丁酸盐和乙酸盐等SCFAs可抑制脱乙酰酶水平。这意味着由于乙酰化促进转录活性的增加,染色质结构变得越来越松弛。事实上,已经证明微生物可以以位点特异性和组合方式影响宿主组织乙酰化和甲基化染色质状态,甚至影响宿主发育和代谢表型。
微生物参与干预
未来关注健康医疗策略时,越来越多地考虑对微生物组的发展及其相应的宿主个体发育变化进行建模。考虑到宿主免疫系统不仅必须能够识别“自身”抗原,而且还必须能够识别共生微生物的抗原,这些变化可以通过宿主免疫成熟来证明。
微生物如何影响主要组织相容性复合体(MHC)的表达,或者宿主杂合度如何通过MHC影响微生物群的多样性,这在很大程度上是未知的,也是一个活跃的研究领域。微生物在癌症和免疫治疗中的作用正日益成为治疗策略发展的目标。蛋白质组学与其他组学策略相结合已被用于研究疾病过程。如果我们不考虑微生物群的影响,那么我们可能会错过开发潜在治疗方法的有意义的见解。尤其是那些与代谢紊乱(如肥胖)或代谢物(如胆汁酸)对器官系统的全身影响有关的疾病。
微生物生态学的历史围绕着适当的系统发生标记基因的测序和比对。 WoSes and Fox(1977)首先将16S rRNA基因用作标记基因,是迄今为止最常用的标记基因,其大规模数据库包含从环境和培养来源(例如SILVA,RDP,Greengenes)获得的全长基因分离株 )(表1)。 新的微生物分类数据库,例如基因组分类数据库(GTDB),不仅建立了16S rRNA基因参考数据库,而且还利用系统基因组学信息提供了一个一致的框架,用于确定从元基因组获得的系统发育背景部分或完整基因组 。
大数据集的可用资源列表

选择合适的引物和平台
除了选择标记基因和合适的数据库外,研究人员还可以在测序方法和平台之间进行选择。 由于Illumina和Ion Torrent等短读平台的局限性,研究人员必须在〜1500 bp的16S rRNA基因的可变区之间进行选择。 取决于微生物群落组成,每个可变区提供不同水平的敏感性和特异性。 然后选择在研究中最能区分普通分类群的引物组和扩增子区域的组合。
目前针对扩增子测序可选择的测序平台和方案很多,不同平台的读长和适用的测序区段以及优势各有不同。16s测序主要的测序区段包括v4、v3v4,v1v2,v6,此外还有全长等不同的区段选择,不同可变区或全长由于引物的不同以及不同种属相应区段内的变异多样性差异,对菌属的丰度评估会有一定的差异。
从长度来看,全长16s长度为1.5kb左右,单菌落的16s全长sanger一代测序仍然是菌种鉴定的主要手段,纳米孔和pacbio的三代测序可以高通量的获得全长序列,对于希望更高分辨率的分析菌种的研究有一定优势。三代的测序准确度目前逐渐改进,直接测序准确度可以在90%以上,纠错后可以提高到97~99%以上,已足够提供高精度的分类。三代目前主要问题在于建库成本相对较高,通过使用barcode可以降低部分但仍然偏高,此外普遍测序深度相对于二代测序要低许多。
目前最主要的可变区选择是v4区和v3v4区,v4区长度为256bp左右,加上两侧引物长度为290bp左右,使用双端2x250bp或2x150bp可以测通,此外如454、life、illumina的测序平台读长也可以主要涵盖该区段读长。例如采用illumina Novaseq测序平台对该项目进行双端测序(paired-end),测序得到了fastq格式的原始数据(样本对应一对序列s_1.fastq和s_2.fastq)。再配对拼接成单条序列。其引物通用性相对是所有可变区中最高的,大量的大规模菌群调查研究都采用v4区作为检测区域,包括人体菌群研究如:hmp,肠道菌群如美国肠道计划agp,欧洲的fgfp等,以及全球土壤菌群调查,目前仍然是国际研究中使用最广泛和认可的检测区域。
illumina的miseq提供了长达2x300bp以及hiseq2500和最近的novoseq提供有2x250bp的测序方案,为进一步利用读长,目前有相当一部分研究选择v3v4区,该区段长度在460bp左右,相较于v4度多出了v3区段约100bp左右的片段,在少部分菌属中可以增加一定分辨率。经过对比,v3v4区的检测结果和v4区在绝大部分菌属中的丰度一致,但由于引物不同,在少量菌属中丰度会有不同偏向,v3v4从otu层面上并未发现较v4区有明显增加。引物的选择和提取、储存方法是影响菌群检测丰度构成的主要因素,不同研究之间的比较需要考虑到实验方案的一致,相同的方案可以直接比较。
当前的宏基因组分析技术已使研究人员能够从环境/宿主来源的样品中获得足够的序列覆盖率,从而获得部分和完整的基因组草图。 该覆盖因子高度取决于物种的均匀度和丰富度。 还可以通过拼接组装元基因组组。 但是,由于难以组装和正确分装高度保守的基因(如核糖体亚基基因),因此它们通常必须使用浓缩的通用蛋白将这些基因组置于系统发育背景中。
组合的通用标记基因被用来构建由环境和寄主衍生序列组成的基因组以及来自培养物收集的少数常见微生物基因组的系统发育。微生物基因组测序的热潮使得有必要构建易于使用的软件包以及分析工具,以帮助生物学家学习如何对其全部或部分的元基因组数据进行分析。这类工具的例子如,QIIME 2、metaWRAP、 Sunbeam、SqueezeMeta、metAMOS、 mg RAST、IMG/M、 Anvi’o、MicrobiomeAnalyst、以及biobakery集合中的各种工具(例如MetaPhlan2、PhyloPhlan、HUMAnN、LEfSe)等。
此外,如果深入研究,还需要望整合疾病指标、宿主蛋白质组学和微生物多样性多组学的联合分析。
根据生物学问题的不同,有许多类型的组学技术,针对DNA、总RNA、mRNA、miRNA、DNA甲基化、蛋白质、蛋白质修饰、组蛋白翻译后修饰、宏基因组学、宏蛋白质组学,测序平台经过多年的改进,现在可以在几天内从少量材料中对大型复杂人体样本进行测序(表2)。已经开发了几种工作流程来对整个基因组、整个外显子组(DNA的蛋白质编码部分)和转录组(mRNA)进行排序,并对特定的癌症或免疫相关基因进行排列。此外,还可以利用亚硫酸氢盐全基因组测序或Illumina的甲基化珠芯片阵列分析修饰,如DNA甲基化。 还可以通过牛津纳米孔技术(ONT)MinION平台和PacBio仪器对长读的DNA和RNA进行直接测序来确定此类修饰的检测。
推荐覆盖率和读数

基因组测序
DNA测序技术的错误率和读取长度各不相同。Illumina短读测序(即Hiseq、Miniseq等)通常具有非常低的错误率,约为每碱基0.25%,但对低多样性文库敏感,如16S宏基因组学和靶向基因方法等应用。长读取技术的错误率较高,PacBio为13–15%,Oxford Nanopore instruments为5–20%。Illumina平台的读取长度最大为600个碱基,但长读取技术通常一次读取可达到10–30 kb。最佳读取长度也取决于应用程序。
大多数测序实验可以收集150-300碱基对读取长度的合适信息,但也有例外。对于全基因组测序(WGS),最长的读取可能是最佳的,但是对于长读取技术,错误率随着长度的增加而增加。有许多研究者把“短读”和“长读”结合起来。由于最近长读取排序技术的出现,关于WGS以外应用程序的最佳长读取长度的信息非常缺乏,但Illumina short read sequencing提供了丰富的最佳读取长度建议。
蛋白质测序
在过去5-10年中,质谱仪通过增加测序深度能力也得到了改进。这项技术已经从使用旧的LTQ质谱仪在细胞系实验中对大约3000个蛋白质进行测序,发展到使用新的Orbitrap Lumos和Orbitrap Eclipse质谱仪对8000-10000个蛋白质进行常规测序。大多数蛋白质组学实验都是采用数据相关采集(DDA)模式进行的。在该方法中,选择从液相色谱(LC)柱洗脱的MS1扫描中最丰富的前20个肽在orbitrap中进行裂解,以产生肽序列MS2扫描。样品混合物的复杂性极大地影响了测序深度和将鉴定多少蛋白质。了解样品的蛋白质丰度和组成是至关重要的。如果转录因子是目标分子,那么在质谱分析之前去除高丰度蛋白质的方法可能是必要的。这对于含有大量分子(如白蛋白和血红蛋白)的血清和血浆样品尤其重要。否则,质谱仪将测序数千个白蛋白分子,并错过最有趣的低丰度蛋白质。
最新的质谱技术利用数据独立采集(DIA)来对MS1扫描中所有肽从LC色谱柱洗脱时的序列进行测序,这与仅对最丰富的峰进行测序的DDA方法相反。
对于复杂的混合物,例如上面的血清示例,DIA方法优于DDA。 这种方法有助于克服受高丰度蛋白质高度影响的复杂混合物。
除了对宿主基因和/或蛋白质进行鸟枪法测序外,我们还可以对微生物组利用鸟枪法测序。
当测序深度很浅时,弹枪宏基因组学/元代谢组学只能采样优势菌群。shot弹枪对微生物组测序的主要挑战是由于采样不足而难以组装基因组片段,将肽组装在一起以进行可靠的蛋白质和生物分类鉴定也同样困难。
尽管存在这些潜在问题,但从各种人体部位和疾病(如唾液、肠道/粪便、颈阴道疾病或慢性肾脏疾病)中对微生物蛋白质组进行深度取样是可能的。然而,每个研究必须考虑的研究/取样设计和分析方法可能有很大差异。
从差速离心到双过滤差速分离,几种样品制备方法已被证明能富集微生物生物量。这些方法通常遵循各种优化的微生物裂解方案,通常涉及机械破坏(如打珠、超声波),辅以酶(如胰蛋白酶)和洗涤剂。在成功溶解后,同样重要的是去除残留的酶、洗涤剂和盐。
元蛋白质组学实验的另一个复杂性是由于同一生物体内的蛋白质具有共享的肽序列这一事实。 为了对蛋白质鉴定有信心,应以高可信度鉴定蛋白质的独特肽段匹配。 当将肽序列映射到数百个具有保守蛋白序列的不同物种时,这变得更加复杂。 质谱法不对蛋白质进行测序,而是测量肽的电荷,并依靠与蛋白质序列数据库匹配的质谱进行蛋白质鉴定。
精心挑选的数据库对于正确分析从这些各种测序平台生成的核苷酸和蛋白质测序数据至关重要。 使读数与参考基因组比对的能力仅与参考基因组中存在的序列和注释信息一样好。 有几种资源可以不断地整理和更新核苷酸序列信息和注释,包括加利福尼亚大学圣克鲁斯分校(UCSC)基因组学研究所基因组,美国国家生物技术信息中心(NCBI)GenBank和RefSeq,DNA元素百科全书(ENCODE)和Ensembl 仅举几例。 通用蛋白质资源(UniProt)包含Swiss-Prot(手动注释和审阅)和TrEMBL(自动注释且未审阅)数据库,以获取蛋白质序列信息。
已经开发了几种数据集成方法来集成某些类型的组学数据。 另外,已经创建了大数据存储库来存储来自各种疾病的测序实验的数据。 这些资源提供了有价值的构建基块和大量生物样本,可用于推动数据集成方法的发展。 当前,数据集成工具实现了多种方法,但通常分为两类:多阶段分析和元维度分析。
多阶段集成模型仅使用数据的两个数字或分类特征构建。 例如,将来自RNA-seq实验的基因计数与来自质谱运行的蛋白质信息相结合。元维度分析试图通过级联或转换将所有感兴趣的数据类型合并到可以同时分析的同时矩阵或“元数据”集中。
后一种方法具有更大的统计能力,但在尝试合并来自不同类型数据集的数据时可能会具有挑战性。 但是,研究人员如何确定最合适的工具或方法?
如上所述,生物学问题是选择的分析方法类型的驱动力,诸如采样,平台类型和数据质量等因素很重要。 样品如何收集和准备?
如果测序深度或质量较低,是否可以有效分析数据? 数据类型兼容吗?
归一化和滤波后损失了多少信号?
这些都是在选择适当工具之前应考虑的所有问题。
不幸的是,数据集成和分析非常复杂,并且对于具有有限生物信息学背景的研究人员而言,目前还没有许多用户友好的工具。 许多工具使用统计语言R,除了强大的生物统计知识外,它还需要专业编程知识。 例如,将蛋白质组学,转录组学和途径分析结合到两个数据集上的R包积分学使用了相关分析和偏最小二乘回归。R包mixOmics使用多元分析进行数据探索,降维和可视化。 通过途径分析,iClusterplus和LRACluster进行的miRNA和基因表达使用聚类来整合甲基化和基因表达数据。
多组学数据集成工具



对于多状态和多维方法,都使用了许多不同的算法,但最常见的算法是聚类,网络分析,数据约简(PCA)和贝叶斯分析。Ray等2014年使用贝叶斯分析,使用从癌症基因组图谱项目收集的数据分析卵巢癌中的基因表达和甲基化数据,并检测到一个基因SPON1,该基因似乎受其CpG位点的甲基化调控。当缺乏生化相互作用的先验知识时,基于相关性的分析是有用的。无论采用何种方法,适当的规范化和数据过滤是非常重要的,因为数据来自多个来源。
还有一些基于网络的工具,如Paintomics,试图使数据分析更容易,但对于缺乏经验的用户来说仍然很困难,研究人员必须对他们的数据有很好的工作知识。此外,还有一些数据库常用于综合组学分析,如癌症细胞系百科全书(CCLE)、癌症基因组图谱计划(TCGA)、与基因组学驱动治疗相关的肿瘤改变(TARGET)和组学发现指数(OmicsDI)。CCLE和TCGA已经描述了数以千计的癌症数据集,可以用于数据挖掘和可视化。TARGET利用临床信息并在其网站上提供分析工具的资源。omicdi提供了一个平台,用于搜索各种生物的公共和受保护数据。
对于任何高质量的研究,进行多组学研究应该首先确定研究的范围和限制。仔细的计划和执行将提高研究的稳健性和可重复性,在多组学研究中尤其重要,因为它们涉及大量的比较、定制的统计分析、大量的财力,时间和精力。一旦研究假设被明确定义,选择一个合适的研究设计,最好地解决研究假设。因此,有几个问题需要评估,
例如:是否有一个或多个干预组与对照组(或其自身)进行比较,或者是否在干预前后对同一样本的效果进行评估?
干预效应是在一段时间内产生的,还是在几个不同的时间点测量样本?
生物样本是否会被单独收集或分析?它的科学依据是什么?
哪些类型的组学平台将提供最有价值的以及如何整合多组学数据?
来自同一生物来源的样本是否可用于所有感兴趣的多组学平台?
理想情况下,所有omic平台的样本将从同一来源收集。
然而,由于样品的特殊限制或材料的可及性和数量,这并不总是可能的,从福尔马林固定石蜡包埋(FFPE)组织生成多组学数据对于某些组学平台可能是不可能的。虽然在选择实验设计时有许多问题需要考虑,但选择研究设计的决定因素通常是其可行性和经费限制。
样品和数据的收集应以数据分析为指导,以减少混淆和技术因素,例如批量效应。这些效应可以在样品收集,制备和存储的步骤中引入。
由于与多组学研究相关的数据的复杂性和大量数据,因此针对特定的研究项目量身定制统计分析至关重要。已提出了多种集成多组学数据的方法,并将其归类为受监督的, 半监督或非监督; 以及基于概念,统计,相关性,网络和模型的集成。
一项研究的统计能力取决于几个因素(下图),其中一些因素可以控制,而另一些因素由于研究及其设计而固定。首先,选择了用于分析的统计方法。虽然有些测试比其他测试更强大,但重要的是验证和满足他们的假设。
影响研究统计能力的另一个因素是单个组学平台测量的变量数量,通常由组学平台决定。例如,基因组学通常测量数百万个变体,转录组学量化了成千上万个分子,和蛋白质组学和代谢组学分析了数千个分子。此外,统计效力受表型或处理效应的大小和差异程度(效应大小)的影响。效果有多明显?组间的信号差异有多大?有多少被测变量受到影响?关于效应大小的信息可以从以前的文献或专家知识中获得,但通常是未知的。
这种情况下,初步研究可以帮助估计效应大小,但由于不稳定,这些估计需要谨慎处理。另一个效力影响因素是测量值的均匀性,描述了样品的自然方差、测量仪器的精度和检测限。随着方差的增大,统计效力将减小。样本的方差可能是多方面的结果,例如样本群体的选择、组织类型的选择或混杂因素。
除了样本方差膨胀外,混杂因素也会在数据中引入偏差,因此,收集样本元数据以减轻某些混淆的影响是很重要的。由于影响研究统计能力的大多数因素是固定的或由研究设计决定的,因此最常用于调整研究统计能力的因素是样本量。

多组学研究中影响统计功效的因素
Graw et al., 2020 Molecular Omics
研究的首要考虑正在调查的疾病或研究问题的背景,以及整合在一起时,什么类型的数据将提供有价值的见解。根据生物学问题、材料类型(新鲜组织、FFPE组织、血清/血浆和细胞系)、DNA/RNA/蛋白质的数量、生物复制的数量以及研究中混杂效应的数量,这些因素将决定数据采集所需的最佳样品制备和测序方法。
样品制备方法,包括每个样品制备的日期、提取的DNA、RNA和/或蛋白质的类型、基因组学的文库生成、质谱分析的蛋白质消化和肽标记方法以及测序平台/仪器,都是研究设计和最终结果解释的关键因素结果。
如果一个样本是在不同的日期制备的,而不是其他生物复制品,这将引入方差和/或偏差,并降低分析的统计能力。如果蛋白质组样品使用多个TMT-10plex批次进行复合,这将在整个测序过程中引入批次效应。这些因素应在样品制备前进行讨论。
同样重要的是要知道什么样的调控特征被捕获用于测序和整合。例如,如果在进行质谱分析之前在样品制备过程中膜蛋白没有溶解,那么膜结合蛋白就不能与基因表达数据整合。质谱数据的一个警告是,缺失值并不一定意味着蛋白质没有表达,只是蛋白质低于质谱仪的检测限。生物学问题应该成为多组学数据整合方法的驱动力。
在大多数情况下,当前的工具利用聚类、网络、数据简化和贝叶斯分析。随着数据获取量的不断增加,产生了大量的数据集,使得机器学习对于有效的分析和数据挖掘变得越来越必要。有必要使用易于获取和记录良好的方法、工具和算法。
机器学习在允许科学家集成多组学数据集方面发挥了越来越重要的作用。通过利用机器在大量生物数据中比较和识别模式的能力,可以用更加准确和有效的方法来阐明复杂的细胞机制,在某些情况下还可以预测临床结果。这是通过计算机独特的能力来实现的,它可以同时观察多个层次的组学数据,从而提供一个更全面的系统视图。
尽管多组学数据集可以为个体提供更深入的理解,但这并非没有成本。组学研究通常依赖于大量的比较、正确的数据类型、适当的统计分析以及大量的时间、技术人员和金钱投入。在构建一个实验时,人们必须清楚什么类型的组学数据可以而且应该被整合以获得对所研究系统的最大理解。
高通量的组学平台并不总是回答研究问题所必需的。传统技术:如酶联免疫吸附试验(ELISA)、免疫组织化学(IHC)和定量聚合酶链反应(qPCR),也是验证特定生物学机制所必需的。事实上,为了验证从组学数据中鉴定出的重要分子是一个真正的阳性结果,通常需要这些技术来验证一个更大的组学研究的结果。
但是每种方法都受到其统计能力、样本量、技术变量、批次效应、测序深度、样本制备和许多其他因素的限制。在设计、进行和分析研究以及解释研究结果时,必须牢记这些因素。因此,如果允许,建议研究设计一开始就让生物统计学家/生物信息学家参与进来。
参考文献:
Graw S, Chappell K, Washam CL, Gies A, Bird J, Robeson MS 2nd, Byrum SD. Multi-omics data integration considerations and study design for biological systems and disease. Mol Omics. 2020 Dec 21. doi: 10.1039/d0mo00041h. Epub ahead of print. PMID: 33347526.
A. Zaman , W. Wu and T. G. Bivona , Targeting Oncogenic BRAF: Past, Present, and Future, Cancers, 2019, 11 , 1197
A. Alvarez-Arenas et al., Interplay of Darwinian Selection, Lamarckian Induction and Microvesicle Transfer on Drug Resistance in Cancer, Sci. Rep., 2019, 9 , 9332 .
K. Yu et al., An integrated meta-omics approach reveals substrates involved in synergistic interactions in a bisphenol A (BPA)-degrading microbial community, Microbiome, 2019, 7 , 16.
G. D. Poore et al., Microbiome analyses of blood and tissues suggest cancer diagnostic approach, Nature, 2020, 579 , 567 —574 .
A. Gonzalez et al., Characterizing microbial communities through space and time, Curr. Opin. Biotechnol., 2012, 23 , 431 —436 Search PubMed .
D. Gurwitz The Gut Microbiome: Insights for Personalized Medicine, Drug Dev. Res., 2013, 74 , 341 —343 .
N. Issa Isaac et al., Metaproteomics of the human gut microbiota: Challenges and contributions to other OMICS, Clin. Mass Spectrom., 2019, 14 , 18 —30