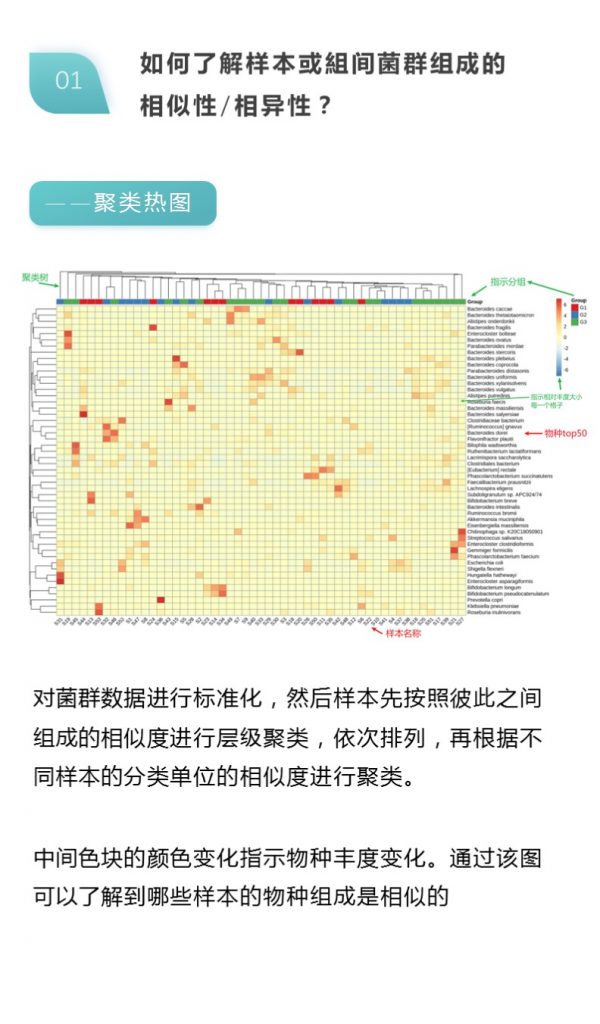
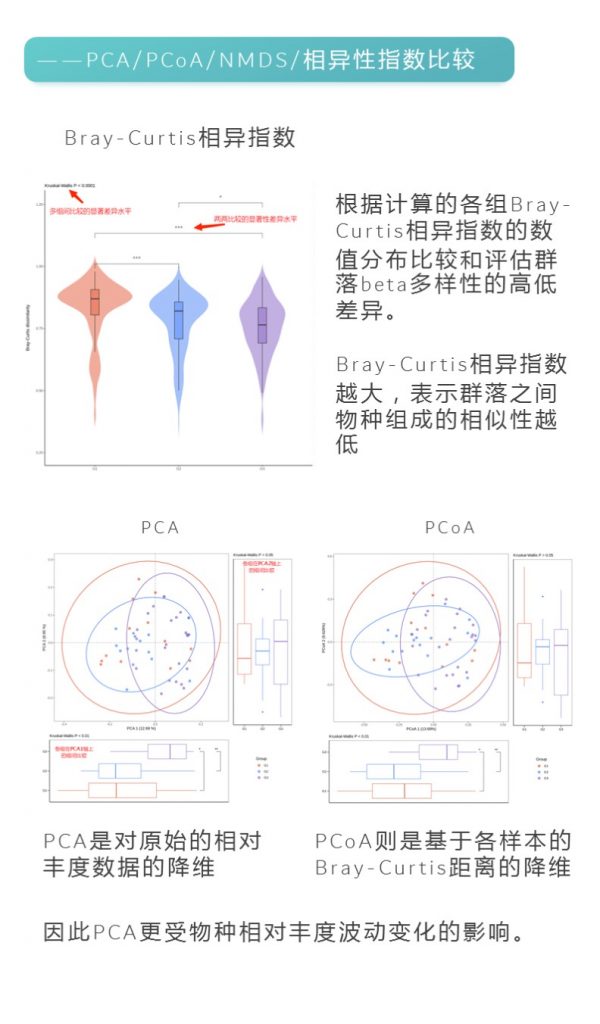
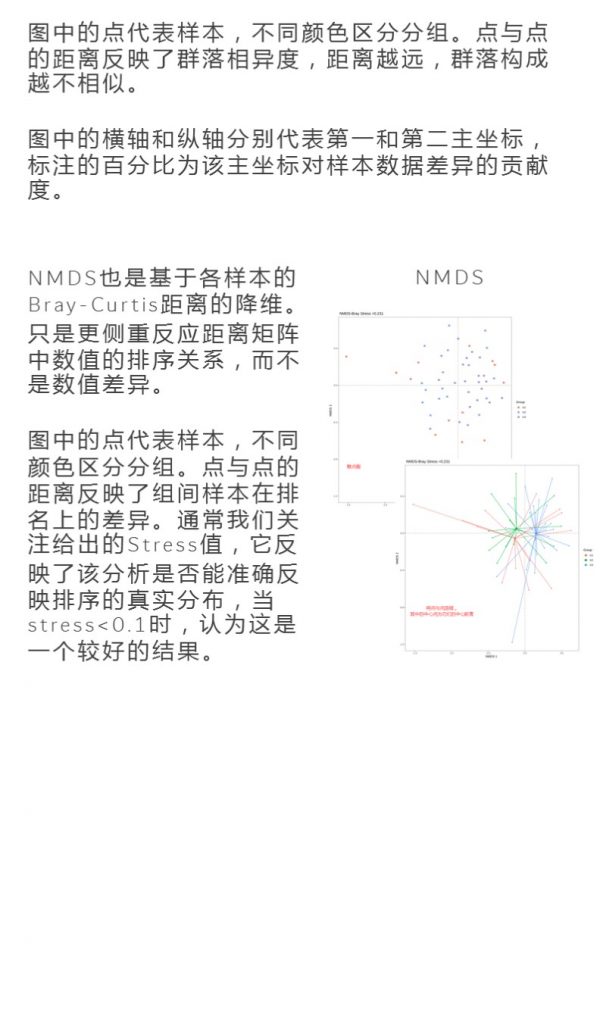
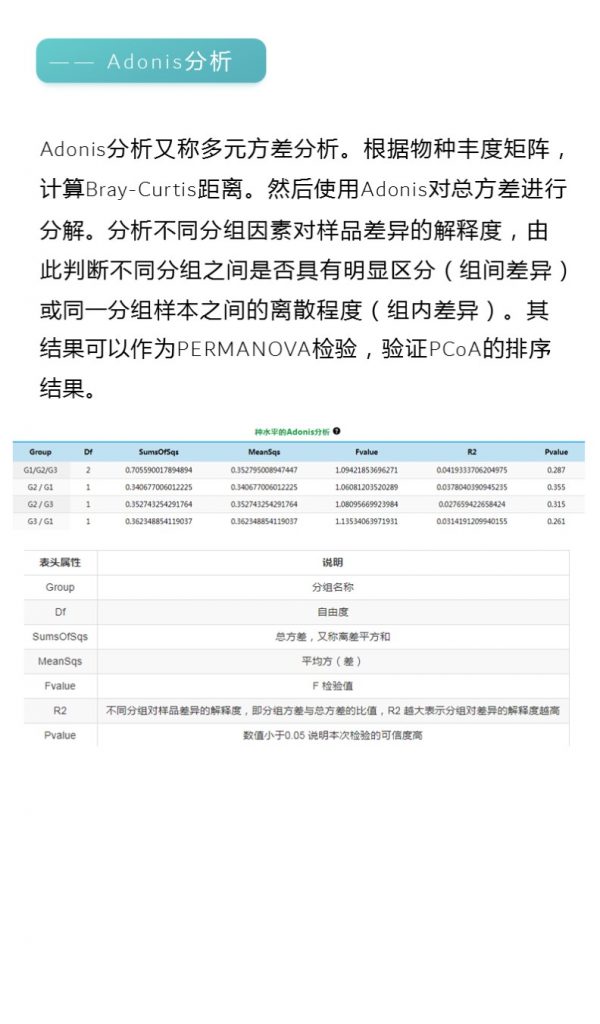
-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康

谷禾最新宏基因组精准检测报告
谷禾利用16srRNA高通量测序技术,在肠道菌群检测方面已经深耕十多年,积累了丰富的经验和海量数据。
16s rRNA测序技术是一种基于细菌16s rRNA基因的微生物鉴定方法,可以快速、准确地分析肠道菌群的组成和多样性,是目前肠道菌群研究广泛使用的一种方法。
在此基础上,谷禾最新又推出肠道微生态宏基因组精准检测报告。

什么是宏基因组?
宏基因组测序也就是shotgun测序,以环境中所有微生物基因组为研究对象,通过对环境样品中的全基因组DNA进行高通量测序,获得单个样品的饱和数据量,基于denovo组装进行微生物群结构多样性,深度全面的了解微生物群体的构成,甚至获得单个菌株的完整基因组。
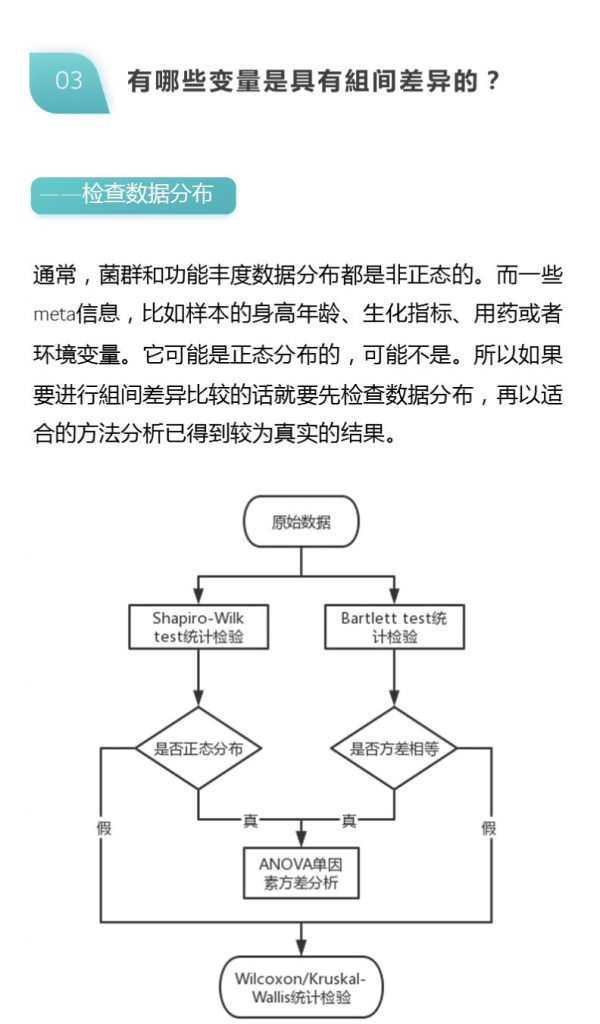
宏基因组检测的优缺点
优点:
宏基因组可以更加精细化开展其基因构成、分布,次生代谢合成,抗生素耐药基因及其演化,微生物群体基因组成及功能等分析。
<来源:谷禾宏基因组精准检测报告>
缺点:
总的来说,宏基因组测序仍存在技术瓶颈和生物学解释的局限性。然而,针对某些特殊情况研究需要,宏基因组测序也是一种有用的微生物组学研究工具。
特殊应用场景
对于一些复杂的多菌种感染,宏基因组能够更精细化鉴定感染菌群的构成,为临床辅助诊疗提供依据。
与传统方法相比,宏基因组学诊断真菌感染的敏感性和特异性更高,适用于一些真菌感染疾病。也可能鉴定出可疑的新病原体,为后续的病原学研究、药物和疫苗开发奠定基础。

<来源:谷禾宏基因组精准检测报告>
谷禾数据库构建
肠道菌群在人体健康和疾病中扮演着重要角色,因此构建一个完善的肠道菌群宏基因组数据库对于研究人体菌群的组成和功能具有重要意义。
宏基因组数据库的物种涵盖范围和菌株构成,直接影响着宏基因组物种鉴定分类的准确性和分类精度。
在选择合适的算法方面也是至关重要的,谷禾整合了最新的NCBI refseq数据库中包含所有细菌、病毒、真菌和寄生虫等微生物物种信息。

<来源:谷禾宏基因组精准检测报告>
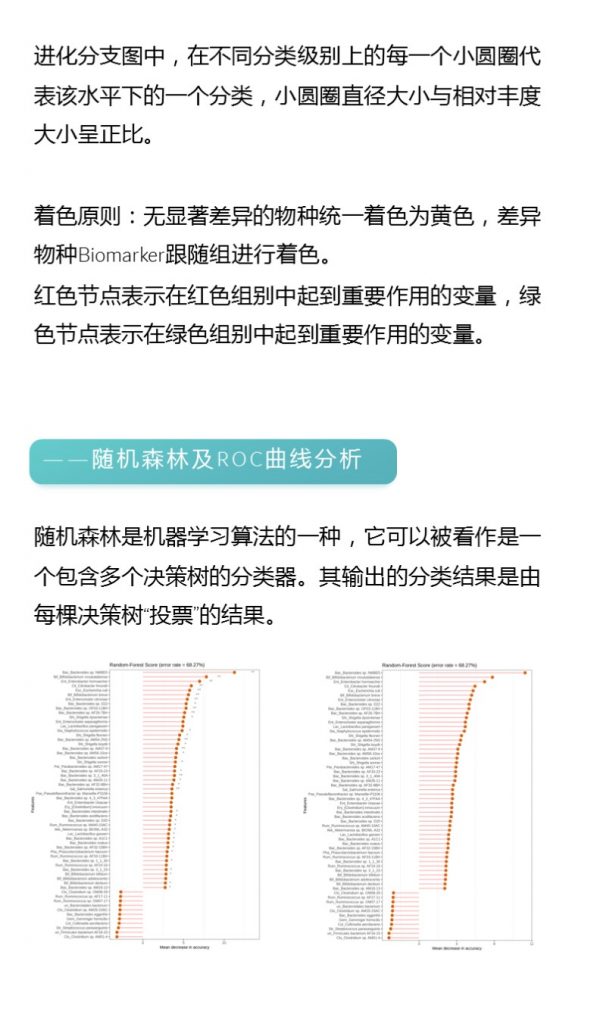
接下来,我们结合谷禾宏基因组精准检测报告中的一些重要模块,包括不同微生物的相对丰度、抗生素耐药、毒力基因等重要信息,更全面的了解宏基因组精准检测报告,包括其在个性化的健康管理和治疗方案制定方面的潜力。
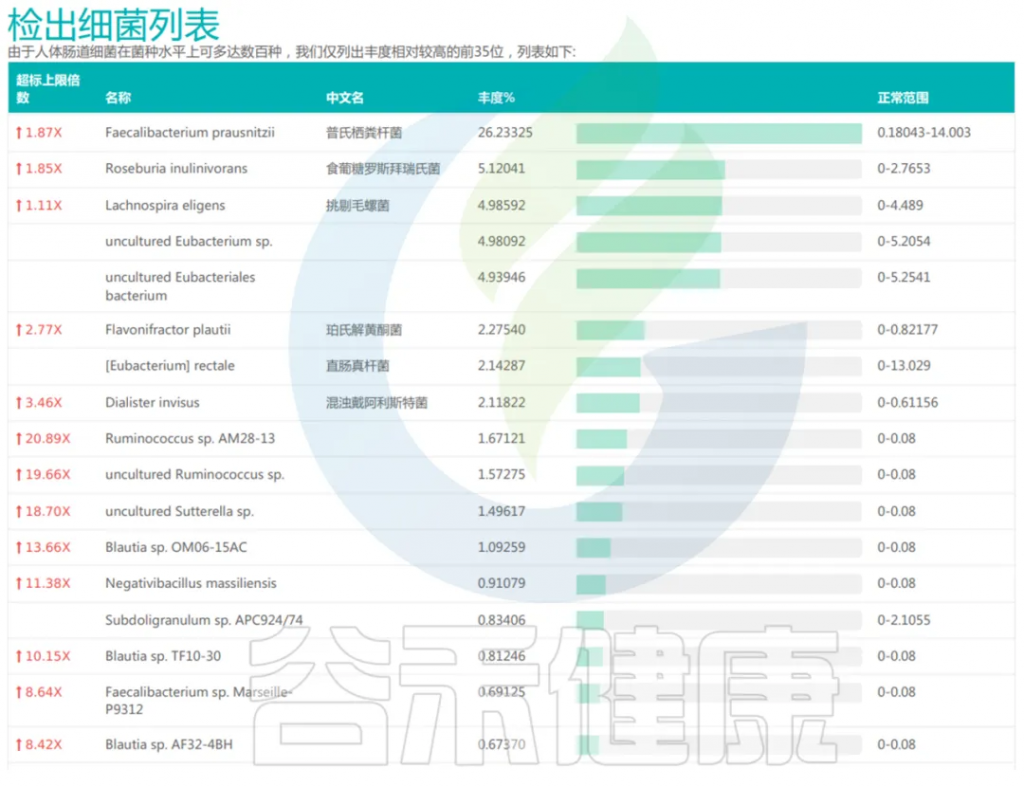
核心菌属
我们将在90%人群检出,人群平均丰度1%以上的菌属为核心菌属,属于人体肠道菌群中最常见和主要的菌属。核心菌属及有益菌累加占总肠道菌群比例低于60%的可能出现肠道菌群紊乱。

<来源:谷禾宏基因组精准检测报告>
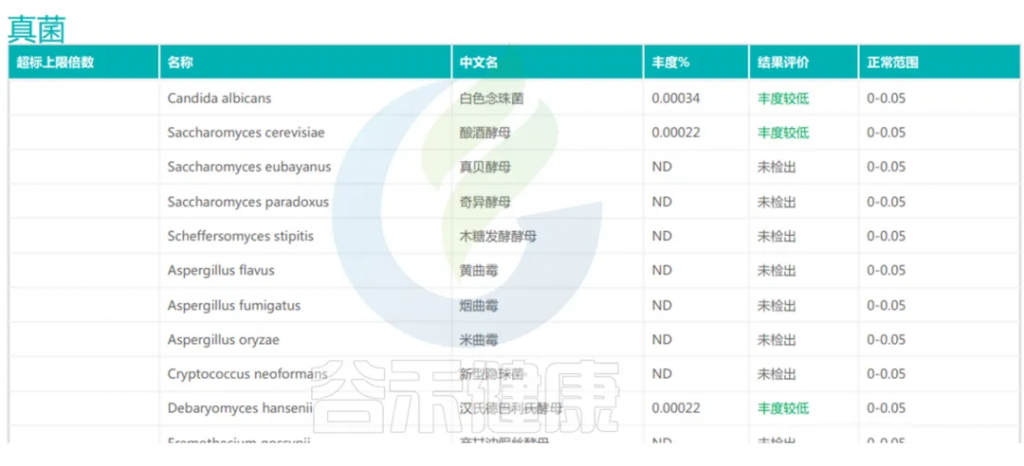
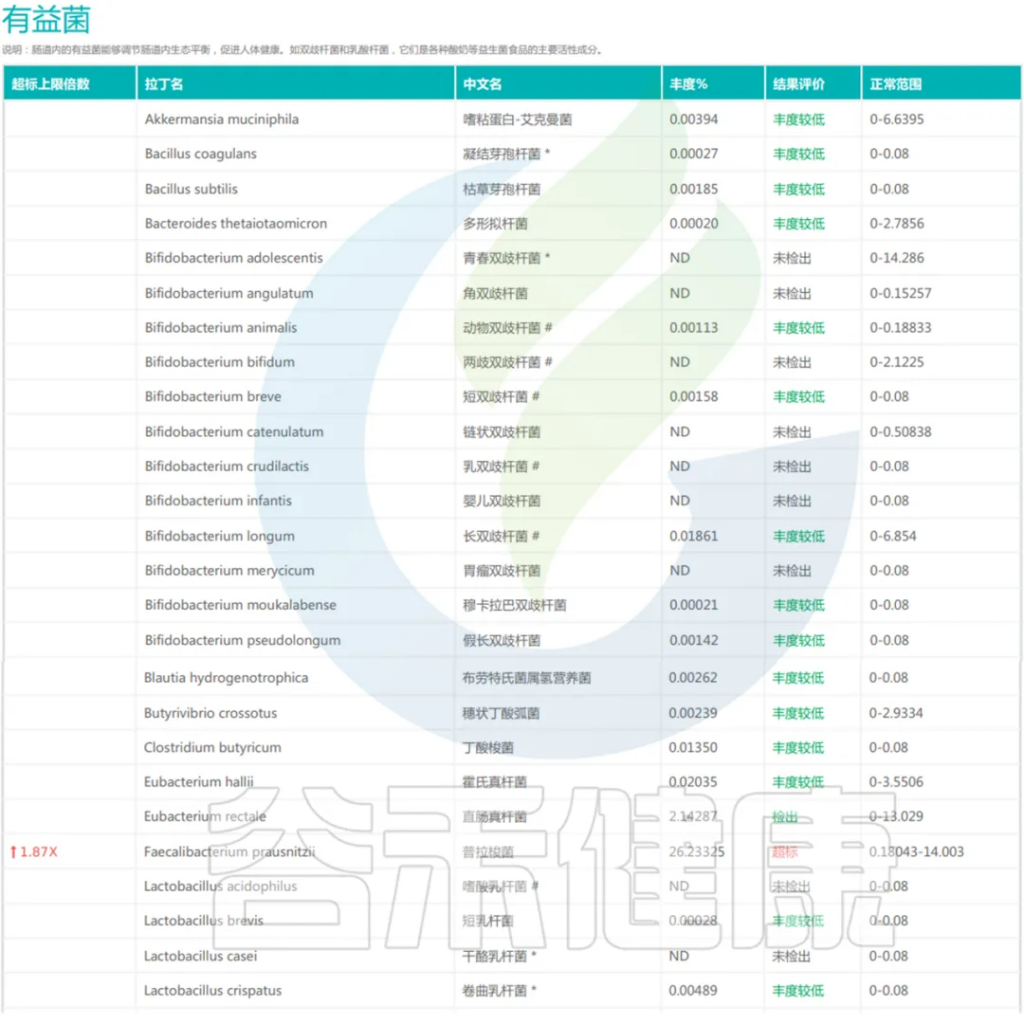
有益菌
肠道内的有益菌能够调节肠道内生态平衡,促进人体健康,如双歧杆菌和乳酸杆菌,保持肠道菌群的平衡对于全面提升整体健康水平至关重要。
对于肠道菌群多样性降低、有益菌减少的肠道疾病患者,可根据其菌群特征,定制个性化的益生菌组合,精准调控肠道菌群。
<来源:谷禾宏基因组精准检测报告>
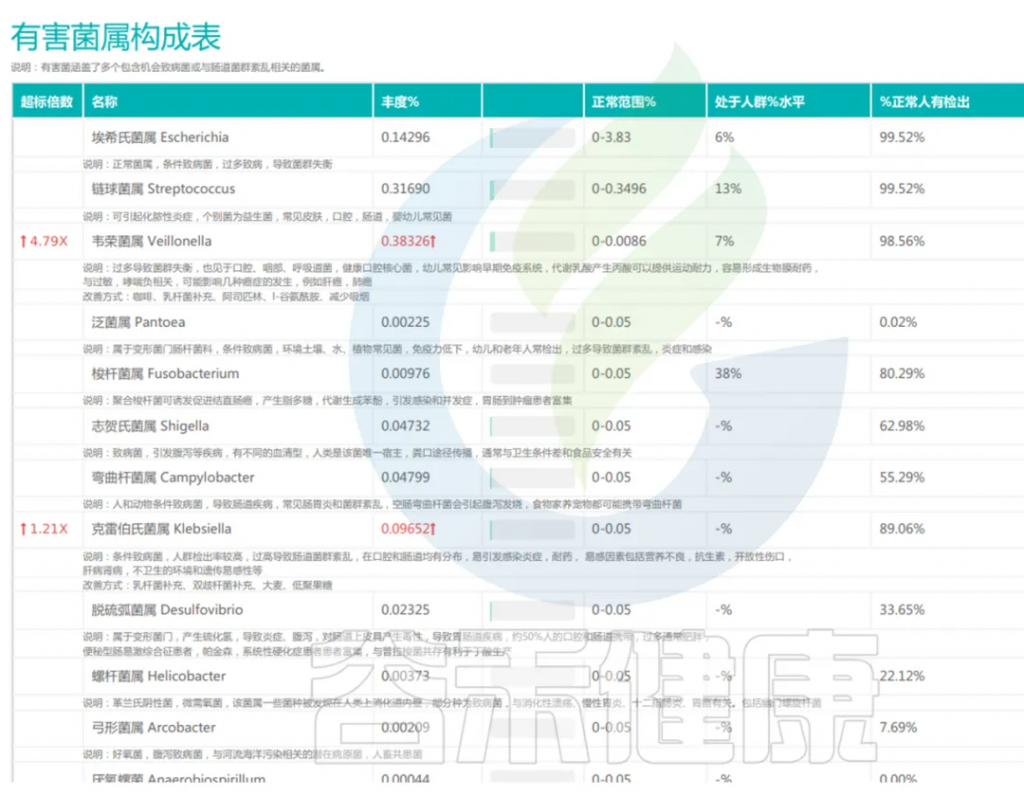
有害菌
了解与疾病相关的有害菌,为辅助诊断和治疗提供依据。例如,对于肠道菌群失衡、有害菌明显增多的患者,可根据其菌群特征,结合其已有的症状,采取个性化的饮食、益生菌、靶向抗菌等综合干预措施,精准调控干预,恢复菌群平衡。
<来源:谷禾宏基因组精准检测报告>
机会致病菌
机会致病菌在肠道内广泛分布,是正常肠道菌群的一部分,但当这类菌过多或免疫力下降时有可能会引发疾病,如大肠埃希氏菌。
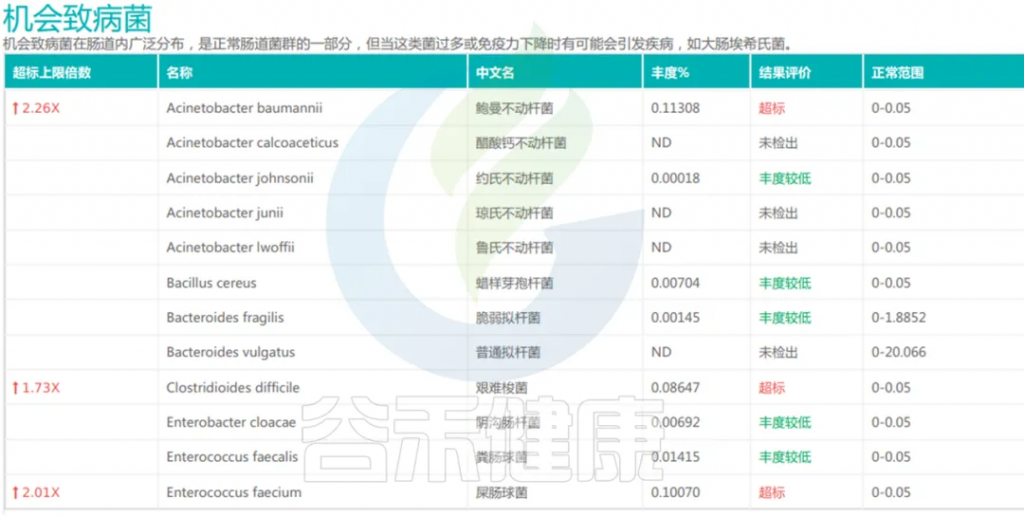
<来源:谷禾宏基因组精准检测报告>
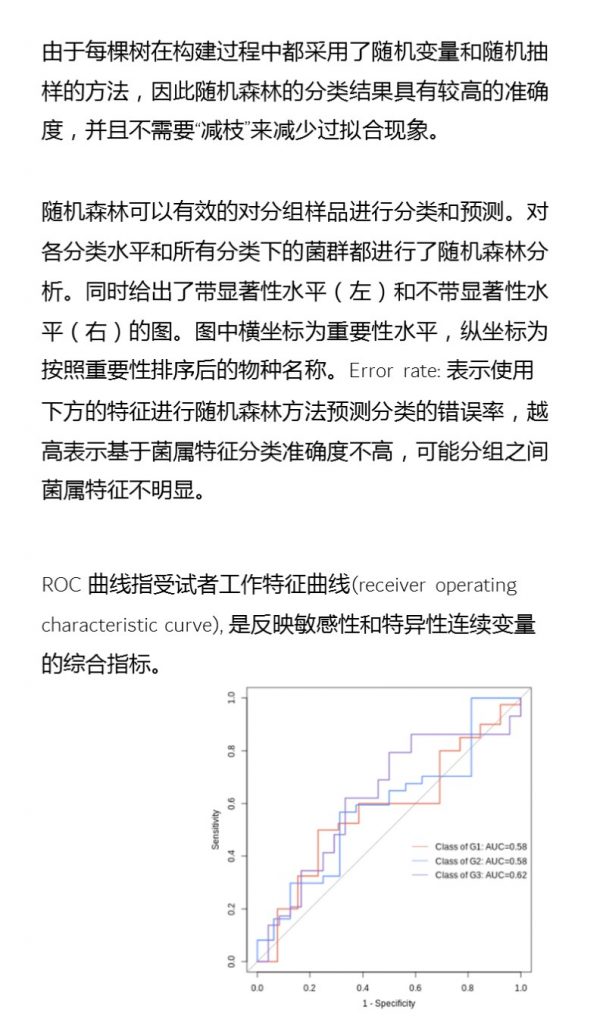
抗生素耐药
宏基因组对抗生素耐药研究也有意义,可以加深对耐药机制的认识,为临床上抗生素干预的选择提供帮助。
分析耐药基因
宏基因组测序可获得细菌群体的全部基因组信息,通过序列比对和功能注释,可鉴定出各种已知和新型耐药基因,全面评估耐药基因的种类和数量。例如,在人体肠道宏基因组中发现了大量β-内酰胺酶等耐药基因。

<来源:谷禾宏基因组精准检测报告>
追踪耐药基因的传播途径
通过比较不同环境(如土壤、水体、动物和人体)中耐药基因的分布情况,可推测耐药基因的来源和传播途径。
辅助指导耐药风险评估和防控策略
宏基因组学评估环境和宿主中耐药基因的分布特点,识别高风险区域和人群,为制定针对性的监测和干预措施提供依据。
毒力基因
宏基因组检测技术在病原微生物毒力基因研究中具有独特优势,可在基因组水平系统分析其毒力基因组成及调控网络,加深对致病机理的理解。

<来源:谷禾宏基因组精准检测报告>
以上是谷禾宏基因组精准检测报告的一些节选,其全面、精准、个性化分析肠道菌群的组成和功能,可帮助评估菌群失衡的风险和预后,为个性化诊疗和健康管理提供科学依据。

谷禾健康

许多流行病的爆发都是病毒引起的,面对新的传染性基因组出现的最佳策略是及时识别,以便于在感染开始时立即实施相应措施。
目前可用的诊断测试仅限于检测新的病理因子。适用于同时检测存在的任何病原体的高通量方法可能比使用基于当前方法的大量单独测试更有优势。
宏基因组学测序、全基因组测序和靶向深度测序是目前用于病毒遗传鉴定和表征的最佳工具。通过使用这些技术,可以正确的对病毒进行分类,确定其变异性,识别与毒性相关的病毒遗传标记,并在现有知识的基础上考虑抗原性和对抗病毒药物的易感性。
尽管宏基因组学领域取得了巨大进步,但对于具体数据分析任务应使用各种方法中的哪一种,仍缺乏共识。
本文重点描述了宏基因组生物信息数据处理所需要的工具,以便于改善使用宏基因组学识别动物来源样本中新出现、再出现和未知的新病毒。
什么是宏基因组学?
宏基因组学是下一代测序的一个领域,可以识别微生物群落,以及基因检测、识别和表征致病因子。它已被证明是病毒遗传特征的关键因素,并导致了使用传统培养技术无法完成的发现。
目前的分子检测使用特定的引物或探针针对有限数量的病原体,而宏基因组学可以接近样本中存在的所有 DNA 和 RNA 分子,从而能够分析相应的宿主基因组及其微生物集合。
在宏基因组组装中鉴定病毒有五个主要步骤:
宏基因组分箱是在物种注释之前可选的附加步骤。分箱的目的是根据根据序列的起源对其进行聚类。
根据这些步骤,列举出以下目前使用较多的主流工具。
宏基因组学的第一步将是执行序列QC,因为从分析中消除技术错误是必不可少的。
此步骤的主要目的是识别不需要的接头序列、过短的序列、低质量的序列或核苷酸以及其他可能存在的数据。根据数据类型,在这一步中可以使用以下几种工具:

对于短读,可以使用FastQC执行质检 (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/) ,它可以检查序列的质量并生成总结报告。
其他QC程序也可以提供相同类型的报告,如MultiQC,它具有与FastQC相同的功能,但有一个主要区别,它可以同时合并多个fastq的QC报告,生成一个总的报告。
对于长读,可以使用longQC或MinionQC来检查序列质量,这两个工具已经应用于从纳米孔的MinION或其他长读取测序仪中获得的数据。
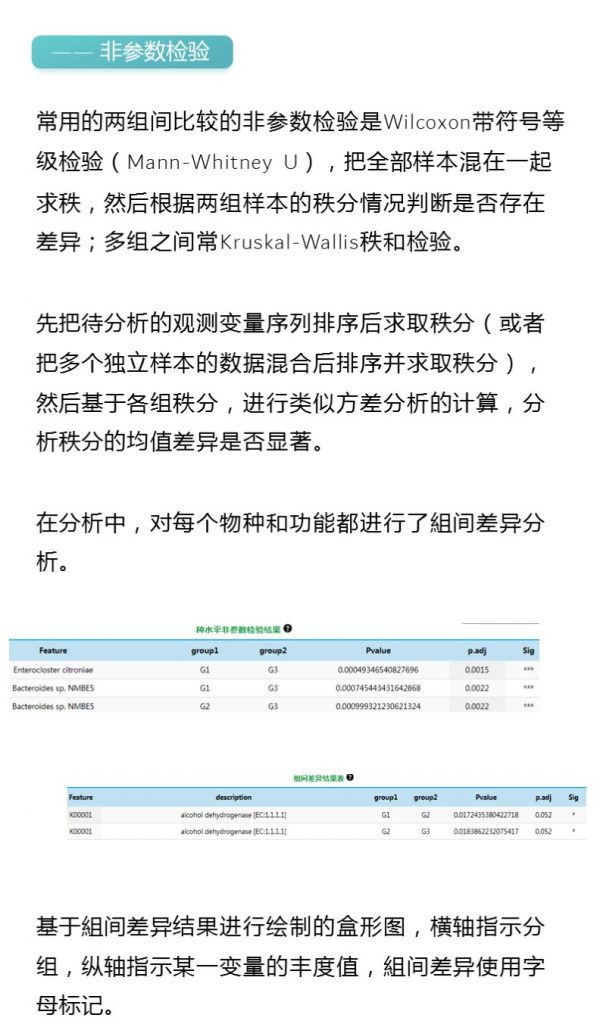
—— 低质量序列修剪工具
序列质检后,就需要修剪工具,删除低质量序列和接头序列。可供使用的工具如下图。

对于短读,常用的是Trimmomatic,其次是Cutadapt和Fastp。
对于长读,NanoPack可用于处理长读数据并可视化QC结果。与Nanopack功能相同的是SequelTools。
——删除测序数据中非靶向或污染序列的工具
删除不感兴趣的序列,这些序列可以从各种来源获得。在对病毒序列的分析中,必须删除宿主序列和被污染序列,它可以减少假阳性,并可以防止嵌合病毒-宿主序列的组装。
如下图,通常使用序列比对的工具:

对于短读,可以使用BWA、bowtie2和BBMap等。
其它工具如FastQ-Screen,可以以fastq格式比对自定义参考序列。
(https://www.bioinformatics.babraham.ac.uk/projects/fastq_screen/)
对于长读,可以使用BWA和BBMap,也可以选择特定的minimap2。
也有专门用于识别和修剪特定微生物类群序列的工具,这类工具通常已包含参考基因组序列,一旦比对上,将通过内置的过滤程序丢弃掉。比如VirusHunter(https://bio.tools/virushunter),用于识别NGS数据中的病毒序列。
某些情况下,可能需要从宏基因组数据中删除非靶向分类群的其他RNA序列类型,如核糖体(rRNA),线粒体(mtRNA),或mRNA类型。这时可以用RiboDetector (https://github.com/hzi-bifo/RiboDetector),因为它专门识别rRNA,从而可以过滤掉rRNA以改进后续分析。
另一种方法是在组装前对序列进行物种注释。使用这种策略,可以过滤掉病毒以外的序列,保留病毒序列以供进一步分析。可以使用kraken2和kaiju。
为了更好的进行物种注释和识别存在的病毒,对序列进行组装,生成contigs,以提供更长的连续序列。宏基因组学中使用的组装类型主要为de novo,即从头基因组组装。
可使用的工具如下图:
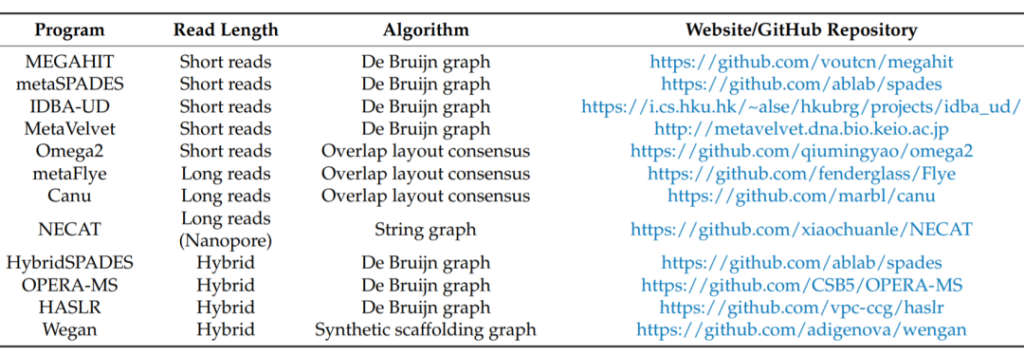
对于短读,推荐MEGAHIT,这是一个针对宏基因组优化的生物信息学组装工具,或者metaSPADES和IDBA-UD,它们也针对宏基因组进行了优化。
除了de novo,还有一种基于参考的组装,也可以用于宏基因组学。只是,并不是在所有情况下都可以获得合适的参考基因组,而且这种方法不能识别新的病毒或以前没有测序的病毒。
对于长读,推荐metaFlye、Canu和NECAT,这些工具可以用于各种技术下产生的数据格式,从纳米孔测序到PacBio,甚至在高保真序列。
对于混合组装,也就是将短读和长读的两个特性结合起来的组装,推荐OPERA-MS和HybridSPADES工具,它们都是用De Bruijn图算法实现的。
宏基因组组装完成,就应该确定组装的质量。用于此目的的工具可以分为两大类:
一类是需要参考基因组的工具,例如MetaQUAST,它使用参考来计算组装的统计信息。一般而言,在宏基因组学研究中,可能很难使用参考基因组,因为通常没有可用的参考基因组或参考基因组的质量很差。
不需要参考基因组的方法,例如DeepMAsED,它使用机器学习来识别错误装配,或者REAPR,是一种使用映射的配对端读长来评估基因组组装准确性的工具。常用的还有BUSCO和CheckM。
最后,VALET(https://github.com/marbl/VALET)可以用于检测宏基因组数据中的误组装,因为它可以根据覆盖范围对contigs进行分类,并避免由于覆盖深度不均匀而导致的假阳性和假阴性。

在宏基因组分析中识别病毒的一个重要步骤是进行物种注释。实现这一步骤的主要方法有两种:
两种方法各有优缺点:
在使用contig(即使用组装序列)进行的物种注释中,分类的对象是较长的序列,它存在一些contigs可能是嵌合的风险。
而直接对reads进行物种注释的统计学意义较小,虽然分析了大量的序列,但序列较短,这种方法可以提供更多样化的结果,只是计算成本会更高。
对于已知病毒的识别,一种是基于k-mer,直接使用参考数据库与reads/contigs进行比对,如kraken2、bracken、CLARK和Centrifuge,
另一种是先翻译序列,然后与参考蛋白质数据库进行比对,如kaiju、DIAMOND和MMseqs2。
还有基于算法的,如BLAST或DUDes,它们使用DUD(Deepest Uncommon Descent)算法。
使用基因标记的,如MetaPhlAn4、IGGsearch和GOTTCHA。
也有专门用来研究病毒组的工具,如VirusTaxo、Metavir2和DeepVirFinder,其主要算法是卷积神经网络(CNN)。
其中如MetaPhlAn4和MCP (Microbiota Community Profiler),包含未知的宏基因组组装基因组的序列,而MCP只能用于识别微生物区研究中的细菌、古菌、真核生物和病毒序列。
由于每个用于物种注释的工具的性能都不同,且都使用了各种算法和参考数据库,所以这种多样性也会导致不一样的结果、耗时和计算成本。
▪ 基于k-mer的物种注释工具似乎是计算效率最高的,虽然它们需要很大的内存。
▪ 基于标记的,对内存的要求较低,但它们只能对来自特定区域的reads/contigs进行注释分类。
▪ 基于比对的工具要比其他的计算成本更高。
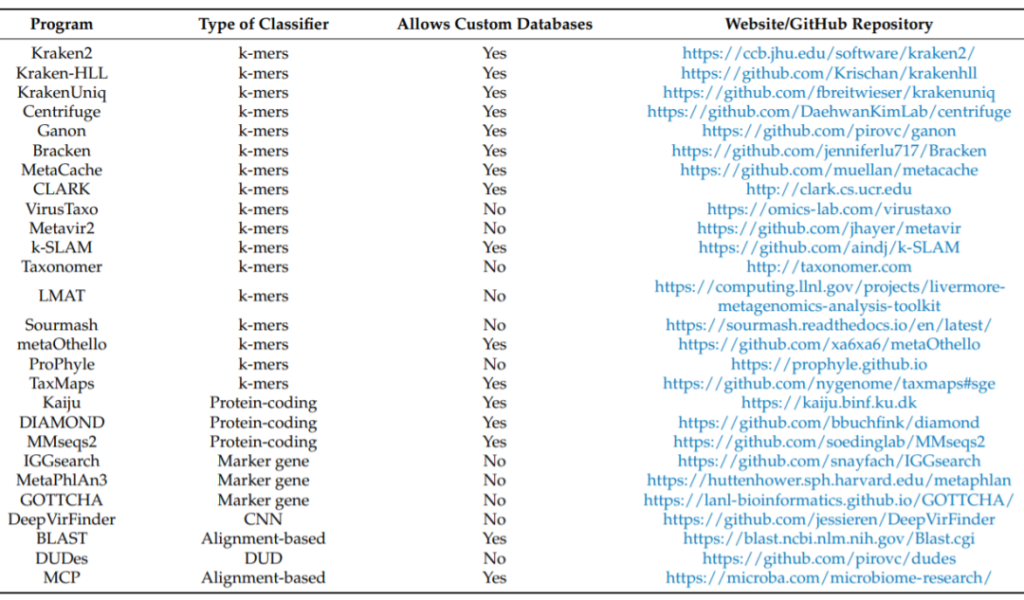
对于新型病毒的识别,现在也有不需要任何参考就能识别病毒序列的工具,即:
• VirSorter(https://github.com/simroux/VirSorter)
• VirFinder(https://github.com/jessieren/VirFinder)
VirFinder是一个基于k-mer的R包,可以以较好的预测识别病毒的contigs;
而VirSorter可以在不同的微生物数据集中识别新的病毒序列。
宏基因组分箱
在物种注释之前可以选择是否执行分箱(binning)。
分箱的主要目的是根据物种对contig进行聚类。根据数据类型,可使用的工具如下图:

CONCOT,它可以根据核苷酸组成和覆盖率数据对宏基因组contigs进行聚类。
GraphBin,它使用组装的连通性信息对contig进行集群化。
但宏基因组分箱并不局限于contigs,对于长读,可使用MEGAN-LR、BusyBee或LRBinner。
近年来,宏基因组学领域取得了许多进展,新技术可以帮助研究人员发现新的病毒,预测疫情,诊断某些疾病等。
长读测序平台也在快速发展,以得出更可靠的结果助力宏基因组分析。虽然已有许多工具和流程被开发出来以便更快更简单地进行数据分析,但还需要进一步发展,例如在数据处理分析中的通用指南的建立,因为虽然出于同一种目的而开发的工具,但由于计算过程不一样,它们在不同任务中的性能缺乏共识。此外,重要的是保持相关数据库的更新与维护。
宏基因组学检测人类样本中任何基因组(包括细菌、病毒、寄生虫和真菌)的能力,对于传染病的诊断具有重要意义。宏基因组学方法也已应用于其他几个研究领域:环境研究(如海洋样本、土壤、污水、农场灰尘) ;7000 年前青铜时代人类样本中的病毒感染;健康、疾病和法医调查中人体肠道微生物组的特征;临床研究 ; 以及新病毒病原体的发现,例如 SARS-CoV-2等。
Ibañez-Lligoña M, Colomer-Castell S, González-Sánchez A, Gregori J, Campos C, Garcia-Cehic D, Andrés C, Piñana M, Pumarola T, Rodríguez-Frias F, Antón A, Quer J. Bioinformatic Tools for NGS-Based Metagenomics to Improve the Clinical Diagnosis of Emerging, Re-Emerging and New Viruses. Viruses. 2023 Feb 20;15(2):587. doi: 10.3390/v15020587. PMID: 36851800; PMCID: PMC9965957.
谷禾健康
癌症是一种复杂的疾病,归因于多因素变化,导致治疗策略困难。
90%的癌症患者死于复发或转移。癌症转移是恶性肿瘤进展的关键步骤,由癌细胞内在特性和外在环境因素决定。
一些微生物组通过诱导癌性上皮细胞和慢性炎症促进癌发生、癌症进展和调节癌症治疗。
关于微生物群在肿瘤发生和临床效率中的作用的大部分认知都与肠道微生物群有关。
然而,研究也证实了肿瘤内微生物群在癌症中的作用。近年来,肿瘤内微生物群已被确定为肿瘤的一个组成部分,并可能在功能上调节转移的各个方面。
肿瘤内微生物群与区分正常组织与癌组织、药物反应者与无反应者癌症、良好与不良预后、转移性与非转移性癌症有关。
肿瘤内微生物群的调节可以减少癌症转移,阻止癌症进展,并重新编程免疫反应。
本文主要集中于肿瘤内微生物群的发现和表征及其在肿瘤转移过程中的独特功能,并讨论了癌症治疗的挑战和意义。
癌症转移通常被定义为:
肿瘤从原始肿瘤部位转移到远端器官的多步骤过程。
这一过程涉及几个步骤,包括入侵、传播、血管内、外渗、定植。
转移的一个关键特征是其极低效率,这是由于癌细胞在成功到达并定居目的地之前,需要应对许多物理、化学和生物挑战。
转移级联期间的应激源包括:
• 细胞外基质(ECM)僵硬
注:肿瘤细胞外基质的硬度约为周围正常组织的1.5倍
• 失巢凋亡
注:失巢凋亡是由于细胞与细胞外基质和其他细胞失去接触而诱导的一种特殊的程序化细胞死亡形式,在机体发育、组织自身平衡、疾病发生和肿瘤转移等方面起重要作用。
• 流体剪切应力
注:压缩、拉伸、剪切力导致的组织变形导致组织液在细胞周围运动。
• 化疗
注:使用化学治疗药物杀灭癌细胞达到治疗目的。
• 免疫监视
注:免疫系统具有识别、杀伤并及时清除体内突变细胞,防止肿瘤发生的功能,称为免疫监视。
确定转移效率的关键是:
了解早期转移细胞如何能够抵抗这些挑战并增强其对不同环境的适应性,以及每种类型的压力对最终转移效率的影响程度。
转移是一个低效的多步骤易位过程
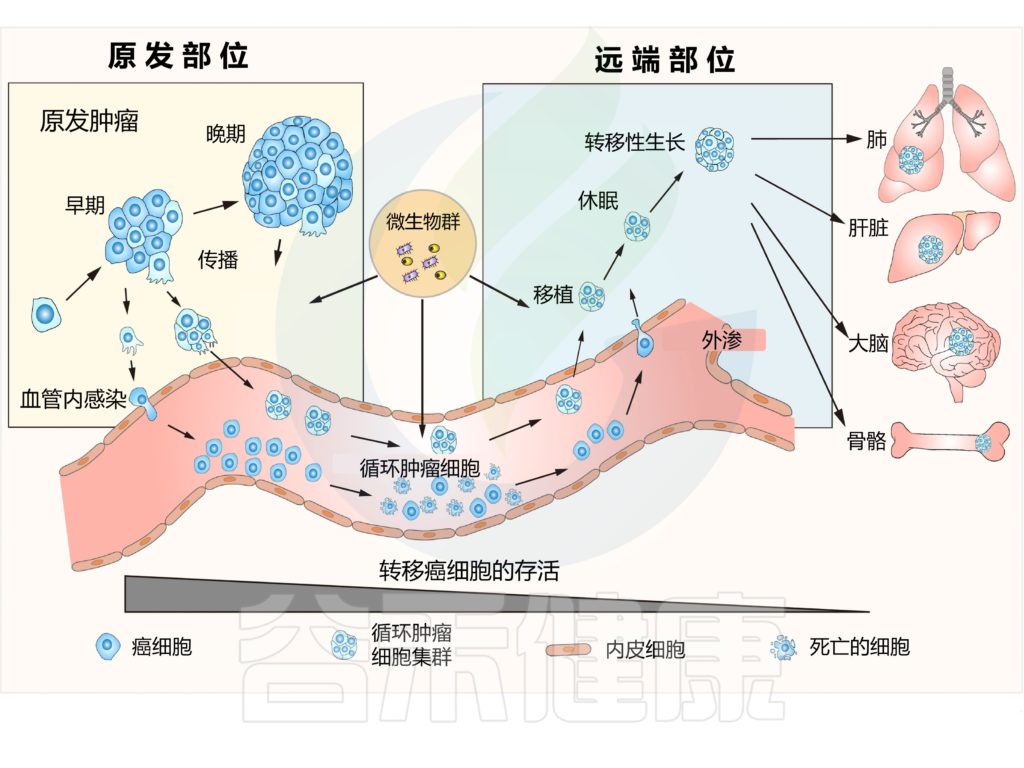
doi.org/10.1016/j.tcb.2022.11.007
新的研究扩大了我们对转移的认知。例如,研究表明转移开始发生在肿瘤进展的非常早期。
集体侵入相邻组织
在这些转移细胞到达远端器官之前,癌细胞甚至可以通过分泌成分远程准备转移前生态位(PMN)。当转移细胞开始迁移时,它们通常会集体侵入相邻组织,并作为寡克隆细胞簇在血流中传播,以增强其定植新生态位的能力。
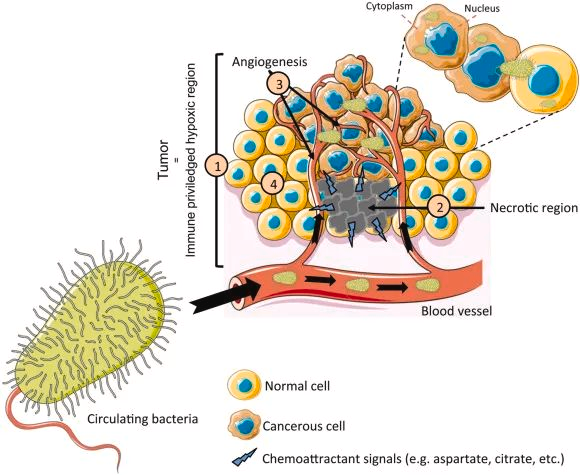
doi.org/10.1016/j.canlet.2021.09.009
改变代谢程序,逃避免疫监视
这些先驱转移起始细胞改变它们的代谢程序以增强它们的转移潜能,并且可以逃避免疫监视并长时间保持休眠状态,直到开始分裂。
转移能力高度依赖于癌细胞内部细胞特性
这些研究使我们对转移细胞生存策略的理解更进一步,并证实了癌细胞转移能力高度依赖于癌细胞内部细胞特性的观点,例如 EMT 状态、干细胞可塑性、遗传学、表观遗传学、染色体不稳定性和代谢适应,以及环境因素,如机械压力、免疫反应、ECM、PMN 和肠道微生物组。
那么,癌细胞获得这些转移性状的驱动力是什么?

在实验上,肿瘤内微生物群已被确定为组织的一个组成部分。这些肿瘤内细菌是癌症进展不同阶段的新参与者,可以从外部相互作用和细胞内部影响癌细胞。
下面一个章节,我们来看肿瘤内微生物群是什么,有什么作用?
我们知道,已经有越来越多的文章阐述肠道微生物组在癌症进展中的作用,这方面我们的理解在迅速增长,然而我们对肿瘤内微生物群的理解仍处于初级阶段。
近期与转移相关的肿瘤内微生物群的研究

doi.org/10.1016/j.tcb.2022.11.007
人类组织,包括癌组织,通常被认为是无菌的,除了结肠、皮肤和口腔。
▸ 肿瘤内微生物群
癌症生物学的最新概念进展是,鉴定出癌症组织中存在微生物群。这些肿瘤组织驻留细菌被归类为“肿瘤内微生物群”。
我们知道,肠道微生物群可以通过代谢产物或通过与免疫细胞的相互作用,远距离影响肿瘤组织。
而肿瘤内微生物群与癌细胞密切接触,因此可能与肠道微生物群有不同的功能模式。
我们其他文章有对肠道微生物组在癌症诊断、预后和治疗反应中的作用进行详细介绍:
肠道微生物群与五种癌症的相互作用:致癌 -> 治疗 -> 预后
因此,本文主要集中于肿瘤内微生物群的发现和表征及其在肿瘤转移过程中的独特功能。
▸ 肿瘤内微生物群发现的证据:
-早前提出假设
一百多年前,威廉·科利发明了科利毒素(化脓性链球菌和粘质沙雷菌的混合物)来治疗一位癌症患者,并观察到肿瘤消退。
他假设“每一种恶性肿瘤都可能有外源性或微生物来源”。然而,在这个假设之后的几十年里,没有直接证据表明肿瘤内细菌的存在。
瘤内微生物群研究的重大突破包括发现、机制等成果
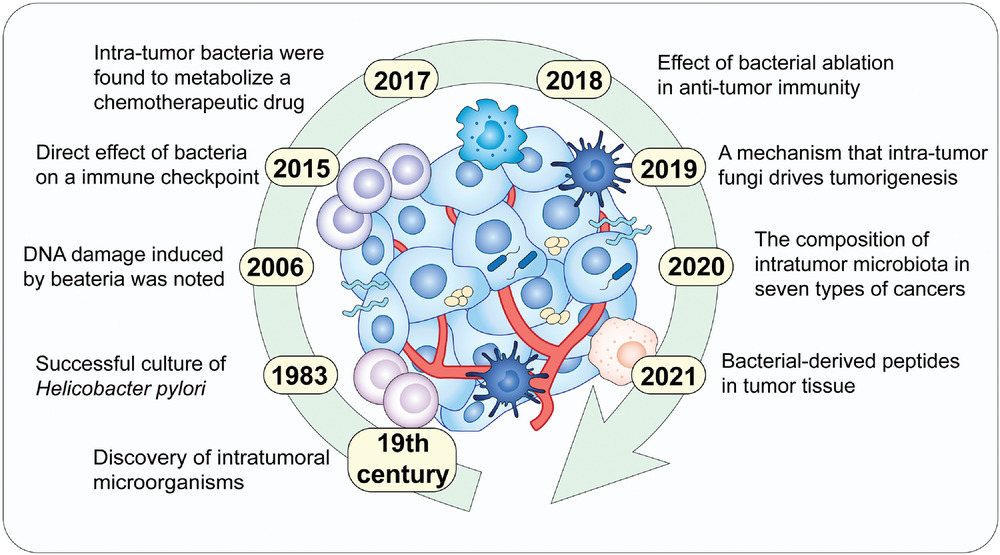
doi.org/10.1002/advs.202200470
-攻克瘤内微生物的检测技术挑战
到现在,下一代测序技术 (NGS) 能够使用 16S rDNA 测序将细菌 DNA 与肿瘤组织区分开来,然而,由于瘤内细菌丰度低和宿主基因组污染严重,从组织处理或试剂中引入的环境噪声信号使数据收集变得复杂,因为它们会掩盖组织的真实微生物概况并削弱结论的稳健性。
这些技术挑战在过去几年已被攻克,多个研究小组报告了大量数据,进一步支持瘤内微生物群的存在。此外,生物信息学微生物特征能够区分健康个体和癌症患者。
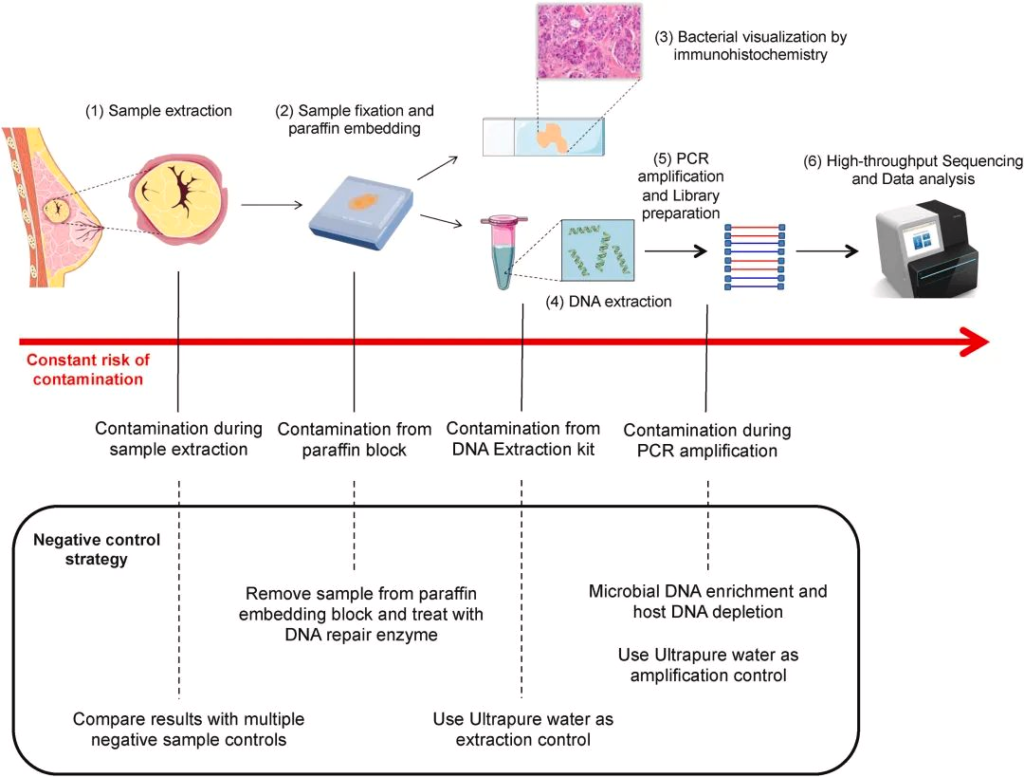
doi.org/10.1016/j.canlet.2021.09.009
识别肿瘤微生物组为癌症研究领域开辟了新的机遇。更好地表征肿瘤内微生物组可能会导致开发新的治疗方法,从而克服传统的癌症治疗方法。下一代测序方法,包括 16S 扩增子测序,可以在组织提取和石蜡固定后,将肿瘤内细菌精确地聚集在确定的细菌亚群中。
此外,宏基因组学对于肿瘤内微生物的鉴定也很重要。
宏基因组
宏基因组是一种针对样本中所有 DNA 的非靶向测序方法,包括微生物群落的全基因组序列,广泛应用于复杂微生物组的分析。宏基因组的分辨率更高,可以达到物种甚至菌株水平。此外,宏基因组学可以提供功能信息。
此外,宏基因组学可以与转录组分析结合使用,以消除死亡微生物和细胞外DNA造成的干扰。
最近的研究表明,最新的宏基因组数据涵盖了更多类型的癌症,这可能促进肿瘤内微生物群领域的新进展。
在瘤内微生物研究中,宿主DNA和环境微生物DNA的污染是最大的障碍。因此,需要开发从 TCGA 中丢弃不可信数据的方法。
在一项分析多种癌症的研究中,研究人员删除了总序列数据的 92.3%,以确保分析中数据的可靠性。2021 年,Dohlman 等人开发了一种去污染算法,可以去除 TCGA 数据中的污染。
随着这些方法的发展,宏基因组学可以为肿瘤内微生物群的研究提供更有力的支持。
▸ 细菌是各种癌症类型中肿瘤组织不可或缺的组成部分和活的居民
各种癌症类型有不同的微生物群。
肿瘤内微生物群的组成与许多类型的癌症有关。器官和组织包括食道、肺、乳腺、前列腺、膀胱、胃、肾、肝、胰腺等,以前被认为是无菌的。下一代测序显示这些器官含有低生物量微生物群。瘤内微生物组是肿瘤微环境的主要组成部分,影响肿瘤发生、疾病进展、耐药性和预后。
不同癌症类型的肿瘤内微生物群生态位
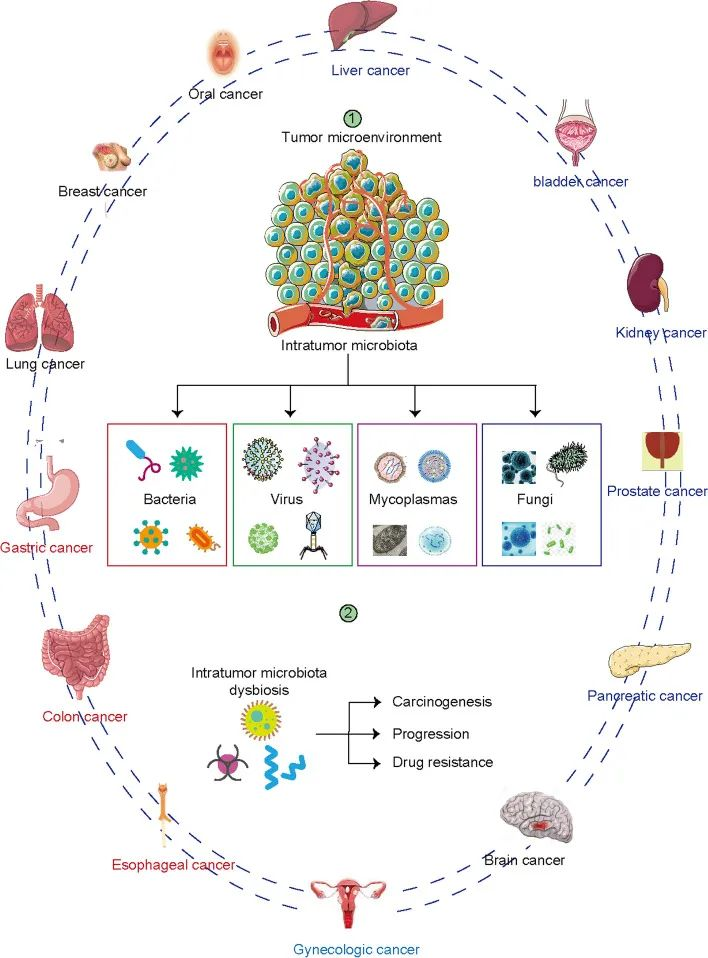
Liu J, et al., Biomark Res. 2022
在暴露于环境的组织(如肺癌和黑色素瘤)中并未发现微生物群丰度最高,而是在乳腺癌,骨癌,胰腺癌中。这表明肿瘤内微生物群的丰度是肿瘤特异性的。
作为癌症生态系统不可或缺的组成部分的肿瘤内微生物群
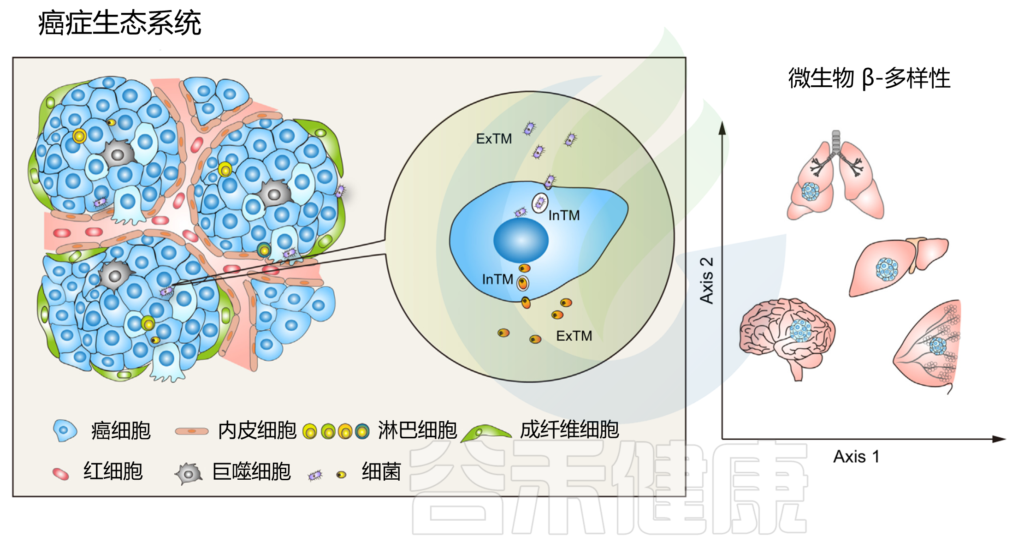
doi.org/10.1016/j.tcb.2022.11.007
如果肿瘤内微生物群存在于广泛的癌症类型中,那么它们来自哪里?
很少有研究专门去调查其原始来源。然而,对来自肿瘤组织的分离细菌菌株的分析提供了一些见解。
在小鼠乳腺肿瘤中,在正常组织对应物中检测到肿瘤内细菌菌株,这表明肿瘤组织从周围组织获得某些细菌。这些细菌菌株在体内的主要栖息地是多种多样的,有皮肤上的葡萄球菌、口腔中的链球菌和肠道中的肠球菌。
鉴于细菌具有在组织之间传播的能力,肿瘤内微生物群可能有多个起源。对鼻咽癌的分析表明,瘤内细菌主要来自鼻咽部,一小部分来自口腔和肠道。
* 也需要通过宏基因组比较和基因追踪分析来进一步加强。
肿瘤内微生物群的来源
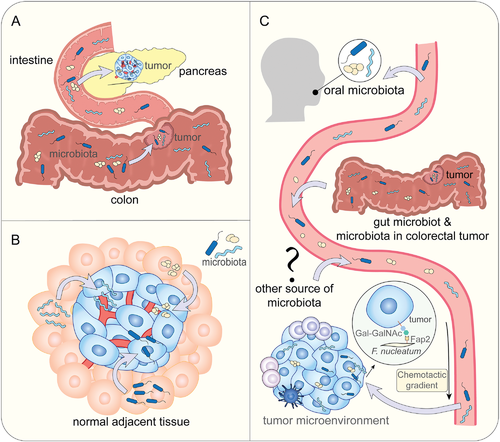
doi.org/10.1002/advs.202200470
A) 通过粘膜屏障从粘膜部位产生的肿瘤内微生物
B) 从正常邻近组织产生的肿瘤内微生物
C) 肿瘤内微生物是血行传播的结果
瘤内细菌的共同特征
1- 丰度低
它们在癌组织中的丰度远低于肠道中的丰度,根据 qPCR 定量和成像定量,0.1-10% 的癌细胞携带细菌,不同的量化方法和/或细菌 DNA 的提取效率引入了差异。
2- 多样性低
癌组织中微生物群落的多样性通常低于正常组织,这表明肿瘤可能形成一个独特的环境,选择性地扩展某些细菌种类。
3- 活的
这些细菌是活的。主要是主要存在于细胞内空间的共生生物。癌组织中不同的细菌栖息地可能与其在与癌细胞相互作用时的多效性作用模式有关。
细胞内外微生物群功能不一
鉴于细胞内和细胞外空间之间存在巨大的分子、生物化学和生物物理学差异,在肿瘤起始、肿瘤进展过程中,与细胞外肿瘤驻留微生物群 (ExTM) 相比,细胞内肿瘤驻留微生物群 (InTM) 可能具有完全不同的功能和免疫相互作用。
长期以来,细胞内细菌一直被研究为参与病原体-宿主相互作用的致病菌菌株。致病菌通过“触发”或“拉链”模式侵入宿主细胞,并能够迅速破开核内体膜进入细胞质。
肿瘤内共生细菌是遵循相同的原则还是使用不同的机制来侵入癌细胞?这方面仍知之甚少。在特定的癌症类型中,如乳腺癌,肿瘤内微生物群落主要以革兰氏阳性和兼性厌氧细菌为主,这表明肿瘤微环境具有选择效应。
不同的肿瘤类型具有不同的血管生成和氧水平、内吞作用和微胞作用以及周围组织中的微生物来源。这些因素共同决定肿瘤内微生物群的组成,并形成肿瘤类型特异性特征。
肠道菌群刺激特定代谢物的产生,调节免疫系统,并重建远端器官的微环境。相比之下,专门研究肿瘤内微生物群在癌症转移中的作用的研究有限。
这个领域的研究还比较浅,缺乏合适的实验工具来准确和特异性地调节肿瘤内的微生物群,同时又不扰乱身体其他部位的共生细菌。这个问题可以通过使用各种抗生素给药方案、使用无菌小鼠和原位细菌再给药来部分解决。
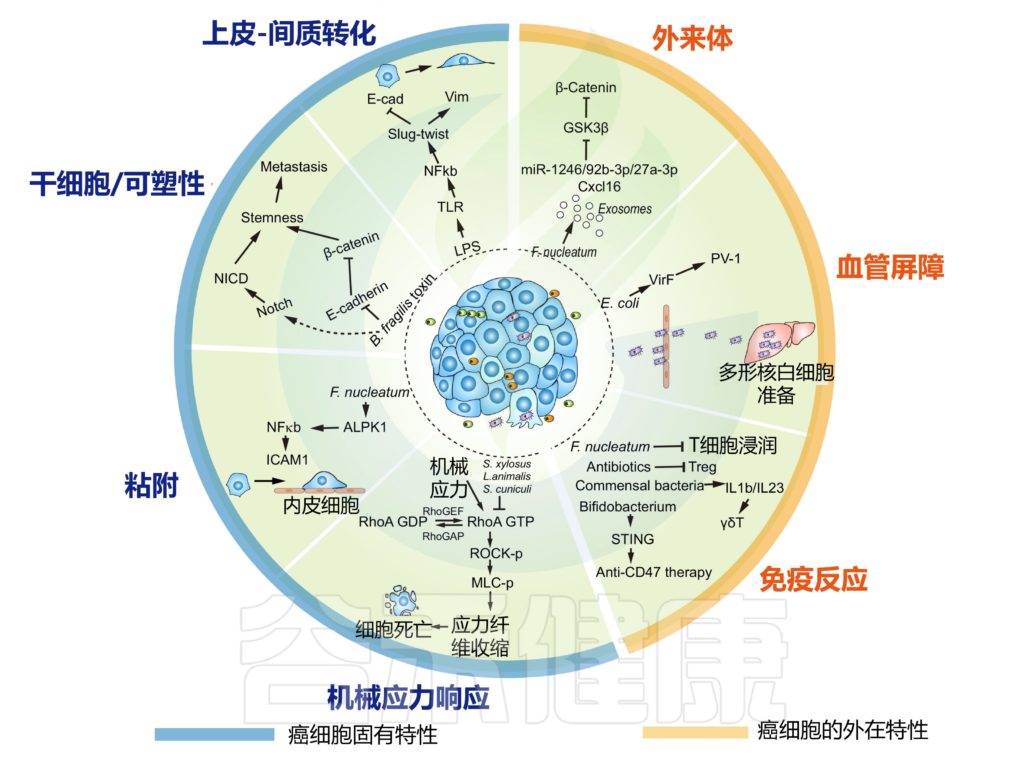
越来越多的证据证实,瘤内细菌可以调节癌细胞的内在特性及其外部环境,从而增强癌细胞的能力并为癌症转移铺平道路。
为了克服转移过程中的物理、化学和生物学挑战,癌细胞通常会改变其内在程序以应对不利的环境。这些包括干细胞程序/可塑性(用于新位点的肿瘤起始)、EMT 程序(用于癌症侵袭和传播)、粘附程序(防止失巢凋亡诱导的细胞死亡)和机械应激反应程序(抵抗机械力诱导的损伤) 。
研究表明,这些程序也可以通过肿瘤内微生物群进行调节。
肿瘤内微生物群改变癌细胞的内在特性并重塑转移中的肿瘤微环境
doi.org/10.1016/j.tcb.2022.11.007
我们先来看看,EMT程序是什么?
EMT程序赋予癌细胞迁移性间充质特征,具有松散的细胞间粘附特性,可动员癌细胞进行侵袭和扩散。这是由 TGFβ 信号通路的激活和与 Zeb、Twist 和 Snail 相关的协调转录程序驱动的。
微生物群和EMT程序之间有关联吗?
答案是肯定的。多项研究表明微生物群与 EMT 之间存在相关性。
在人类乳腺癌细胞系中,肿瘤驻留脆弱拟杆菌分泌的毒素诱导迁移和侵袭表型,EMT 相关的 Slug 和 Twist 的表达升高。在位于乳腺导管的肿瘤细胞中,脆弱拟杆菌的定植刺激了远端器官转移的增强。
这种功能调节是否仅限于细胞外肿瘤驻留微生物群,还是也适用于细胞内肿瘤驻留微生物群,以及不同的肿瘤驻留细菌对 EMT 的影响有多普遍,仍然是一个悬而未决的问题。
然而,有证据表明,脂多糖能够在依赖于 TLR-NFκB 通路的正常人肝内胆管上皮细胞中诱导 EMT.
在 EMT 驱动的小鼠结肠癌模型中,微生物群的存在对于肿瘤的发展至关重要。
这些研究支持组织驻留微生物群与 EMT 计划之间存在联系。
癌细胞的可塑性和干性是转移启动的另一个重要因素。
研究发现,脆弱拟杆菌毒素可以裂解 E-cadherin,触发下游 β-catenin 核定位,伴随 Notch 效应子 NICD 在乳腺癌中的核聚集。
在小鼠移植肿瘤模型中,Wnt 和 Notch 信号通路的后续激活,导致干性和肿瘤生长以及转移进展。
在自发性 MMTV-PyMT 乳腺肿瘤模型 [具有多瘤病毒中间 T 抗原 (PyMT) 的小鼠乳腺肿瘤模型在小鼠乳腺肿瘤病毒 (MMTV) 长末端重复序列下表达],各种肿瘤驻留细菌物种侵入 PyMT 癌症细胞触发了乳腺干细胞程序的富集。 由于与细菌侵入的癌细胞的体内分离相关的挑战,尚不清楚干细胞程序是否可以在生理细胞环境中被肿瘤内细菌激活。
癌细胞渗入血流引发细胞死亡程序
癌细胞渗入血流伴随着粘附丧失,这通常引发失巢凋亡,或其他形式的细胞凋亡的细胞死亡程序。癌细胞表面粘附分子的表达增强了它们的存活,并防止了转移失败。
在人类结直肠癌细胞系中,结直肠癌中常见的具核梭杆菌通过上调粘附分子 ICAM1 显着增强癌细胞对内皮细胞的粘附。这种增强的粘附力使癌细胞能够在尾静脉注射测定中外渗并引发新的转移灶。ICAM1 的上调部分是通过细菌依赖性激活 Alpk1-NFκB 通路实现的。
循环癌细胞受机械应力的影响导致细胞损伤
除了失巢凋亡依赖性细胞死亡外,循环癌细胞还会受到血液中各种机械应力的影响,从而导致细胞损伤,例如流体剪切应力,并在远端器官中,导致结构限制。
这些应激源部分被粘附分子(如整合素)感知,由 RhoGTPase 信号级联传递,并由 Yap/Taz 转录因子协调。
小鼠肿瘤模型的新发现表明,InTM 在侵入宿主癌细胞时会触发流体剪切应力反应,并且这种反应与细菌物种促进转移的能力相关。
被细菌侵入的癌细胞可以携带细菌,游走至远端器官,促进癌细胞的存活。这种表型是 InTM 特有的,因为通过调节 RhoAGTPase-Rock-actin 细胞骨架重组途径,癌细胞变得更能抵抗机械应力。引发这种反应的细菌机制仍不清楚。
然而,从肉毒梭状芽胞杆菌中分离出来并被多种细菌共享的 ADP-核糖基转移酶 C3 胞外酶是一个潜在的候选者,因为 C3 对细胞是不可渗透的,并且与膜穿透肽融合的 C3 经常被细胞生物学家用来解离肌动蛋白应力纤维并增强细胞扩散。
除了直接调节癌细胞外,瘤内细菌是重要的炎症介质,可以在癌细胞周围形成特定的微环境,从而间接促进癌症转移。
调节 PMN 的关键因素之一是细菌本身
结直肠癌研究表明,肿瘤驻留细菌能够通过毒力因子 VirF 调节肠道血管屏障。PV-1 表达升高的血管屏障受损,促进了细菌从原发性结直肠肿瘤传播到肝脏,并在癌细胞到达之前建立了 PMN.
注:PMN-迁移前生态位
患者体内较高的 PV-1 水平与较高的细菌负荷和较远的转移有关。这种依赖于细菌的 PMN 远程控制是一个新概念,可能对癌症以外的疾病有影响。
肿瘤外泌体可以调节 PMN 并决定转移器官的趋向性
肿瘤外泌体含有多种功能性脂类、蛋白质、RNA和DNA,释放到细胞外环境中调节靶细胞,重塑微环境。
源自具核梭杆菌侵入的人结直肠癌细胞,分离出含有 miR-1246/92b-3p/27a-3p 和 Cxcl16 的外泌体。这些外泌体在调节结直肠癌细胞迁移方面发挥作用,并通过靶向 GSK3β 激活 Wnt-β-catenin 信号通路显著增加肺转移。
这意味着邻近的癌细胞不一定需要被细菌侵入才能转移;相反,它们也可以通过旁分泌外泌体信号来动员以启动转移。
瘤内细菌最显着的特征之一是它们可以被免疫系统识别,从而触发特定的免疫反应
有许多关于肠道菌群失调与异常免疫反应之间关联的报道,但肿瘤内微生物群在调节免疫系统中的作用仍不清楚。
一方面,抗生素治疗和细菌再给药试验显示肿瘤内细菌抑制免疫反应的证据
在乳腺癌中,瘤内具核梭菌以免疫介导的方式加速肿瘤进展和肺转移,瘤内给药具核梭菌减少浸润的 CD4+ 和 CD8+ T 细胞。
在小鼠黑色素瘤癌症模型中,肺组织的抗生素治疗降低了细菌负荷,显示出调节性 T 细胞减少,T 细胞和自然杀伤 (NK) 细胞活化增强,同时肺转移显着减少。
在转基因小鼠肺癌模型中,肺部共生细菌激活了 γδT 细胞,这是一种 T 细胞亚群,通过刺激骨髓来源的 IL1β 和 IL23 并引发肿瘤炎症来促进淋巴和骨髓谱系的炎症反应。
另一方面,肿瘤内细菌可以触发抗肿瘤免疫。
例如,益生菌(鼠李糖乳杆菌)的施用强烈促进了针对小鼠黑色素瘤肺转移的肿瘤免疫。
此外,瘤内注射双歧杆菌可刺激 STING 通路,增加树突状细胞数量,并促进基于抗 Cd47 的免疫治疗。
因此,肿瘤内细菌的免疫调节作用是复杂的,并且依赖于环境,并且可能是细菌物种特异性的和/或受其细胞内/细胞外居住状态的高度影响。
传统癌症疗法的限制
迄今为止,主要的癌症疗法基于手术、放疗和化疗。尽管对大多数确定的肿瘤有效,但它们都有缺点,依赖于冗长、乏味的程序,非特异性地对抗肿瘤,通常无法区分恶性组织和健康组织。
由于缺乏对肿瘤样区域的特异性,某些癌细胞得以存活并定植在附近的组织中,从而导致潜在的癌症复发。靶向健康组织可能会产生意想不到的副作用,从而导致严重的致癌 DNA 损伤。
所有这些缺点,加上对治疗产生耐药性的持续风险,与癌症死亡率和发病率的增加有关。
90%的癌症患者死于复发或转移。
肿瘤内微生物群的作用可以通过具有肿瘤内微生物群信息的癌症患者的生存数据来评估。
在胰腺癌患者中,与短期幸存者相比,长期幸存者往往具有更高的微生物群落多样性。
此外,肿瘤内微生物群特征(假黄单胞菌Pseudoxanthomonas–链霉菌Streptomyces–糖多孢菌Saccharopolyspora –克劳氏芽孢杆菌Bacillus clausii)被确定与生存相关。
在其他癌症类型中,尽管样本量有限,但据报道特定的肿瘤内微生物组特征也与转移有关。
在对 800 多个患者样本进行分析的鼻咽癌临床研究中,肿瘤内细菌载量被确定为一种强有力的预后工具,可以区分恶性进展的风险。这些研究证实了肿瘤内微生物群的预后价值,并支持其在临床肿瘤进展中的作用。
然而,在临床上特异性调节肿瘤内微生物群具有挑战性。
有几项关于抗生素治疗和癌症风险、癌症反应和生存的回顾性研究,但它们很少专门设计用于剖析肿瘤内微生物群的消除和患者预后。
这些广泛的抗生素治疗数据分析报告了癌症发病率的增加和对免疫疗法的一般反应受损。鉴于已经确定肠道微生物组与免疫检查点抑制剂治疗密切相关,目前尚不清楚肠道肿瘤微生物组在调节癌症进展方面是否具有相似或不同的作用。
相比之下,一项胰腺腺瘤研究表明,抗生素治疗与晚期转移性胰腺导管腺癌的更好预后相关。
鉴于抗生素在效力、吸收效率、细胞渗透性以及给药途径和时间窗的可变性方面存在巨大差异,所有这些变量都可能导致肠道微生物组和细胞内/细胞外肿瘤微生物组概况的根本差异。因此,迫切需要精心定义的肿瘤内微生物群调节临床研究集。
肿瘤内微生物群数据在癌症筛查和治疗中的应用
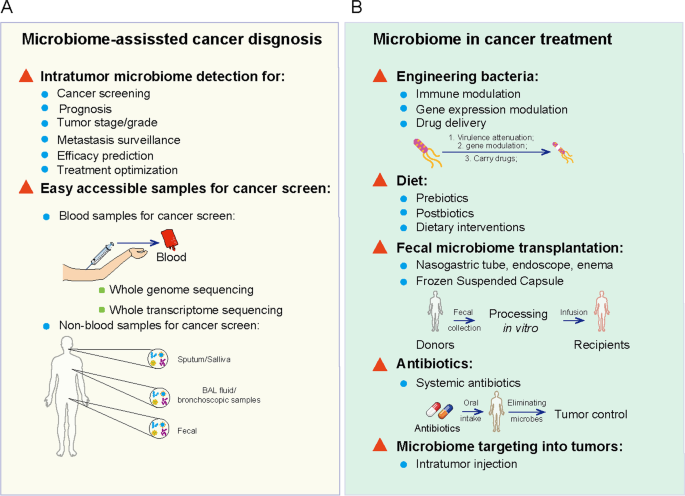
Liu J, et al., Biomark Res. 2022
A) 来自临床样本的数据可能有助于开发新的癌症筛查和预后,包括来自肿瘤部位和易于获取的样本的微生物群模式。
B) 肿瘤内微生物群可用于癌症治疗,包括工程菌、饮食调节、粪便微生物组移植、抗生素和肿瘤内微生物组注射等。
新兴研究揭示了肿瘤内微生物群在癌症转移的各个步骤中的生物学功能。这些肿瘤内微生物群不仅是肿瘤环境的传感器、肿瘤病理类型、药物反应和预后的指标,而且在功能上也参与肿瘤进展。
肠道细菌的宿主内进化会导致共生菌株变成致病。因此,需要进一步的研究来测试肿瘤内细菌促进癌症转移的能力是否源于细菌进化。这或许可以解释不同的细菌种群及其在正常组织和癌组织中的各种功能,以及为什么某些肿瘤类型比其他肿瘤发展得更快。
未来,肿瘤内微生物领域将受到更多关注,该领域有四个方面可能成为未来研究的重点:
肿瘤内微生物群可以作为癌症筛查的生物标志物。
包括肿瘤内微生物组衍生的个性化数据,这些数据可以将食管癌、胰腺癌、肺癌和口腔癌患者与健康人区分开来。分析肿瘤内微生物群特征,可能为患者的预后提供潜在的生物标志物。
此外,肿瘤内微生物群为癌症治疗带来新的机遇。
考虑到肿瘤内微生物群的异质性,个性化治疗策略因其高效和靶向作用而具有吸引力。
肿瘤内细菌的细胞外和细胞内定位使它们成为药物载体的完美候选者,可以在肿瘤细胞内外递送,以倒带细胞间和细胞内信号网络。
与其他抗肿瘤疗法一样,细菌疗法和抗生素也可以与其他疗法结合使用,例如免疫疗法和化学疗法。
使肿瘤内微生物群正常化和移植某些微生物也是提高抗肿瘤治疗效率的潜在策略。
癌症疗法正面临着巨大的转变:传统疗法正逐渐被更精确和复杂的疗法所取代。了解肿瘤内微生物群对癌症发生和发展的不同贡献,将有助于制定癌症预防和治疗策略。
主要参考文献:
Fu A, Yao B, Dong T, Cai S. Emerging roles of intratumor microbiota in cancer metastasis. Trends Cell Biol. 2022 Dec 13:S0962-8924(22)00258-6. doi: 10.1016/j.tcb.2022.11.007. Epub ahead of print. PMID: 36522234.
Liu J, Zhang Y. Intratumor microbiome in cancer progression: current developments, challenges and future trends. Biomark Res. 2022 May 31;10(1):37. doi: 10.1186/s40364-022-00381-5. PMID: 35642013; PMCID: PMC9153132.
An Y, Zhang W, Liu T, Wang B, Cao H. The intratumoural microbiota in cancer: new insights from inside. Biochim Biophys Acta Rev Cancer. 2021 Dec;1876(2):188626. doi: 10.1016/j.bbcan.2021.188626. Epub 2021 Sep 11. PMID: 34520804.
Heymann CJF, Bard JM, Heymann MF, Heymann D, Bobin-Dubigeon C. The intratumoral microbiome: Characterization methods and functional impact. Cancer Lett. 2021 Dec 1;522:63-79. doi: 10.1016/j.canlet.2021.09.009. Epub 2021 Sep 10. PMID: 34517085.
Wang Y, Guo H, Gao X, Wang J. The Intratumor Microbiota Signatures Associate With Subtype, Tumor Stage, and Survival Status of Esophageal Carcinoma. Front Oncol. 2021 Oct 27;11:754788. doi: 10.3389/fonc.2021.754788. PMID: 34778069; PMCID: PMC8578860.
Xie Y, Xie F, Zhou X, Zhang L, Yang B, Huang J, Wang F, Yan H, Zeng L, Zhang L, Zhou F. Microbiota in Tumors: From Understanding to Application. Adv Sci (Weinh). 2022 Jul;9(21):e2200470. doi: 10.1002/advs.202200470. Epub 2022 May 23. PMID: 35603968; PMCID: PMC9313476.
Huang Y, Zhu N, Zheng X, Liu Y, Lu H, Yin X, Hao H, Tan Y, Wang D, Hu H, Liang Y, Li X, Hu Z, Yin Y. Intratumor Microbiome Analysis Identifies Positive Association Between Megasphaera and Survival of Chinese Patients With Pancreatic Ductal Adenocarcinomas. Front Immunol. 2022 Jan 25;13:785422. doi: 10.3389/fimmu.2022.785422. PMID: 35145519; PMCID: PMC8821101.

谷禾健康
宏基因组测序可以使我们深度全面地了解微生物群的构成,对于缺乏深度研究和高质量参考基因组的样本,宏基因组获得的较为完整的基因组不仅可以丰富参考基因组数据库,同时可以提供更加准确的物种分类。
关于宏基因组的介绍可见我们之前的文章:

在宏基因组分析过程中,可能遇到的问题,及问题相关解决思路如下:






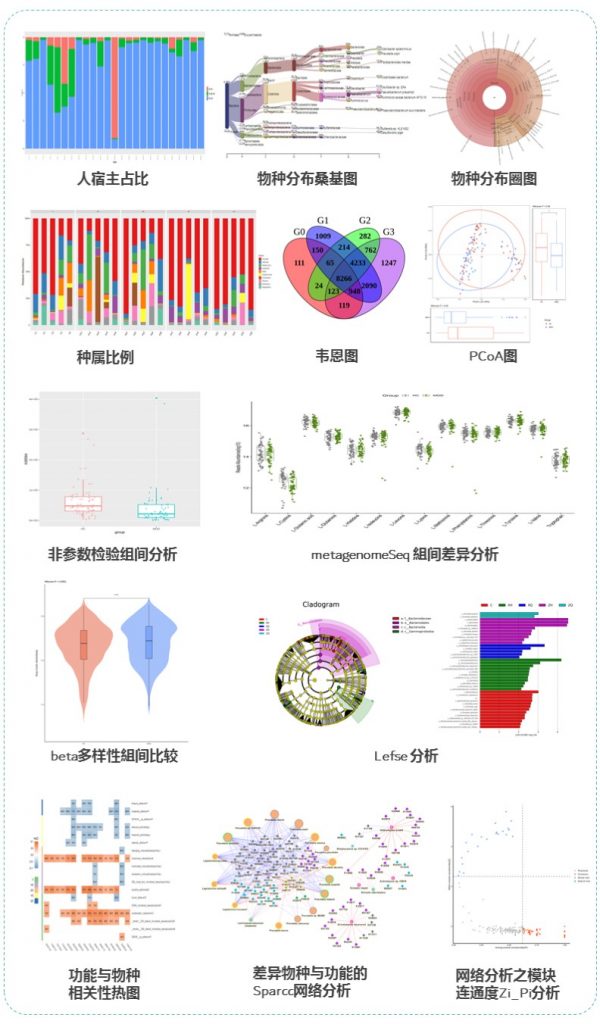








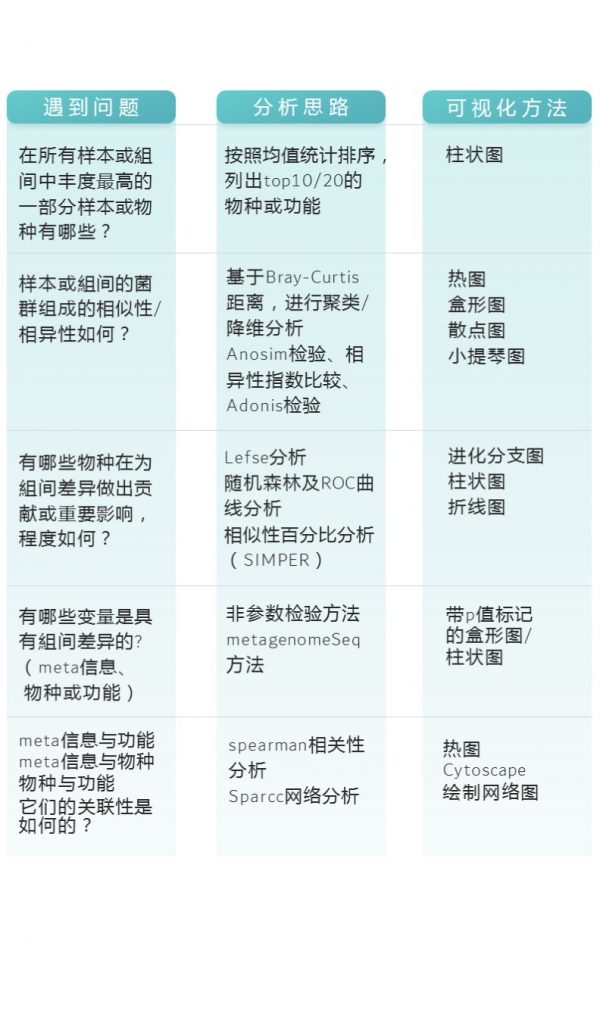






























更多关于宏基因组科研服务详询:
商务经理:13336028502(微信同号)

谷禾健康

尽管地球上微生物类群的繁多,但只有一小部分得到了培养和有效命名。因为大多数菌无法在非常特定的条件下培养分离鉴定。
在过去十年中,宏基因组研究的重要性已经凸显,因为它能够评估细菌基因库并发现当前实验室培养技术无法掌握的新细菌基因组。这些数据对于扩大我们对地球上微生物多样性的理解至关重要。
由于宏基因组测序数据由来自多个物种和菌株的 DNA 序列片段组成,通常有数千个来自不同生命领域,因此此类分析的主要挑战是正确确定每个 DNA 序列片段的真实来源。不幸的是,这些步骤容易出错,因此必须对结果进行严格审查,以避免发布不完整和低质量的基因组。
最近,比利时研究人员新开发MAGISTA,这是一种评估宏基因组基因组组装质量的新方法,基于随机森林的方法估计MAGs的完整性和污染度,解决了当前基于参考基因的方法经常被忽视的一些缺陷。
MAGISTA是基于宏基因组bins内的contig片段之间的无对齐距离分布,而不是一组参考基因。该方法利用了来自整个 bin 的信息。为了正确评估此方法,并说明基于参考的工具的缺点,最近,比利时研究人员构建了一个高度复杂的 DNA 模拟群落,由 227 个细菌菌株组成,并且具有不同程度的相似性。

方 法
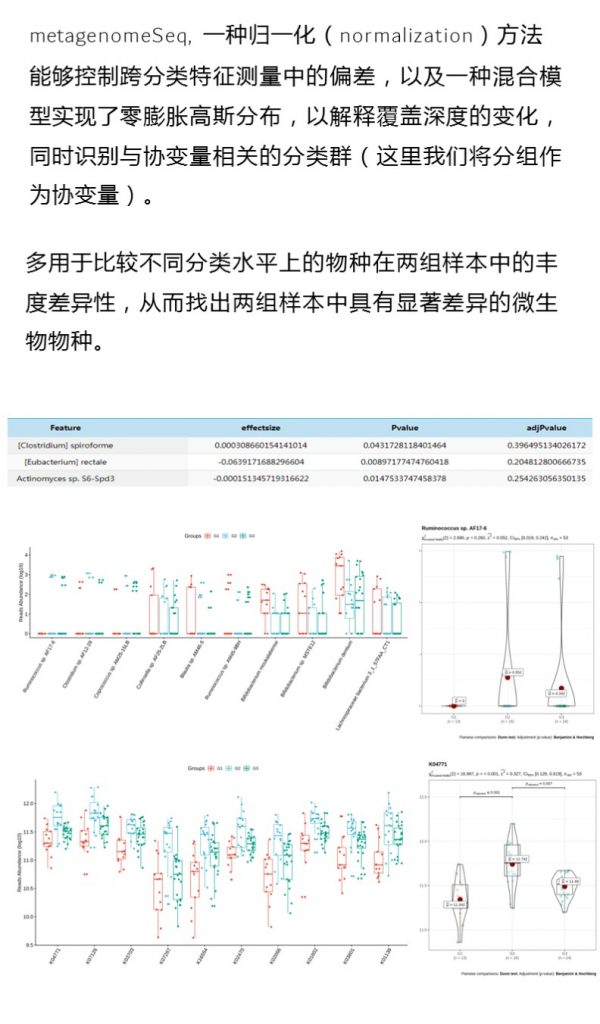
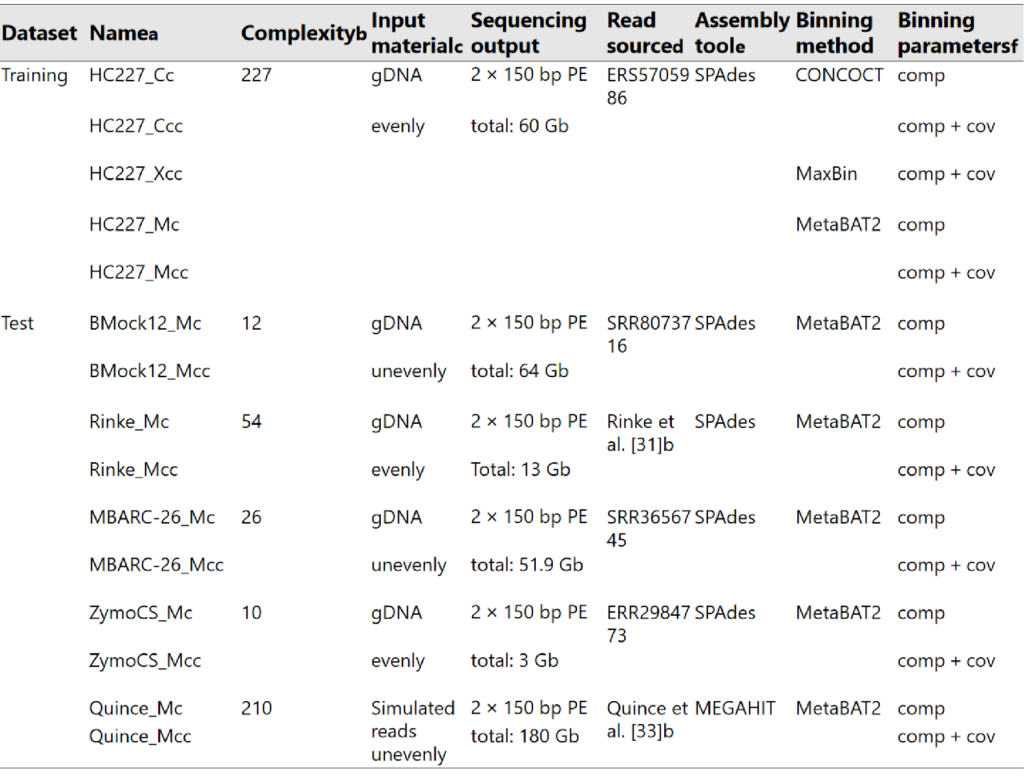
训练集来(HC227)自 227 个细菌菌株,测试数据集由五个公开可用的短读(short reads)子集构成,其中四个含有来自复杂度相对较低的基因组 DNA 模拟群落的reads。具体情况如下图所示。
Complexity列指示菌株数;Assembly tool列表示所使用的用于组装的软件;Binning method列表示所使用的用于分箱的工具;Binning parameters列表示所使用的用于评估分箱质量的指标,comp为完整度,cov为覆盖率。
MAGISTA计算步骤:
输入binning后的每个bins
-●-
第 1 步:选择适合的片段大小与距离计算方法
-●-
首先将每个 bin 中的每个 contig 拆分为固定长度的片段,然后使用四种不同的方法(即 PaSiT4、MMZ3、MMZ4 和 Freq4)计算一个 bin 中的片段之间的所有距离。对于每种方法,都选择了特定的片段长度,以便为不同的生物产生不同的特征分布。
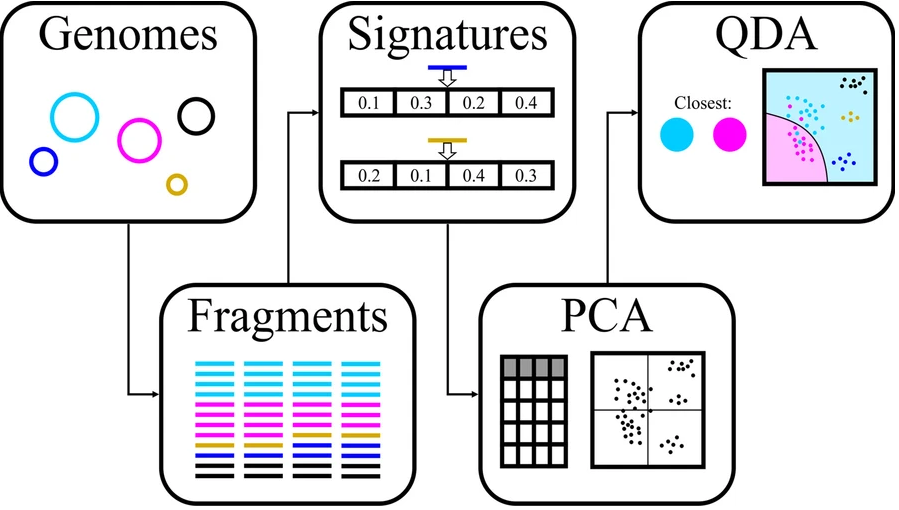
每种方法的最终片段长度的选择是通过不同方法分析整合决定的,方法如下图所示。每组的设计中至少两个基因组来自同一个家族,两个基因组来自相同的顺序但来自不同的家族。这些基因组被人为地分成所需长度的片段,并为每个片段计算目标特征。
对于每组五个基因组,混合所有片段并根据它们的特征进行主成分分析(PCA),然后进行二次判别分析,用于生成分类器,旨在区分每组中重叠最多的两个基因组。对该分类器的准确度取平均值,结果用于选择方法和片段长度的最终组合。
-●-
第 2 步:模型中特征变量的选择
-●-
为每种方法选择片段长度后,使用平均值、标准差、偏度、峰度和中位数以及 2.5%、5%、10%、90%、95% 和 97.5% 百分位数计算距离分布。此外,还计算了 1 kb 片段的 GC含量分布。以及每个bin的大小,共计66个特征变量。
-●-
第3步:模型构建
-●-
使用 R (v 4.0.3) 包“RandomForest”中的“RandomForest”函数和默认参数训练随机森林模型。同时使用R包lm再建立一个线性模型执行线性回归,输入经对数转换后的特征变量值,用于交叉验证分析。
主 要 结 果
一个高度复杂的基因组DNA模拟群落
由来自 227 个细菌菌株的基因组 DNA 组成,这些菌株属于8 个门(Actinobacteria, Bacteroidetes,Deinococcus-Thermus, Firmicutes,Fusobacteria,Planctomycetes, Proteobacteria和Verrucomicrobia),18 类,47目,85科,175属,197种。
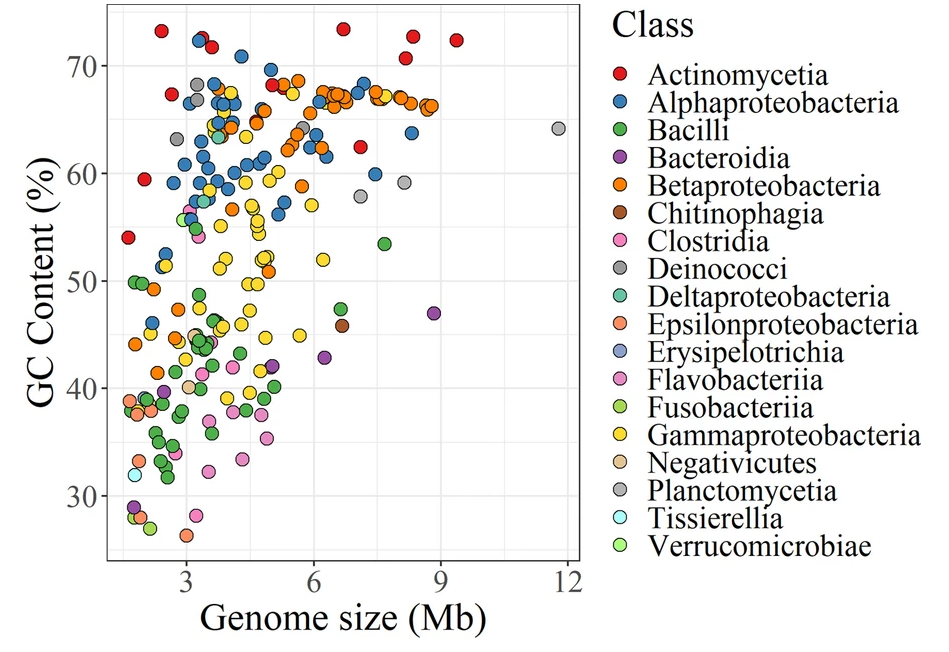
编辑
上图为模拟群落中的细菌菌株的基因组大小和GC含量(从26.3%到73.4%)散点图;
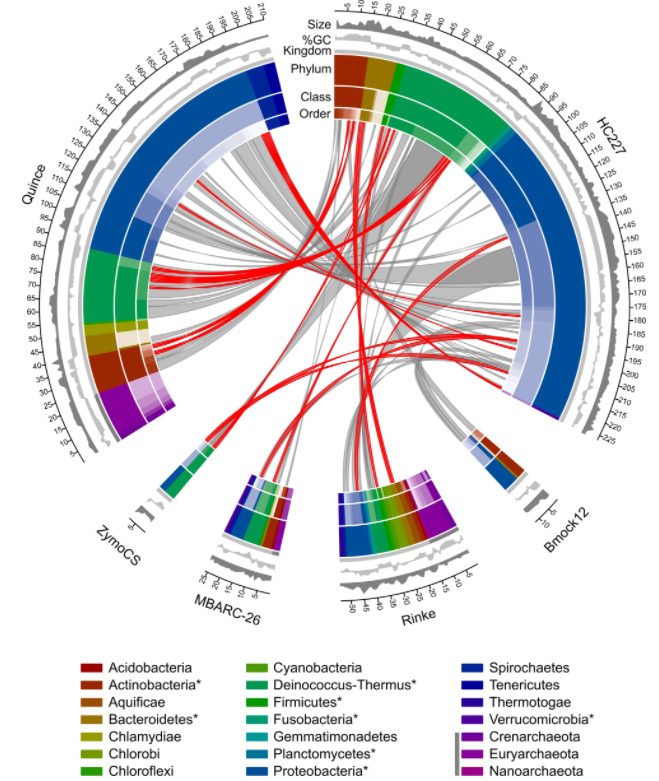
编辑
图为训练集与测试集中物种之间的关系图。红色线条表示在训练集中存在的菌种,灰色线条表示在训练集中存在的菌属。环状图中的不同颜色代表不同分类水平。图例中存在于训练集中的菌门用*标记,存在于古生菌的菌门用深灰色色带标记。
CheckM中基于单拷贝标记基因(SCMG)来评估 bin 质量的存在的缺陷
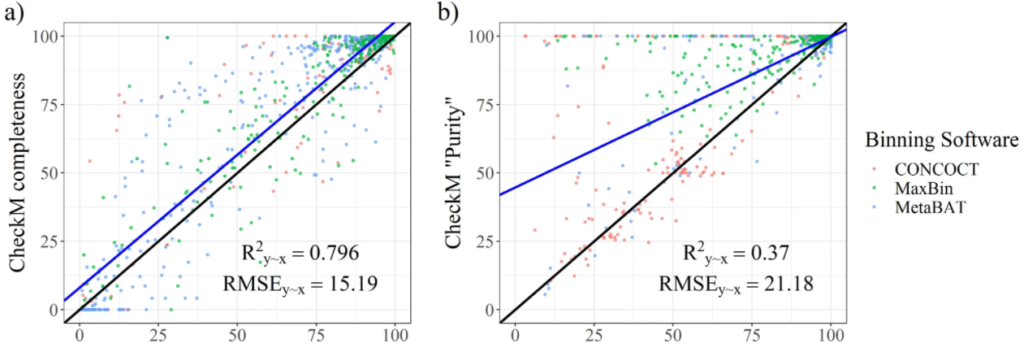
图a和b分别为从CheckM中输出的完整性指标和污染度。使用R^2y∼x(解释方差的百分比),RMSE(相对于实际值的均方根误差)两个参数评估结果。结果表示CheckM高估了bin的质量。许多受污染的bins被预测为接近未受污染。
使用MAGISTA分析模拟群落中的bins
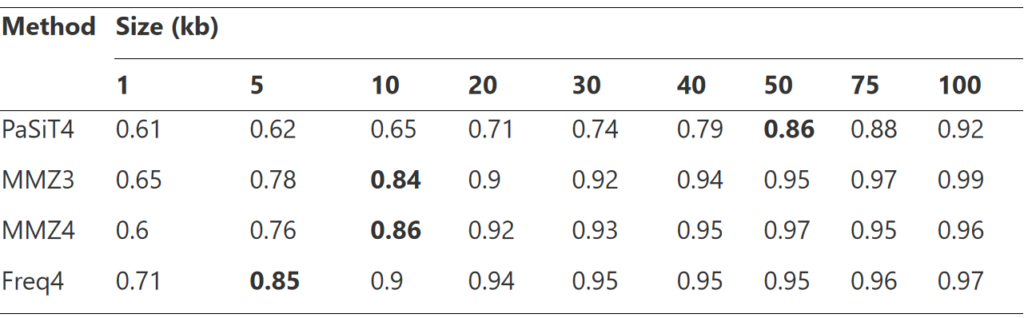
首先选择最佳片段大小用于计算距离分布,如上图所示,考虑了 1、5、10、20、30、40、50、75 和 100 kb 的片段,最终选择了粗体所示的片段大小。
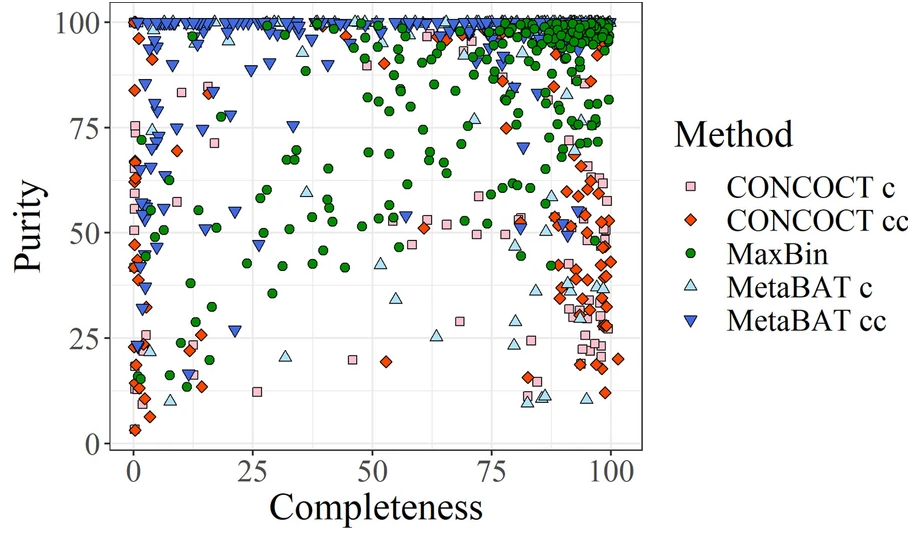
图为concont、MetaBAT和MaxBin产生的bins的完整性和污染度信息。
由于通过模拟生成这样的数据集并不能准确地表示真实的结果,所以使用了binning软件的结果,提供了一组不同质量的真实的bins。训练数据集的完整性和未污染度均在90%以上。
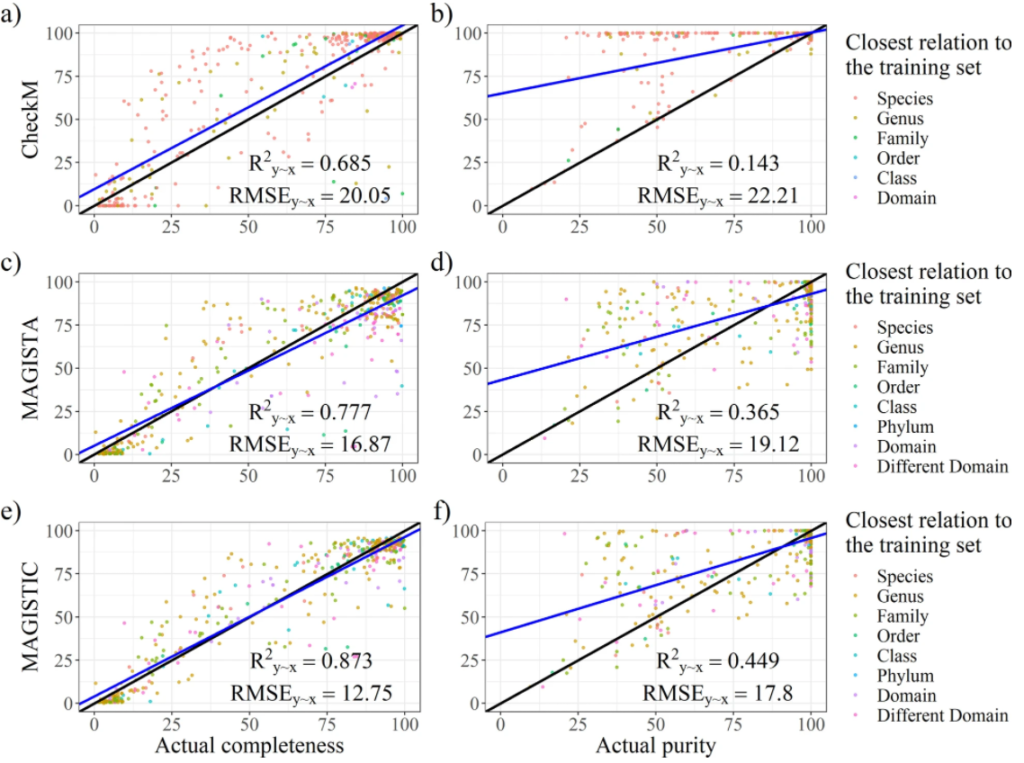
最后是模型构建,建立完整性和污染度的预测模型。并进行了模型评估,如图所示。分别对CheckM、MAGISTA 和 MAGISTIC测试了其性能。CheckM是现在主流的一款评估bin质量的工具。MAGISTIC是一款结合了CheckM和MAGISTA 的工具。使用解释方差的分数(R2y∼x)和均方根误差(RMSE)作为评估性能的指标。对于完整性的预测,MAGISTA 优于 CheckM。对于污染度的预测,MAGISTA 的表现优于 CheckM。
结 论
研究人员开发了一种新的用于预测高度复杂的宏基因组组装基因组bin的质量的方法,MAGISTA。是基于 SCMG 的低复杂性宏基因组方法的一个同样好的替代方法。除了MAGISTA之外,还通过结合CheckM的结果,使用MAGISTIC生成了一个更准确的预测。
研究人员在文章中指出MAGISTA 和 CheckM 都没有达到足够的准确度来被认为是可靠的。MAGISTIC 产生了比 MAGISTA 更好的结果。
在附加分析中,将测试集分为了两个子集,从真实和模拟reads中获得的bins,对此再进行分析,结果表示,CheckM 对于“真实”子集表现良好(但相比MAGISTA 和 MAGISTIC还是较差),对于“模拟”子集部分表现较差。而MAGISTIC相比MAGISTA会更准确些。但是文章中并没有详细说明MAGISTIC的工作流程。
查看作者在github上公开的软件说明,地址如下。但是没有说明和给出输出文件的内容。个人认为还不太成熟。
https://github.com/LM-UGent/MAGISTA
参考文献:
Goussarov G, Claesen J, Mysara M, Cleenwerck I, Leys N, Vandamme P, Van Houdt R. Accurate prediction of metagenome-assembled genome completeness by MAGISTA, a random forest model built on alignment-free intra-bin statistics. Environ Microbiome. 2022 Mar 5;17(1):9. doi: 10.1186/s40793-022-00403-7. PMID: 35248155; PMCID: PMC8898458.
谷禾健康

最近的Nature 和 Nature Medicine 连发表了好几篇关于肠道菌群的文章,包括肠道菌群与神经互作,和基于这个原理的针对自闭症的临床治疗方案。心血管疾病的微生物组和代谢特征等。
今天我们主要介绍心血管疾病中冠状动脉疾病的相关重要研究发现和意义。
复杂的疾病,如冠状动脉疾病(CAD),往往是多因素的,由多种潜在的病理机制引起。尽管冠状动脉疾病在预防、诊断和治疗方面取得了巨大进展,但仍然是世界范围内发病率和死亡率的主要原因。目前对冠状动脉疾病的治疗基于传统的和可控制的冠状动脉疾病风险因素,只能取得部分成功。
冠状动脉疾病的发展包括血管壁上动脉粥样硬化斑块的逐渐生长,这通常与代谢状态受损有关。人体接触环境分子的主要部位是胃肠道,其中膳食成分被微生物群转化,利用产生代谢物传播到全身器官。
血液充当体内分子的液体输送器, 特别是数以千计的循环代谢小分子,它们可以帮助我们了解体内生物过程状况,并且是研究冠状动脉疾病多因素性质疾病的宝贵来源。肠道微生物组积极参与血液代谢物的代谢。
几种肠道微生物群衍生的循环代谢物与心血管疾病相关:
三甲胺 N-氧化物
三甲胺 N-氧化物被确定为人类心血管疾病的标志物,进一步的证据表明在小鼠模型中具有促动脉粥样硬化性和促血栓形成。
硫酸吲哚酚
硫酸吲哚酚在细菌色氨酸酶降解色氨酸后在肝脏中产生,并被证明与动脉僵硬和外周血管疾病有关。
对甲酚
对甲酚是苯丙氨酸和酪氨酸的结肠细菌发酵产物,显示与心血管事件增加相关。
近期,以色列科学家招募了下列人群,采集其粪便和血清样本进行了全面的多组学分析,同时调查详细的医疗、生活方式和营养问卷等。
通过对粪便样本宏基因组测序(每个样本1000万 reads,约3G/样本)和对血清样本的进行非靶向质谱LC-MS测量了 961 种代谢物的水平,包括脂质、氨基酸、异生物质、碳水化合物、肽、核苷酸和大约 30% 的未命名化合物。
通过 Nightingale Health 的质子核磁共振 ( 1 H-NMR) 平台测量了另外 228 种血浆代谢物和比率,并使用了一个独立宏基因组数据集MetaCardis进行验证(该数据集样本来自于北欧血统队列,在地里区域上与该研究样本来源不同,这样可以分析遗传,饮食差异变量)。
MetaCardis数据集主要由四个主要群体组成:缺血性心脏病、健康对照组、代谢匹配的对照组和未经治疗的代谢受损对照组(详细数据集描述可以参看原文)
一、ACS的肠道微生物组特征
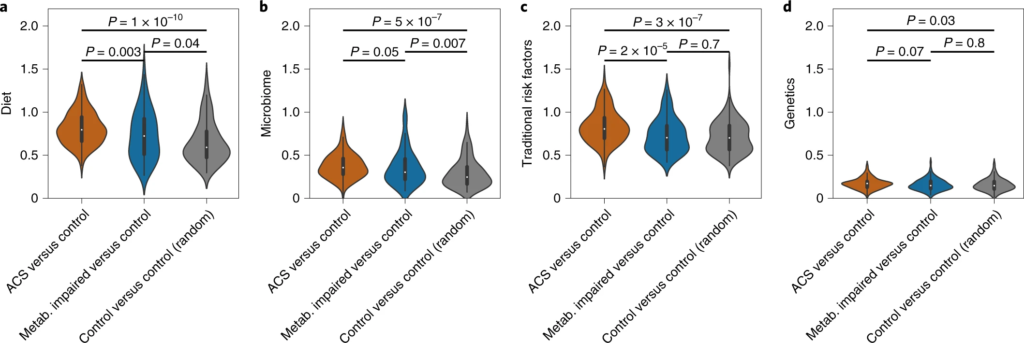
1. ACS 患者的变形杆菌丰度更高
这与之前的大多数研究结果一致,变形菌增多会导致处于炎症状态,是生态失调的标志。
20个在 ACS 或对照个体中显着富集的细菌,包括产丁酸盐的细菌如:梭菌属(Clostridium)、Anaerostipes hadrus嗜热链球菌(Streptococcus thermophilus)和Blautia菌属,以及Odoribacter splanchnicus 和大肠杆菌。
2. ACS患者队列中一种梭菌科的细菌物种 SGB 4712缺乏
在20 个显着富集的基因组中,鉴定到了一种以前未知的梭菌科细菌物种,索引为 SGB 4712。为了进一步验证该结果稳定和实用性,使用另外一个来自北欧血统地理上分布不同的队列,MetaCardis宏基因组数据集进行验证,与该研究结果一致,该物种的相对丰度随着具有 CAD 传统风险因素的种群逐渐减少。
3. SGB 4712关联15种显著差异的代谢物,其中包括降低心血管疾病风险的独立标志物——麦角硫因(ergothioneine,天然氨基酸)
对照组相比, 鉴定到SGB 4712 菌种与15 种循环代谢物的水平显着相关,在 MetaCardis 研究中,所有 15 种代谢物与 SGB 4712 的相关系数均可以重复,其中 10 种相关性仍然显著。
值得注意的是,SGB 4712与麦角硫因呈正相关,麦角硫因是一种天然存在的氨基酸,在体外显示对细胞应激源具有抗氧化和细胞保护能力,最近被证明是降低心血管疾病和人类死亡率风险的独立标志物。
此外,SGB 4712 与七种化学结构未知的化合物有关。其中包括 X-11315 和 X-24473,预测它们来自饮食,并与 SGB 4712 呈正相关。
图一 ACS 的微生物组和血清代谢组学特征
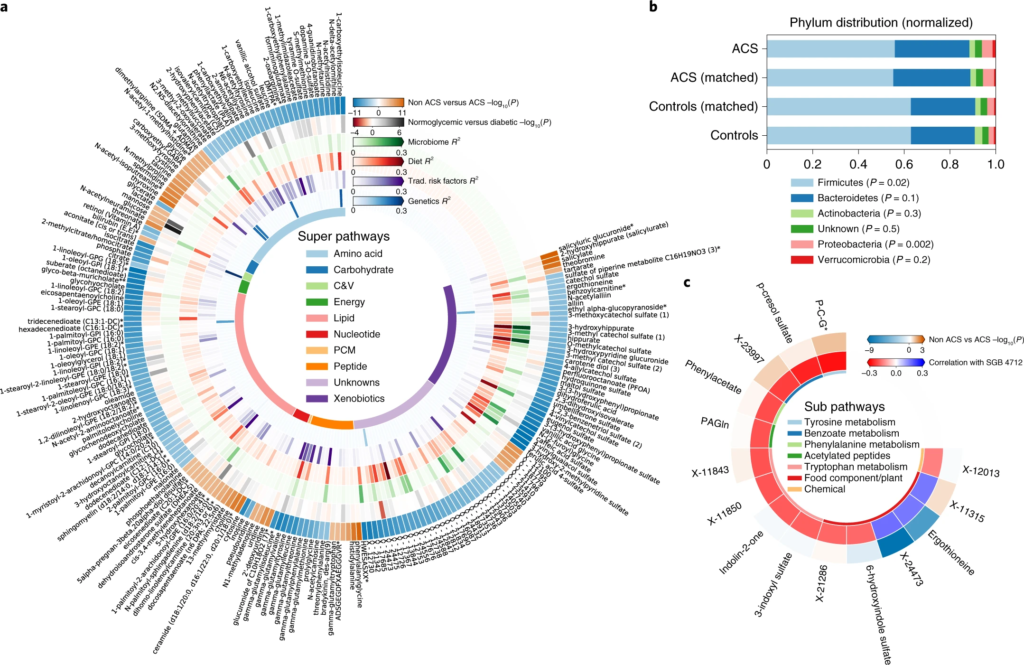
圆形热图显示 ACS 和非 ACS 对照组之间显着差异的前 200 种代谢物,与年龄、性别、BMI、吸烟状况和 DM 相匹配(方法)。每个切片代表一个代谢物,其名称显示在图表的外层周围。
这些结果突出了SGB 4712菌种在 CAD 发展中具有潜在的保护作用,由一系列循环血液代谢物介导,其中一些以前被证明在元生物途径中发挥核心作用,而另一些则未知。
因此,在实验研究中进一步验证后,这些代谢物可能会形成降低 CAD 风险的新目标。
二、ACS 的代谢特征因人而异
1. ACS 患者的血清代谢物水平个体化差异较大
虽然 CAD 患者具有共同的内表型,但他们通常表现出生物学上不同的疾病特征。为了更好地了解 ACS 的个体水平变异性,作者试图检查与非 ACS 对照的代谢偏差,并询问它们是否是个体特异性的。
计算了他们的个体偏差,并根据之前根据饮食、微生物组、传统风险因素和遗传学估计的 EV 对每个个体的前 100 个偏差代谢物进行加权。最后发现ACS 患者与其匹配对照的代谢偏差是因人而异的。
急性冠脉综合征患者的血清谱在血清代谢物水平上表现出广泛的扰动,包括533种显著改变的代谢物。
ACS的血清代谢组遵循一种主要的消耗模式,因为在对照组参与者中,358种代谢物(67%)的平均测量值较高。然而,这一趋势在主要的生物途径中并不一致。但是,与富含 ACS 的代谢物相比,饮食和微生物组在与 ACS 耗尽代谢物的偏差相关联方面更为显着(双尾 Mann–Whitney U-检验,P-value小于10 -20),这表明微生物组对 CAD 起保护作用。
值得注意的是,超过 90% 的显着扰动的代谢物无法用血糖状态来解释,这表明这种变化背后还有其他机制。所以进一步分析了其他系列综合因素(包括宿主遗传学、微生物组和饮食),得到一个重要发现就是:饮食和微生物组可以更好地解释 ACS 缺乏或含量低的代谢物,而传统的风险因素可以更好地解释 ACS 富集的代谢物。
图2 代谢偏差由潜在决定因素解释,并与临床参数相关
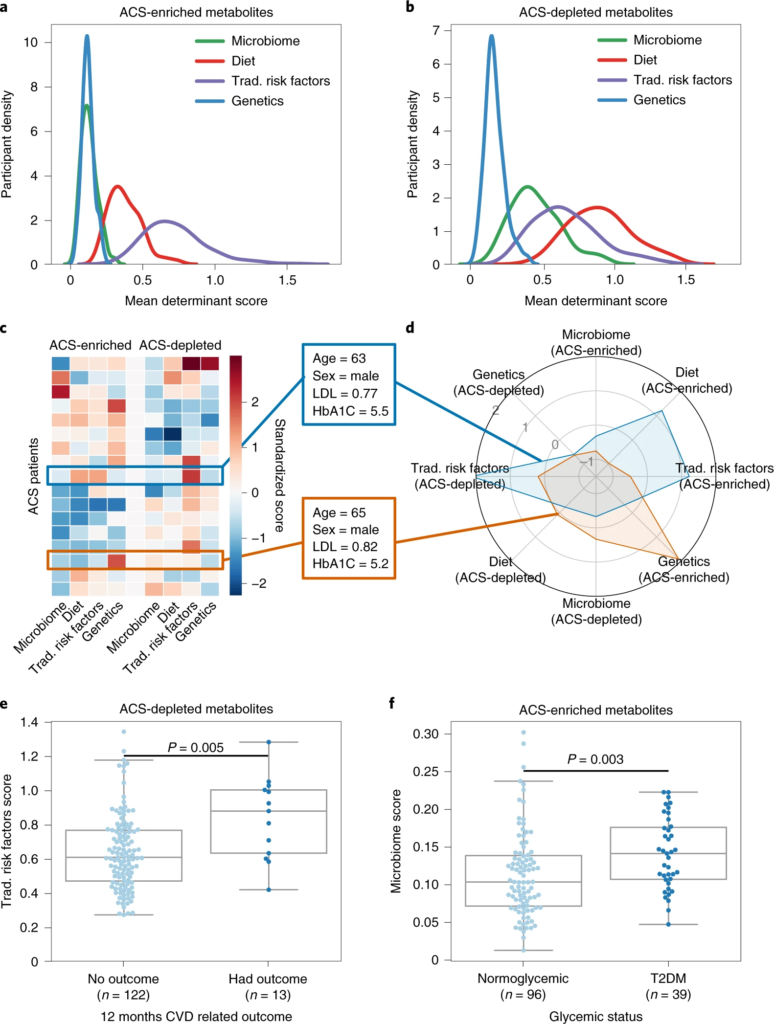
a、b、密度图显示 ACS 参与者的分布(y轴)与代谢物的潜在决定因素(微生物组、饮食、传统风险因素或遗传学)的平均加权R 2 ( x轴);富含 ACS 的代谢物。
2. 相似的临床特征,但其动脉粥样硬化负担的代谢机制却不同
虽然一些患者可能具有相似的临床特征,但他们的潜在生理状态和疾病轨迹可能不同。为了强调这种 CAD 患者的变异性,作者选择了 ACS 患者的常规危险因素的同质亚组。其中包括 17 名 60 至 70 岁的男性患者,低密度脂蛋白 (LDL) 在 0.70–1.30 mg ml -1范围内,糖化血红蛋白 (HbA1C) 低于 6%。尽管具有相似的临床特征,但该 ACS 患者亚组在代谢偏差方面表现出异质性。
三、微生物组在CAD早期阶段发挥作用
动脉粥样硬化是一种经过多年发展的进行性疾病,其中动脉粥样硬化斑块形成的每个阶段的特点是不同的病理过程。在早期阶段,血管壁上的动脉粥样硬化斑块的生长通常与代谢状态的损害有关。
为了解释每个代谢成分在 CAD 发展的时间轴上的参与,作者将个体代谢偏差的分析应用于代谢受损的对照(定义为 T2DM、高血压或血脂异常的诊断,或 BMI > 35),以及到非 ACS 个体的随机子集。
在比较这三组的分数时,我们发现分数分布存在一致的差异。与微生物组和饮食相关的代谢异常呈现出渐进的趋势,与对照组的随机子集相比,代谢受损的对照参与者的代谢物存在显着偏差。
这表明,微生物组和饮食对ACS的贡献可能是通过受损的代谢状态介导的,而不是代谢受损个体中尚未表现出的与传统风险因素和遗传学相关的代谢物异常。
a – d,归因于饮食 ( a )、微生物组 ( b )、传统风险因素 ( c ) 和遗传学 ( d ) 的代谢偏差分数计算三个亚组:(1) ACS 个体 ( n = 135) 与非 ACS 对照与年龄、性别和 BMI 相匹配(橙色);(2) 患有代谢障碍的非 ACS 对照(定义为:诊断为 T2DM、高血压或血脂异常,或 BMI > 35;n = 102)与其他年龄、性别和 BMI 匹配的非 ACS 对照(蓝色);(3) 一组随机的非 ACS 个体 ( n = 132) 与其他匹配年龄、性别和 BMI(灰色)的非 ACS 对照。
四、血清代谢组学预测ACS患者 BMI 更高
肥胖是 CAD 的主要独立危险因素,影响已知的危险因素,如血脂异常、高血压、葡萄糖耐受不良和炎症状态,以及可能尚未认识到的机制。BMI 测量被用作肥胖的标志和代谢健康的指标。
为了研究肥胖作为 CAD 的独立危险因素,该研究设计并彻底验证了基于血清代谢组学的 BMI 模型,并表明较高的预测 ΔBMI 对应于更广泛的动脉粥样硬化疾病。
作者分析了CAD 患者的 BMI-代谢组平衡是否以及如何被破坏。使用了梯度提升决策树 (GBDT) 算法预测 BMI,结果表明在非ACS受试者中发现的代谢组-BMI模式在ACS患者中受到干扰。
为了研究这些扰动,作者测试了对照组和 ACS 测试集中预测和测量 BMI 之间的差异,这里称为 ΔBMI。结果发现,与非 ACS 受试者相比,该研究的模型预测 ACS 的 ΔBMI 更高。
为了验证这些结果的稳健性,作者试图根据其他类型的代谢组学数据和独立队列来复制这些发现。将相同的预测程序应用于基于 NMR 的代谢组学数据,并观察到ACS 和对照之间 ΔBMI 的更大差异,应用于为发表的MetaCardis 队列数据中得出在所有 BMI 范围内,与血糖正常的缺血性心脏病患者相比,患有糖尿病的缺血性心脏病患者的 ΔBMI 显着更高。
进一步分析推断哪些特定代谢物是 ACS 患者高 ΔBMI 的主要驱动因素,发现两种脂质在对照组中与 BMI 呈负相关,后者在患有更广泛疾病的患者中也显着减少,这两种脂质分别是:
1-(1-enyl-palmitoyl)-2-oleoyl-GPC (P-16:0/18:1)
1-(1-enyl-palmitoyl)-2-linoleoyl-GPC (P-16:0/18:2)
最近的研究表明,脂质1-linoleoyl-GPC (18:2) 与肥胖和 T2DM呈负相关,并且脂质水平的增加显着降低了T2DM的风险。该研究发现 1-linoleoyl-GPC (18:2) 和 1-(1-enyl-palmitoyl)-2-linoleoyl-GPC (P-16:0/18:2) 在对照组中与 BMI 呈负相关,并且在患有更广泛 CAD 的患者中显着耗尽,这表明这些代谢物可能作为降低 CAD 风险的潜在靶点。
此外,两种代谢物都含有一条亚油酸链,一种必需脂肪酸,与 T2DM 风险呈负相关。然而,这些假设应在干预性研究中进一步检验。
迄今为止,大多数研究都集中在寻找在 CAD 患者中增加的新代谢物,而该研究对 199 名 ACS 患者进行了全面的多组学分析结果强调, ACS 的代谢组学特征是缺乏多种血清代谢物,其中许多与饮食和微生物组有关。
其中一个重要的发现是以前未知的细菌物种 SGB 4712,它在 ACS 患者和独立验证队列中都显着缺乏或偏低。通过进一步将这种细菌与心脏毒性和心脏保护代谢物的水平联系起来,证明了特定细菌基因组的缺失可能与 CAD 风险增加相对应,并提出在后续干预研究中评估的具体目标。总体而言,这些发现因此为 CAD 患者的预测甚至治疗提供了一种新方法。
迄今为止,大多数研究都对 CAD 患者进行了批量分析,寻找人群水平的风险因素,而不是关注个体水平的生物变异性。在这项研究中,作者使用全面的代谢组学和微生物组分析,呈现了 CAD 内部变异性的深度映射。总之,结果揭示了新的范式和治疗方向。
参考文献:Talmor-Barkan Y, Bar N, Shaul AA, Shahaf N, Godneva A, Bussi Y, Lotan-Pompan M, Weinberger A, Shechter A, Chezar-Azerrad C, Arow Z, Hammer Y, Chechi K, Forslund SK, Fromentin S, Dumas ME, Ehrlich SD, Pedersen O, Kornowski R, Segal E. Metabolomic and microbiome profiling reveals personalized risk factors for coronary artery disease. Nat Med. 2022 Feb;28(2):295-302. doi: 10.1038/s41591-022-01686-6. Epub 2022 Feb 17. PMID: 35177859.

谷禾健康

微生物物种的遗传变异研究通常包括单核苷酸多态性(SNPs)、结构变异(structural variants ,SV)和可移动遗传元件(mobile genetic elements,MGEs)。
在宏基因组中,SNP被用来量化种群结构、追踪菌株和鉴定微生物表型的遗传决定因素。然而,现有的基于比对的宏基因组SNP检测方法需要高性能的计算和足够的覆盖深度来区分SNP和测序错误。
为了解决这些问题,美国加利福尼亚大学研究人员使用高质量基因组,构建了 909 个人类肠道物种中 1.04 亿个 SNPs 的目录,并使用针对该目录的独特 k-mers 表征来自 7,459 个样本的肠道菌群的全球种群结构,开发了GenoTyper for Prokaryotes(GT-Pro),可以对宏基因组的这些 SNPs进行快速基因分型的方法。该研究成果近日公开在《Nature Biotechnology》发表。
该方法与使用读长对齐的方法相比,GT-Pro 更准确,速度快两个数量级,作者构建了一个GT-Pro数据库,基于大约25,000个宏基因组样本,并展示了GT-Pro如何用于数千种菌群的菌株水平探索,可以实现在个人电脑上快速高效地对数百万个SNP进行宏基因组分型。

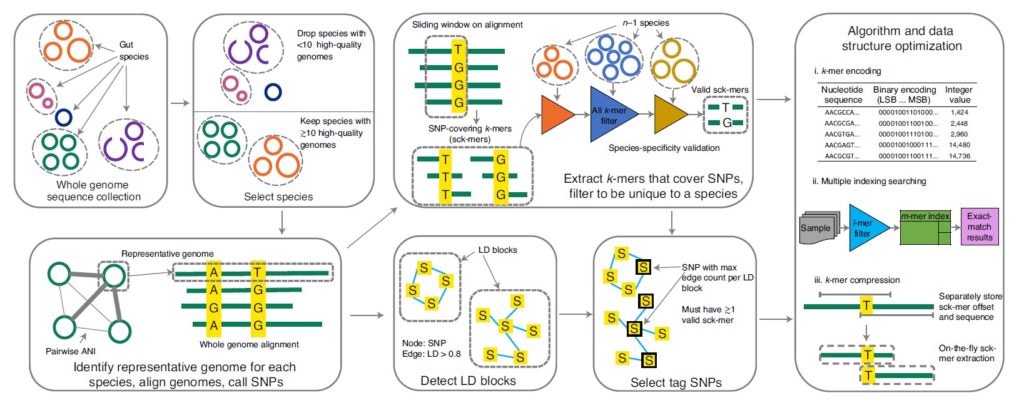
如图,按箭头方向所示。
首先从全基因组序列中识别高质量基因组的物种(去除<10 个高质量基因组的物种,高质量基因组:≥90% 的完整性和≤5% 的污染),对于每个物种,一个有代表性的基因组是根据平均核苷酸一致性(Average Nucleotide Identity,ANI)和组装质量指标选择的,确定代表性基因组后,对每个物种,通过MUMmer软件将每个同种基因组(conspecific genome )与代表性基因组比对,确定SNP,在这些SNP中选择常见的双等位基因SNP用于分型(site prevalence ≥90% and minor allele frequency >1%)。
接下来提取覆盖SNPs的k-mers(sck-mers),过滤出独有的物种,同时检测LD块,并选择具有物种特异性的sck-mers的SNPs和该块中其它SNP的最高LD。LD块为基于跨基因组的共现模式将 SNP 聚类成linkage disequilibrium block。检测LD块使用R2 阈值 (0.81) 。具有物种特异性的sck-mers即删除了两个或多个物种共有的任何sck-mer。
最右边的方框里简要是GT-Pro的算法和数据结构的优化方法。也是该研究的主要目标之一,正是利用了该方法构建的SNP索引才能实现快速地分型。
首先是k-mers编码,选择了k=31,以便使用64位整数编码,通过这一步骤,GT-Pro 数据库缩小了四倍。
其次是多索引检索和进一步压缩SNP数据结构。
优化后的GT-Pro数据库由两个表组成:
(1)10.6 GB的sck-mers表,包含每个k-mer的4字节条目;
(2)2.4 GB的sc-span表,包含每个等位基因的24字节条目。
所需的总存储空间为13 GB,是原始sck-mer表的bzip2压缩的两倍。也使得GT-Pro可以在个人计算机中高效运行。
1.从模拟宏基因组中准确识别SNP
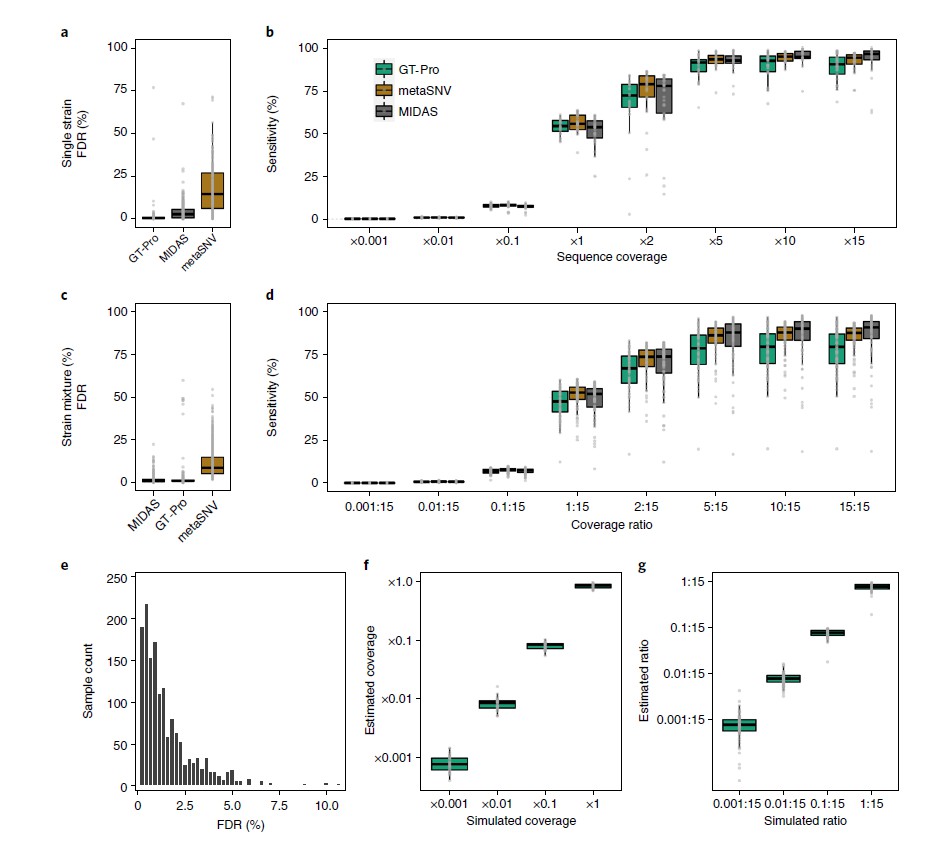
比较GT-Pro、MIDAS和metaSNV宏基因分型的准确性,使用232个未用于开发这些方法的人类肠道分离株的模拟宏基因组(大约2600万次reads)。
图a为FDR比较,假阳性指不正确的基因型,是由测序错误和读数映射到错误位点导致的。假阴性指缺失的基因型,在没有读数映射时产生。在宏基因组中,FDR最低的是GT-Pro(中位数,0.4%),而 metaSNV 最高(中位数,14.5%)。
图b为对图a的灵敏度调查,用于直接比较不同方法。敏感性是指在GT-Pro数据库中检测到分离株基因组(参考和非参考等位基因)中存在基因型的概率。结果表示,随着覆盖度的加深,GT-Pro的灵敏度损失较小。
图c为比较三个工具在一对同种分离株但不同覆盖率下的FDR,目的是检查宏基因组分型方法对菌株混合物的表现。其中一个菌株始终为15倍的覆盖率,另一个菌株的覆盖率从 0.001 到 15 倍不等。FDR包括纯合位点和杂合位点。
总体而言,GT-Pro的 FDR与 MIDAS 相似但低于 metaSNV。
图d为对图c的灵敏度调查,敏感性是指正确判断reads所模拟的基因组的基因型(纯合位点和杂合位点)的概率。GT-Pro 的灵敏度低于基于比对的方法,基于比对的宏基因分型通常使用覆盖率和等位基因频率过滤来减少错误的杂合性调用。
图e为基于图a中模拟的等位基因,从tag SNPs推算的基因型的FDR。结果表示大多数物种的 FDR 较高但仍低于 5%。
图f和图g,为了探索 GT-Pro 是否能用于定量估计物种丰度,使用从单个分离株和对同种分离株中模拟的宏基因组,比较了sck-mer匹配reads的平均数量和已知的基因组覆盖率。结果表示GT-Pro等位基因的调用和计数可以用一个小的校正因子来估计物种和菌株的相对丰度。
所有的结果表示,在模拟宏基因组的测试中,metaSNV 和 MIDAS 对于丰富的物种(>5×覆盖度)和保守位点表现良好,但 GT-Pro 对典型覆盖率值、非参考和杂合位点更准确和敏感,同时对错配和测序错误更为稳健。只是,与 metaSNV 和 MIDAS 相比,GT-Pro 无法检测其数据库中缺少的新 SNP。
结论是,在保守的基因组区域仔细选择sck-mers能使 GT-Pro 能对来自鸟枪法宏基因组数据的已知 SNPs 进行敏感和特异性的基因分型。
2.从模拟宏基因组中准确识别SNP
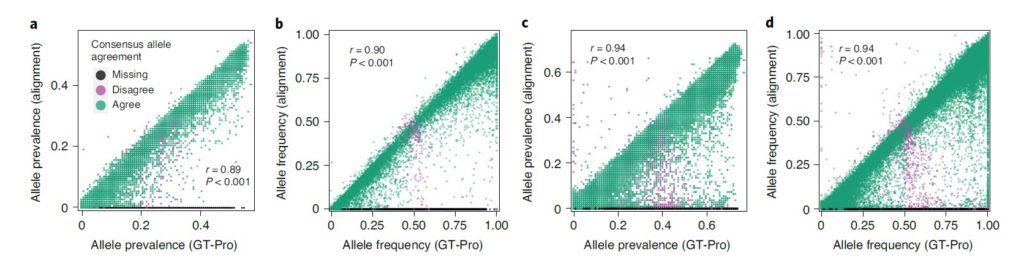
使用GT-Pro对肠道微生物组样本进行宏基因组分型,结果与基于比对的MIDAS宏基因组分型比较。
图a和b分别为流行率(prevalence)、平均等位基因频率(Average allele frequency)
图c和d类似图a和b,只是物种不同。
每个点代表一个 SNP,颜色表示两种方法的共有等位基因(即样品中最常见的)是否相同(绿色),两种方法都返回某些样品的基因型,但共有等位基因不同(紫色)或仅GT-Pro 返回基因型(黑色)。
结果表示对于高覆盖率物种,基于比对的方法能检测到GT-Pro数据库中没有的SNP,而GT-Pro 在中低覆盖率物种中检测到更多SNP位点。这部分结果也与模拟宏基因组测试时的结论一致。
1.使用GT-Pro的SNP估算结构变异
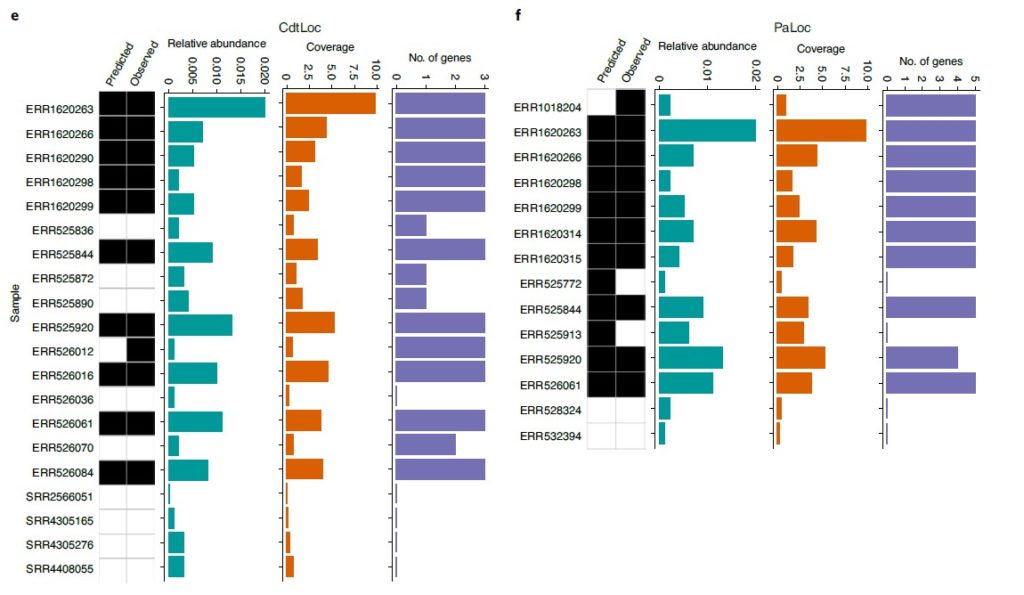
研究人员试图使用GT-Pro的SNP推断附近基因或操纵子的存在,从而作为结构变异的生物标志物。
首先对艰难梭菌的毒性控制位点CdtLoc和PaLoc的侧翼区域使用GT-Pro检索SNP。
接着用艰难梭菌的参考基因组训练了一个随机森林分类器,用于预测来自混合群组(n = 7,459)的人类肠道宏基因组中存在/不存在艰难梭菌毒素基因位点。
图e和f分别代表CdtLoc基因和PaLoc基因,对每个样本,最左边的热图,第一列为预测的,第二列为基于比对方法得到的,黑色表示存在,白色表示不存在。
从左到右的条形图分别指艰难梭菌的相对丰度、全基因组序列覆盖率,从毒素位点检测到的基因数目,所有这些都是通过比对到艰难梭菌的代表性基因组来估计的。结果表示预测到艰难梭菌毒素位点的概率>0.6。
对CdtLoc的几个预测与宿主的表型相关(P < 0.001),包括5名艰难梭菌阳性和CdtLoc(+)的克罗恩病患者,这与该人群对艰难梭菌病理的高易感性相一致。与此相反,CdtLoc基因座在大多数可检测到艰难梭菌的健康婴儿中没有被预测,这与婴儿期艰难梭菌常见的无症状定殖一致。这些结果表明,GT-Pro可以预测具有临床相关性的linked structural variants。
2. 使用 GT-Pro 捕获新的种内遗传结构
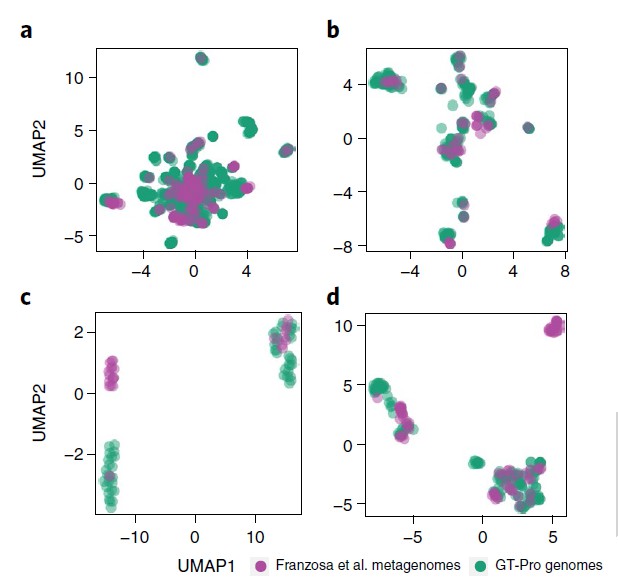
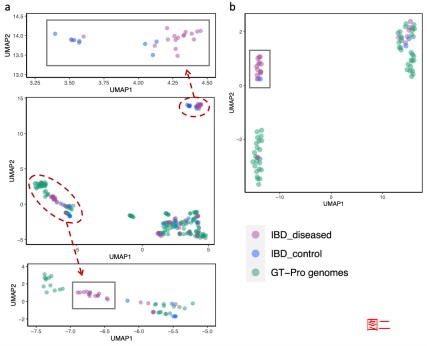
GT-Pro 可以对从参考基因组中鉴定的已知 SNP 进行宏基因组分型分析。但研究人员认为GT-Pro还有更广阔的发展,假设GT-Pro可以基于 SNP 等位基因的不同组合检测新的菌株变异。
为了验证该假设,研究人员使用GT-Pro 对最近发表的北美炎症性肠病 (IBD) 队列 的 220 个粪便宏基因组中发现的物种进行基因分型。使用UMAP降维分析,每个图都是将UMAP应用于一个物种GT-Pro SNPs基因型矩阵的结果。每个点代表该物种的一个菌株(杂合宏基因组的主等位基因)。紫色为队列样本,绿色为GT-Pro基因组。
结果表明 GT-Pro 的数据库代表了这些个体的常见菌株多样性,对于大多数物种,如图一的a和b,粪便样本组与参考基因组聚集在一起,相比之下,对于少数物种。
如图二的c和d,分别是新的亚种,观察到基因型与数据库中任何参考基因组不同的粪便样本群,包括一些富含IBD患者的样本。这说明可以使用 GT-Pro 常见 SNP 发现新的亚种遗传结构。
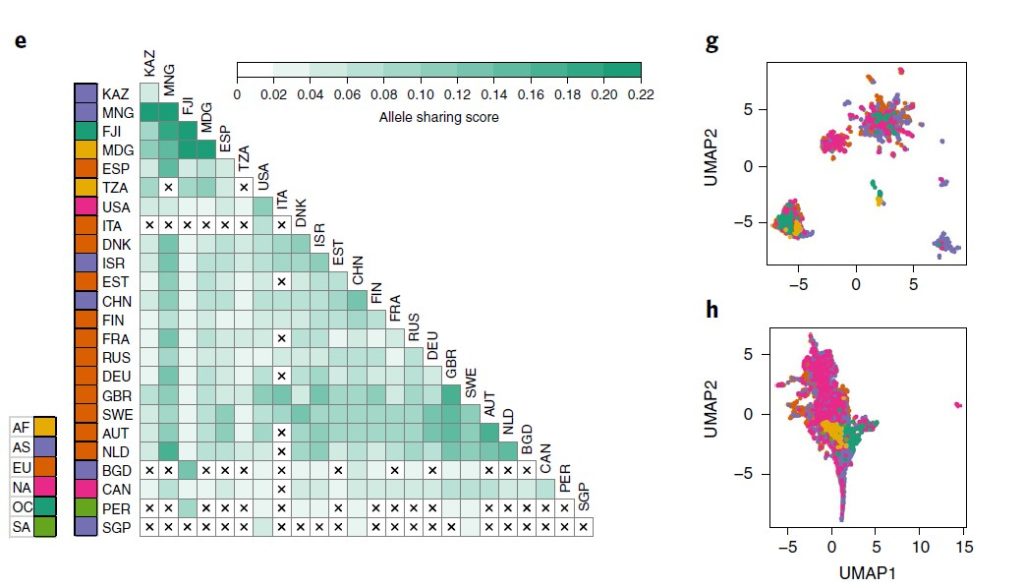
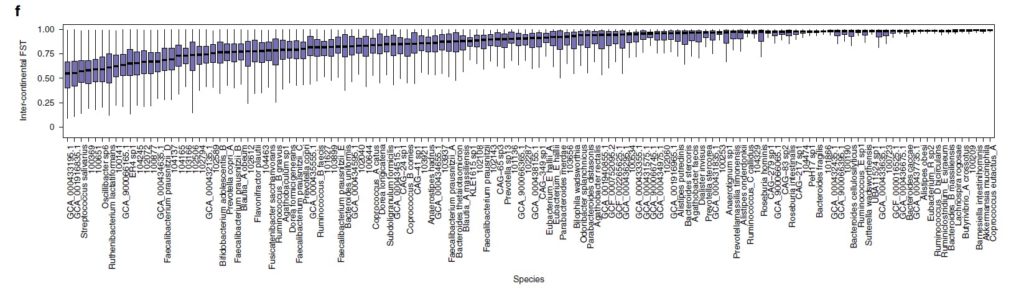
3. GT-Pro 探索全球人类肠道微生物组遗传变异
来自六大洲 31 个地点的 7,459 个肠道样本中发现的 881 个物种的 5180 万个 SNP的多个物种的种内遗传变异荟萃分析。
图e来自不同国家的宏基因组间的等位基因平均共享分数的热图。打叉单元格表示由于样本对不足(<5,000)而导致分数缺失。
图f为78 个常见物种的洲际种群分化分析(大陆内部与大陆之间的遗传相似性,用 F 统计检验测量亚种群 (FST) 中捕获的总遗传变异的比例)。
每个箱线图代表一个物种的洲际 FST 分布,按中位数排序。图g为通过直肠Agathobacter rectalis(物种ID 102492)的GT-Pro宏基因组基因型中的种内遗传变异捕获的地理模式的示例。
图h为为基于图g中相同样本的物种相对丰度的UMAP 分析。每个点都是一个宏基因组样本。颜色与图e示意一致。
结果表示,等位基因共享与工业化程度以及宿主关系明显关联;洲际种群间的分化程度有巨大差异,具有高FST的物种显示出明显的宿主集群,但不是所有宿主集群都与地理相关。
这与菌株在宿主中殖民的生活方式和环境的作用相一致。相比之下,在基于物种相对丰度的UMAP分析中,宿主间并没有明显集群,这表明宏基因组基因型可能揭示了在丰度分析中缺失的微生物生态学和微生物群落-宿主关系。
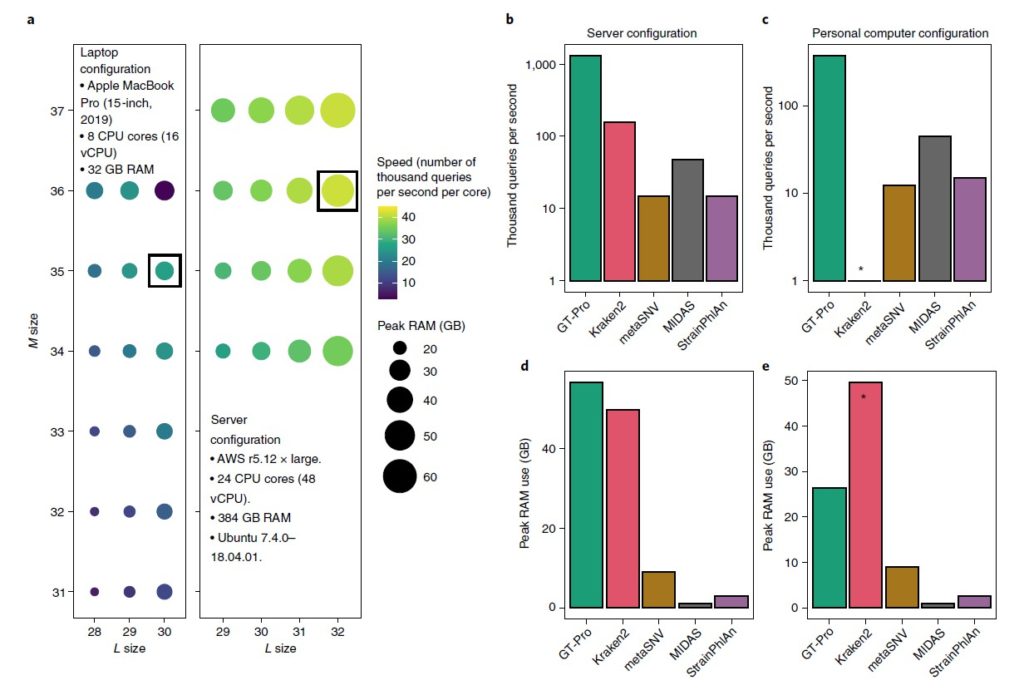
图a评估GT-Pro在笔记本电脑(左)和服务器环境(右)中的计算性能,以bits为单位。颜色表示处理速度,圆圈大小为RAM使用峰值。黑色方框表示最优状况。
图b-c为GT-Pro与metaSNV、MIDAS、StrainPhlAn、 Kraken2之间的速度比较,分别在服务器环境和笔记本电脑下比较。
图d-e为RAM使用峰值的比较,分别在服务器环境和笔记本电脑下比较。* 由于超出可用 RAM,Kraken2 无法在笔记本电脑中运行。
这些分析表明,与其他方法相比,GT-Pro 在服务器上大约快 8.5-570 倍,在笔记本电脑上快 8.3-163.6 倍。平均而言,处理每个宏基因组只需要在服务器上不到 4 秒,在笔记本电脑上大约需要 13 秒(平均为 497 万次读取)。虽然 GT-Pro 比其他方法更快,但它在服务器上需要 1.1-53.7 倍的 RAM和笔记本电脑上的 2.9-29.2 倍的 RAM(不包括内存不足的 Kraken2)。因此,只要计算机具有足够的 RAM,GT-Pro 数据结构和算法就可以极大地加速宏基因组分型。
研究人员在该文章中使用GT-Pro大约分析了2.5万个宏基因组,展示了GT-Pro是如何快速准确的识别SNP以及探索结构变异、种内遗传变异等。GT-Pro不使用基于比对的方法,而是类似于Kraken2,通过编码k-mers来快速检索,并适用于个人计算机或服务器环境。
但是它也不是完美的,目前GT-Pro存在的不足和如何应对:
第一,GT-Pro 数据库并未捕获所有人类肠道微生物多样性:但是通过基因组测序,会持续扩大SNPs的数量和涵盖的物种。
第二,GT-Pro 类似于基因分型阵列,因此不能识别新的 SNP,这需要其他方法,例如基于比对的宏基因组分型或单细胞基因组测序。
第三,由于基因组集合中存在高度相关的物种,少数物种缺乏物种特异性的 sck-mers。替代策略,例如使用更长的 k-mer 或不太常见的 SNP,可以对这些物种使用 GT-Pro 。
第四,尽管非常严格的挑选了用于构建GT-Pro的基因组和SNPs,但不可能完全排除错误(例如,不完整、污染和物种错误分类)。
最后,GT-Pro 不直接对结构变异进行基因分型。
“
考虑几个GT-Pro的未来发展方向,比如:
将GT-Pro与下游算法结合起来,以识别代表新微生物菌株的SNPs簇,或准确标记参考数据库中已知菌株的SNPs;
将GT-Pro的计算框架扩展到其他微生物环境中;为短插入缺失和结构变异开发无比对宏基因组分型;
将微生物组应用于精准医学,综合识别与疾病或其他特征(如致病性、抗菌耐药性、药物降解)相关的SNPs;
将GT-Pro用于检测污染、重组和跟踪变化,比如变异或菌株随时间、宿主生活方式和地理位置的变化。
主要参考文献
Shi ZJ, Dimitrov B, Zhao C, Nayfach S, Pollard KS. Fast and accurate metagenotyping of the human gut microbiome with GT-Pro. Nat Biotechnol. 2021 Dec 23. doi: 10.1038/s41587-021-01102-3. Epub ahead of print. PMID: 34949778.

谷禾健康
随着科学技术的巨大进步,产生了大量的“组学”数据。理解生物系统各个层次产生的大量序列和结构数据是关键,由此产生了“生物信息学”。
“生物信息学”是一个跨学科领域,主要是用计算算法来组装、评估、理解、可视化和归档与生物分子相关的数据。
从基因组测序、基因及其功能预测到蛋白质分析,如蛋白质结构和功能预测、系统发育研究、药物和疫苗设计、生物体鉴定,以及支持和推进生物技术领域的研究,都需要用到生物信息学。
今天给大家推荐一本书《Advances in Bioinformatics》,帮助广大科研工作者更容易进行研究,从而对生物学有新的见解。

该书共23个章节。涵盖了蛋白质组学、代谢组学、DNA测序和NGS技术、基因组分析、生物计算、神经网络分析、大数据分析、软计算、人工智能到进化生物学、疫苗和药物设计、生物合成学和癌症生物学应用等,从这些领域出发,综述了生物信息学在其中的应用、发展、帮助和已经获得的成果,并探讨了未来的发展方向。
1. 生物信息学简介及其应用
生物信息学在DNA测序与分析、基因组测序及其注释分析、进化生物学的计算、比较基因组学、基因和蛋白表达分析、蛋白质和DNA、RNA的结构分析、免疫信息和药物设计的技术应用。
2. 生物信息学工具和软件
重点介绍了Banqit、Spin、WEBIN、Sequin、Sakura等序列提交软件;ADIT、PDB_Extract等分子结构提交软件;SRS、Entrez、Getentry等序列检索工具。此外,还详细讨论了BLAST、CLUSTALW/X等序列比对工具,以及Swiss-Model、Modeller、JPred、3D-Jigsaw和ModBase等结构预测工具。
3. 生物信息学在生物科学中起到的作用
重点介绍了生物信息学在基因组学、转录组学、蛋白质组学和代谢组学等主要“组学”领域的作用,以及在其它领域,营养基因组学、化学信息学、分子系统发生学、系统学和合成生物学的应用。还讨论了生物信息学在这些领域的多样化发展。
4. 蛋白质分析:从序列到结构
介绍与蛋白质序列和结构分析相关的各种数据库和方法。这类研究的主要应用之一是在药物发现和开发方面。
5. 进化生物学
进化是一个物种或种群经历遗传特征变化的动态过程。对进化的研究被称为进化生物学。进化生物学研究的关键是序列变异,这是通过比较DNA或蛋白质序列来检测的。迄今以及开发了不同的计算工具来比对所获得的序列和识别序列变异。进化基因组学的应用正在从研究人类进化到研究各种病毒的进化。许多病毒对人类健康具有严重威胁。本章介绍了PAML、PhyML、MrBayes、RAxML、MSA、MUSCLE 、MAFFT 等用于系统发育分析的计算工具。并详细讨论了进化的计算原理。
6. 基于web页面的调控序列分析的生物信息学方法
本章主要概述了在线分析哺乳动物基因组中调控序列方法,以及用于调控序列分析的在线生物信息学工具。
7. 用于SNP分析的生物信息学资源综述
基因变异是导致生物物种内多样性的关键。单核苷酸多态性(SNPs)是遗传变异的主要形式。单核苷酸多态性在理解生物表型差异的进化过程中至关重要,而且还被用于各种疾病的诊断和治疗。本章详细介绍了用于人类和其他非人类基因组的SNP分析。此外,还讨论了在生物信息学领域需要解决的挑战和差距,以便在未来有效地研究SNPs。
谷禾健康
2020年,深度学习算法AlphaFold2在从原始序列预测蛋白质三维结构方面取得了里程碑式的成果。
宏基因组学产生的大量测序数据,让人们得已窥见未经培养的微生物的生物合成潜力。与初级代谢途径相比,参与次级代谢的酶往往催化不同底物的特殊反应,这些途径为发现新的酶学提供了丰富的资源。
到目前为止,从环境DNA(eDNA)研究中发现新的酶或功能大多数是通过PCR筛选或基于活性位点的筛选方法获得的。作为另一种选择,鸟枪法宏基因组学也具有从eDNA中直接发现新酶的能力,还可以避免由于PCR或活性导向的功能宏基因组学工作流程引入的共同偏差。
最近发表的一篇长综述,为宏基因组学在酶学领域构建了一张宏伟蓝图。文章中比较了发现酶的方法,包括系统发育学、序列相似性网络、机器学习技术等。也讨论了各种实验策略来测试计算预测,包括异源表达和筛选。
除了这些广泛使用的方法,还补充了一些新兴技术如宏组学、单细胞基因组学、无细胞表达系统等方法及建议。这里,我们沿着作者给出的路线,为大家做个导读。
首先作者在文章中明确指出两点,文章中主要关注天然产生的酶,而不包括通过工程或定向进化策略获取的非自然酶。其次是生物合成基因簇(bgc)中编码的细菌酶,因为这些酶是天然产物中研究最广泛的。
另一点宏基因组DNA序列与从微生物分离物中获得的基因组DNA没有本质上的区别。两者都是来自生物系统的核苷酸序列。从结构上讲,宏基因组样本中的BGCs与分离物参考基因组中的BGCs基本上没有区别,除了有时由于组装过程中引入的相邻边界和错误而更碎片化。一些宏基因组BGCs甚至在可培养生物的基因组中具有同源簇。
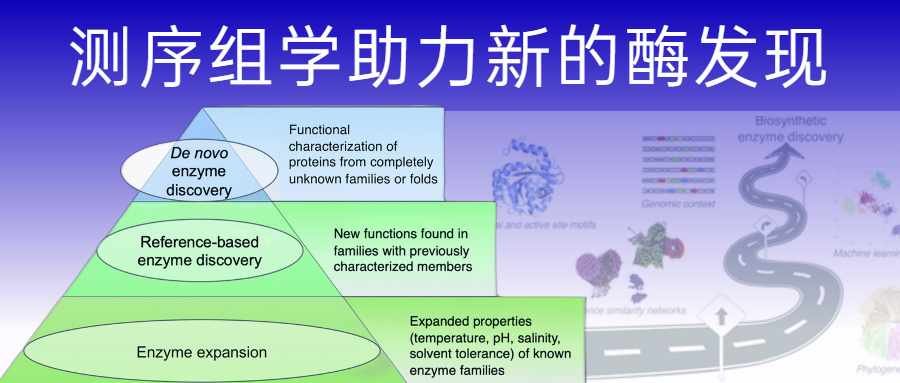
如下图,使用三层金字塔说明,越往下说明这个类别在宏基因组酶研究中数量更多。
Robinson S Let al., Nat. Prod. Rep., 2021
第一层
即金字塔尖端,指的是识别出全新类型的生物催化剂,也就是说这类酶必须属于没有任何功能特征成员的蛋白质折叠或家族。到目前为止,大多数新发现的酶的例子都来自可培养的细菌和真菌,而不是eDNA和未培养的微生物。也因此,在宏基因组中识别出的蛋白质家族中还存在极大的探索空间。
第二层
指的是基于参考发现的酶,是在已发现的蛋白质家族中对新的反应类型的表征。
第三层
代表了宏基因组酶研究中占比最大的一部分,指发现了具有不同底物种类的酶,或具有不同反应条件的酶,包括温度、pH、盐度或溶剂偏好。
在发现酶的方法中,将鸟枪法宏基因组学测序与功能宏基因组学(活性导向分离和基于PCR方法)之间进行比较。

Robinson S Let al., Nat. Prod. Rep., 2021
活性导向分离方法筛选功能宏基因组文库是宏基因组领域最早发展起来的方法之一,方法核心是鉴定出所需表型的克隆,例如从fosmid、cosmid或人工染色体文库克隆。由于该工作流程不依靠序列同源性,因此对从头发现新酶特别有效。
基于PCR方法的筛选核心是简并引物以扩增编码感兴趣的蛋白质结构域的eDNA基因。基于扩增的常见的生物合成标记物的分析已经被广泛地应用于检测新的BGCs和天然产物。例如,一类全新的钙依赖性抗生素,苹果酸,是通过基于PCR的土壤亚基因组腺苷酸结构域筛选检测到的。
鸟枪法宏基因组学是指直接的、非靶向的eDNA测序。由于不需要PCR扩增和大肠杆菌等文库宿主,所以在鸟枪法测序过程中引入的偏差较少。产生测序数据的速度比构建宏基因组fosmid或cosmid文库快得多。其最大的挑战是从复杂环境样本中足够数量和质量的eDNA和足够的测序深度来检测和纠正个别读数中的错误。关于检测稀有生物的BGCs,可以使用Samplix技术。
这一小节重点介绍了发现酶的三种方法,虽然各有参差,但是殊途同归,依靠这些技术新的酶不断被发现。不容忽视的是应用于鸟枪法宏基因组测序数据的生物信息算法和技术的进步为酶的发现提供了新的途径。但是参与天然产物生物合成的酶是如何帮助从宏基因组数据集中获得要点,以提高我们对未培养微生物的次级代谢功能的认识呢?作者提出一个问题,“是否存在发现酶的温床?”
鸟枪法宏基因组测序完成后,就需要执行下游生物信息分析,使数据可公开存取使用,例如JGI IMG/M、iMicrobe或MGnify这些站点,整合了大量的基因组数据,可以分析可以存储。
这里作者特别介绍了MGnify,MGnify的制作作者强调它是为了“搜索微生物暗物质”而开发的。MGnify的一个好处是能够使用HMMs查询宏基因组,而不是使用基本的基于序列比对的搜索方法,如BLAST或DIAMOND。
虽然这两种方法都是有效且快速的方法,但HMMs对于鉴定更遥远的同源基因特别有用。
(MGnify:https://www.ebi.ac.uk/metagenomics/)
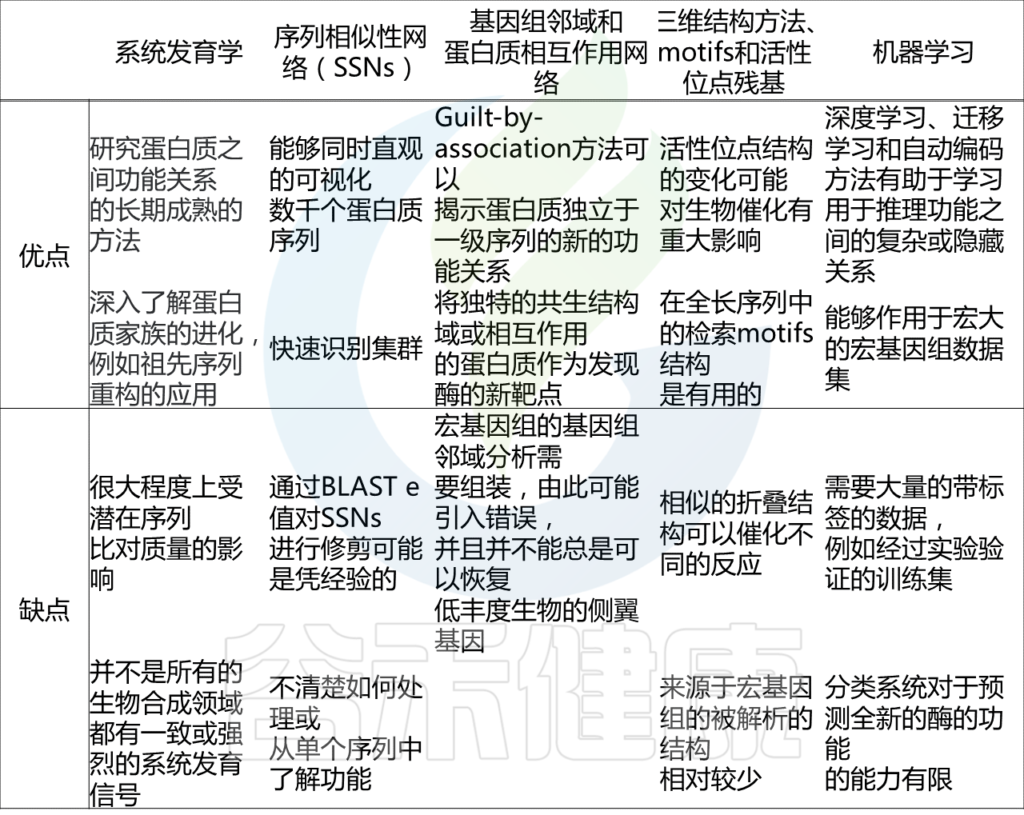
预测蛋白质家族中新的酶功能的计算方法之间的比较
Robinson S Let al., Nat. Prod. Rep., 2021
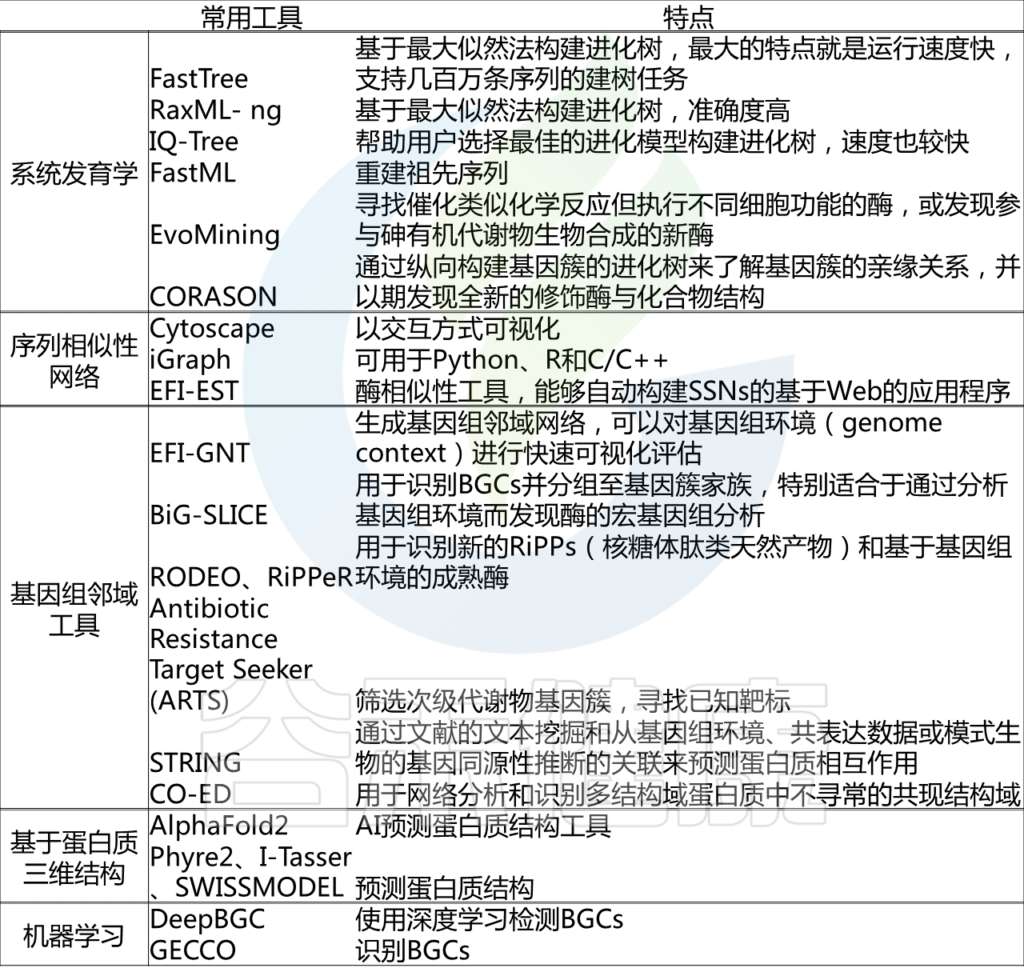
基于以上的计算方法,整理了作者在文章中列举的一些常用工具:
Robinson S Let al., Nat. Prod. Rep., 2021
无论是用鸟枪法还是功能筛选的宏基因组学发现酶,最后都需要对酶进行表征。
1 质量控制
当选择蛋白质在实验室中进行鉴定时,重要的第一步是质量控制,以去除可能存在测序错误或不能编码全功能蛋白质的嵌合体和截断序列(truncated sequences)。可以根据相似性对蛋白质进行聚类,并自动选择有代表性的序列,比如CD-HIT和UCLUST工具。
根据数据集的大小,可能需要进一步的过滤步骤。
最明显的策略之一是选择在可培养生物体中也存在的宏基因组序列,因为这可以在原生宿主中进行功能表征。其次是从嗜热生物体中选择蛋白质,这些蛋白质往往编码热稳定性更高的酶。还有选择更稳定和表达更好的蛋白质,包括过滤不具有高GC含量、跨膜区或无序区的蛋白质。
作者建议使用多种标准来对需要实验鉴定的蛋白质序列进行排序,通过这种方式,预测工具中的个体偏差可能会被基于集成的方法部分抵消,以确定最有希望的能够表征发现的酶的蛋白质。
2 蛋白异源表达
一旦识别了感兴趣的酶或BGCs,必须设计异源表达的构建。不幸的是,大多数用于功能宏基因组学方法的宏基因组文库准备的载体通常不适用于异源表达。由于Fosmid/Cosmid载体的最大插入大小为45 kb,许多完整的BGCs也没能完全被捕获到宏基因组文库中。
除了经典的限制性内切酶克隆和Gibson组装方法外,人们还开发了新的方法来提高将大型BGCs克隆到异源宿主的效率和方便性。
一种流行的方法是转化偶联重组技术(TAR),它利用酵母中的同源重组系统将土壤和海绵宏基因组中重叠的eDNA cosmid/fosmid克隆拼接在一起。
3 酶活性的筛选
当感兴趣的酶被表达出来后,就要对它们进行体内或体外的活性分析。酶筛选方法通常在通量(throughput)和通用性(generalizability)之间进行权衡,如下图:FACS(流式细胞荧光分选技术)、NIMS(纳米结构启动质谱技术)、SAMDI-MS[ 结合无细胞蛋白质合成和自组装单层解吸电离(SAMDI)质谱技术]、Microfluidics(微流控技术)、mRNA display(通过体外核糖体翻译,有效地将肽链到自己编码的RNA)。
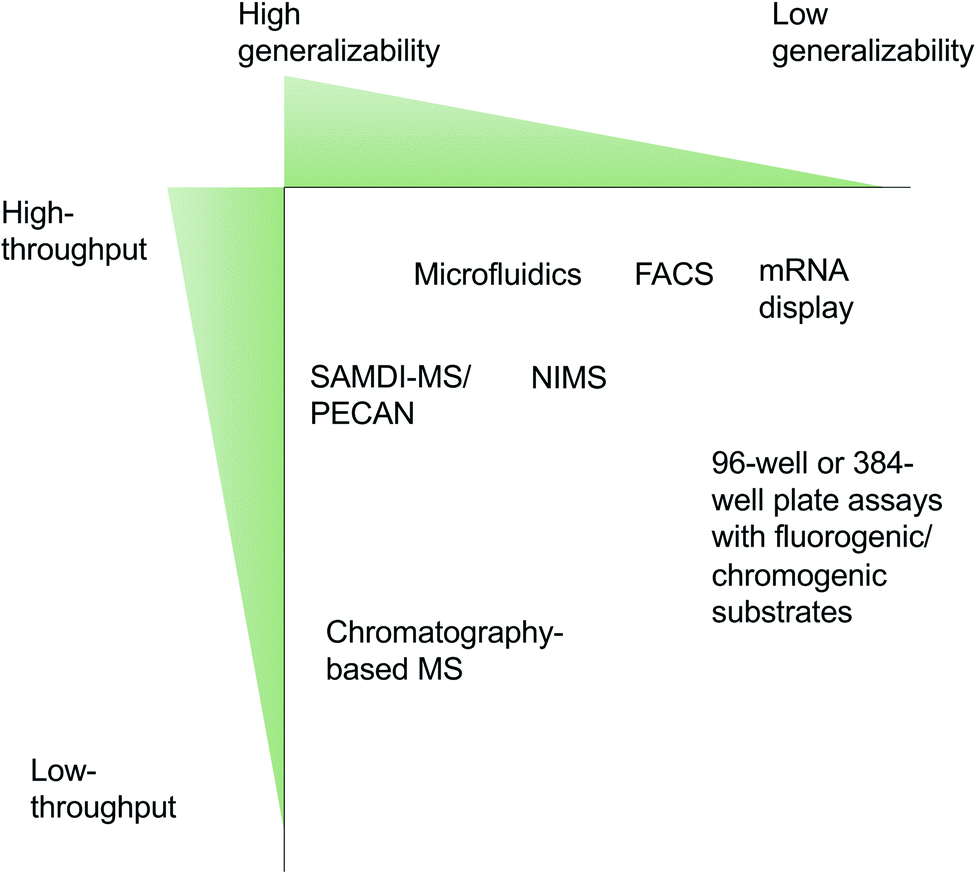
Robinson S Let al., Nat. Prod. Rep., 2021
对该领域的未来提供一个展望,着重于新兴技术与宏基因组学工作流程相结合,以加速酶的发现。
1 宏组学
将各种宏组学技术(包括宏转录组学、宏蛋白质组学和代谢组学)整合到酶发现工作流程中,可以成为一个强大的框架,将基因型与表型联系起来,以产生假说。例如用RNA-Seq分析了一种未知的钼依赖酶DADH在人体肠道中参与多巴胺分解代谢的过程;一项堆肥微生物群落的宏转录组分析结果发现了糖苷水解酶家族中的一个异常酶,这个酶带有exo-1,4-b-xylanase活性等。不同的多组学数据集的整合为酶的发现提供了新途径。
2 单细胞基因组学
单细胞基因组学依赖于微生物细胞的分选,通常采用微流控技术或流式细胞仪(FACS)的方法,然后用高保真聚合酶裂解和全基因组多重置换扩增(MDA)。单细胞基因组学并不依靠于相似细胞的种群是无性繁殖的假设。
因此,单细胞基因组学研究揭示了从海洋浮游植物到癌细胞的各种系统中显著的种群内基因组变异和进化。这一新兴的研究领域需要进一步应用单细胞和空间转录组方法,以更好地了解微生物群落结构和微环境如何影响生物合成基因的表达。
3 微流控
基于微流控的分选方法已被广泛应用于定向进化和蛋白质工程研究,但很少用于挖掘基因组引导酶的发现。最近的一项研究使用光学镊子和微流控技术,根据单个细胞的拉曼光谱对复杂的微生物群落进行分类,这在下游单细胞测序或培养工作中有许多应用。通过对分选的细胞进行下游单细胞测序,活的单个细胞的化学表型可以直接与它们的基因型联系在一起。只是,微流控技术在从宏基因组中发现新的生物合成酶方面的应用目前还没有广泛使用。
4 无细胞系统
无细胞系统为所需DNA序列的快速转录和翻译创造条件,而不受维持细胞生长的限制。与体内表达系统不同,无细胞平台还允许产生有毒的代谢物,这些代谢物通常会杀死异种宿主。为了进一步提高产量,包括mRNA display、MALDI-MS和液滴微流控等筛选方法已经与无细胞平台相结合。对于一些生物合成途径,DNA模板在短短几个小时内就能产生高产量。
5 与序列无关的方法
文中描述的绝大多数技术都依靠基于序列或基于结构的同源性来推断蛋白质功能。然而,当预测“未知的未知因素”时,这些方法往往达不到预期,即重新发现与一个或多个特征蛋白家族没有序列或结构相似性的酶。与序列或结构无关的方法在天然产物研究中也很少使用,因为大多数识别BGCs的计算方法都依靠与常见生物合成结构域的同源性。
decRiPPter是一种基因组挖掘工具,用于检测新的RiPPs和BGCs。decRiPPter算法的核心过滤步骤是使用泛基因组比较来检测分布在分类群内的操纵子,这些操纵子可能参与了次级代谢功能,而不是初级代谢功能。Krousterman等人用DecRiPter分析了1295个链霉菌基因组,鉴定了一个新的RIPP成熟酶家族,催化一种新的肽类天然产物的脱水和环化反应。
1 新的发现往往发生在蛋白质家族的近邻
虽然这不是一个普遍规律,但与已知功能的参考蛋白相比,序列同源性低的蛋白质比序列同源性高的酶更容易适应不同的底物,并催化出新的反应类型。
2 跳出比色测定法的框框,进入未知的蛋白质空间
对2014年1月至2017年3月发现的宏基因组酶进行的荟萃分析发现,>84%属于脂肪酶/酯酶或纤维素酶/半纤维素酶类别。同样,>82%是通过基于活性的筛选发现的。显然,目前的宏基因组筛选方法偏向于工业相关的酶类,这些酶类也可以用标准比色法检测出来。
3 不再局限于大肠杆菌,寻找新的宿主
一项对照研究发现,一般环境细菌中只有30-40%的基因可以在大肠杆菌中表达,只有7%的高GC含量的DNA可以在大肠杆菌中表达。在功能宏基因组学方面,假单胞菌、链霉菌、红球菌、芽孢杆菌甚至古生菌已经被用作文库宿主和具有穿梭载体的多宿主表达系统(multi-host expression systems)。同样,非传统的异源表达宿主(如亚硝型分枝杆菌)已经被开发用于从宏基因组BGCs发现新的酶。
相关阅读:
ResistoXplorer——基于Web的耐药基因组数据可视化,统计和探索新分析工具
参考文献:
Robinson S L, Piel J, Sunagawa S. A roadmap for metagenomic enzyme discovery[J]. Natural Product Reports, 2021.
E. J. Culp, N. Waglechner, W. Wang, A. A. Fiebig-Comyn,Y.-P. Hsu, K. Koteva, D. Sychantha, B. K. Coombes,M. S. Van Nieuwenhze, Y. V. Brun and G. D. Wright,Nature, 2020, 578, 582–587
N. S´elem-Mojica, C. Aguilar, K. Guti´errez-Garc´ıa,C. E. Mart´ınez-Guerrero and F. Barona-G´omez, Microb.Genomics, 2019, 5, 445270
M. G. Chevrette, K. Guti´errez-Garc´ıa, N. Selem-Mojica,C. Aguilar-Mart´ınez, A. Yanez-Olvera, H. E. Ramos- ˜Aboites, P. A. Hoskisson and F. Barona-G´omez, Nat. Prod.Rep., 2020, 37, 566–599.
谷禾健康

导语
微生物在在地球上无处不在,适应了几乎所有可用的生态栖息。 微生物在不同物种和个体之间差异性很大,存在着广泛的微生物多样性。
野生动物之所以能够耐受病原菌的感染和有毒食物的威胁以及抵御多种疾病,可能与其体内或体表生存的微生物密切关联。然而,与已被广泛研究的人类微生物群相比,野生动物的微生物群受到的关注较少。
当宿主有着共同的饮食或共同祖先,尤其是哺乳动物,通常肠道菌群构成也更为相似,不过这种相关性在鱼类、两栖类、鸟类和非脊椎动物中较弱。在许多情况下,肠道微生物都参与宿主的关键生理过程,包括代谢特殊的饮食化合物。
近日,以色列魏茨曼科学研究院Eran Segal团队采用一致的方法从全球四大洲采集了406份动物粪便样品,包括121份养殖样品和285份野生样品。共涉及184个动物物种,包括哺乳类、禽类、两栖类、硬骨鱼类等的物种。这些物种在分类单元、觅食/取食行为、地理分布、性状等方面具有较高的多样性。
使用宏基因组学来分析这180多个物种的肠道菌群,使用从头基因组组装,构建并在功能上注释了5000多个基因组的数据库,其中包括1209种细菌,但是其中75%未知。
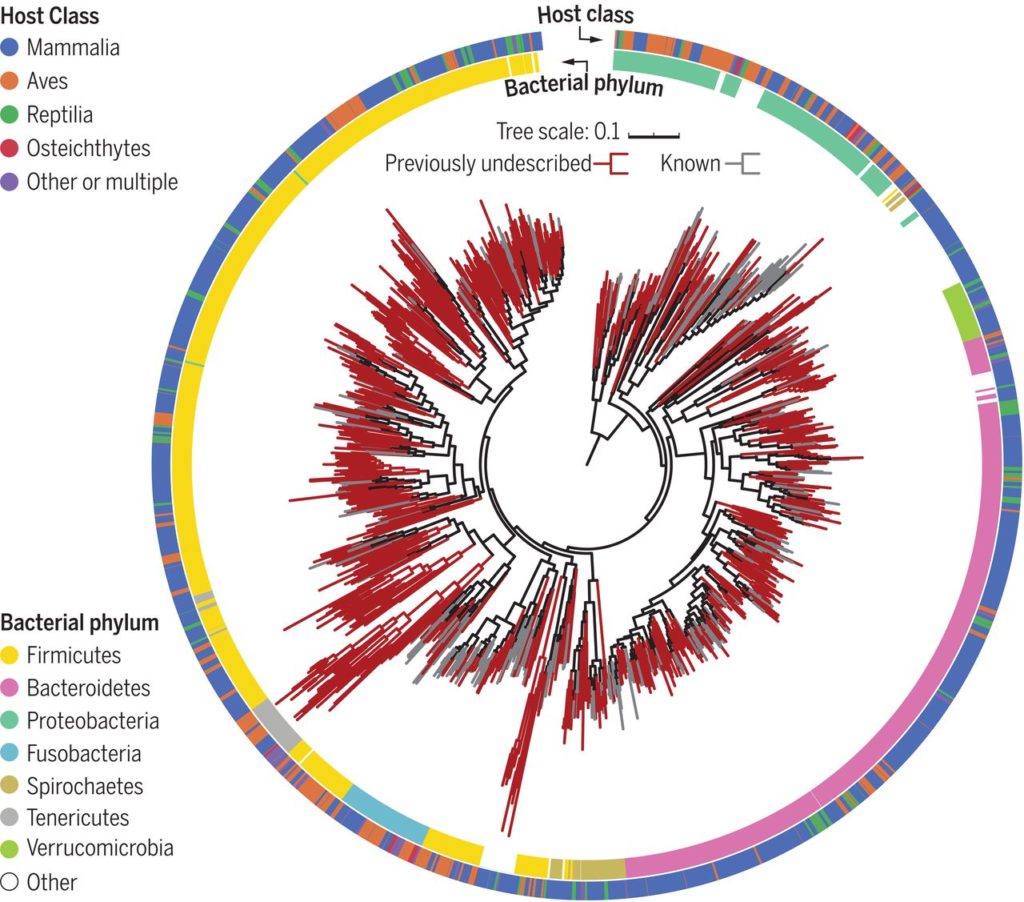
在这项研究中组装的1209个基因组的最大似然比的系统发育树。
内有色环和外有色环分别表示细菌门和宿主类别。先前未描述的基因组进化枝为深红色。
一,坚持野外采样。野外采样,尤其对于动物采样存在很多挑战,但是证据表明圈养动物会改变微生物组,而且,过往大量有关哺乳动物的研究大部分来来自于是圈养动物,包括大小鼠。这次大规模全球野外采样,可以扩大和了解动物宿主栖息微生物的机会。
二,要获得广泛的野生动物代表性,需要在全球不同的生态环境采样,并从具有不同特征和喂养方式的多种动物中取样。此外,该研究为每个物种手工制定了特质,包括饮食适应性,活动时间和社会结构,使我们能够系统地研究微生物群组成与宿主表型之间的关系。
三,为未知物种的大规模注释细菌基因组数据库,并确定了与这些动物的性状和分类相关的多种微生物模式,并强调了其潜力作为发现新的工业酶和治疗剂的主要未开发资源。
微生物的组成,多样性和功能含量与动物分类,饮食,活动,社会结构和寿命相关。动物微生物群系是生物功能的丰富来源,可能会对生物技术产生影响,包括抗生素,工业酶和免疫调节剂。
此外,野外动物表现出适应性,例如安全食用腐烂,感染病原体的肉类和有毒植物,可以产生强效毒素、生物发光以及各种疾病和微生物病原体具有特异性免疫力、再生能力并且在某些物种中具有极长的寿命。这些适应性中的某些,例如毒素产生和生物发光至少部分是由生活在动物体内/上的微生物共生体赋予的。该研究构建和功能注释从自然栖息地的野生动物中提取的微生物群的综合数据库可以对动物性状与其微生物群之间联系的进行全面了解。
例如,为了证明在动物微生物群中可以发现新的细菌功能,作者在实验中验证了细菌毒素——食用腐肉的欧亚兀鹫(Gyps fulvus)的MAG中发现的代谢蛋白酶。这些蛋白酶可用作抗菌化合物,具有抗菌活性潜在的应用包括对抗人类食物中毒。
欧亚兀鹫(学名:Gyps fulvus):体长95-105厘米,尾长24-29 厘米,翼展240-280厘米,体重6-11千克。是一种大型的褐色鹫。栖息在海拔高达2,500米的范围内。主要以山羊、鹿和瞪羚等野生动物,以及人类养殖的绵羊、山羊、牛和马为食。靠灵敏的嗅觉来找寻腐烂的动物尸体,并常常为抢一块肉而争个不停。而且习惯把头伸进动物尸体的腹腔内,啄食内脏和肌肉。分布范围非常广泛,遍布欧洲、中东和北非,也分布于印度、喜马拉雅山脉。在地中海沿岸国家最常见。
这项研究的最大贡献是其丰富的、系统生成的数据集。很容易想象,微生物保护和新出现的抗生素耐药性等不同领域的突破是由这些亚基因组的发现推动的。在欧亚兀鹫微生物群中发现的蛋白酶证明了从野生动物微生物群中进行生物勘探的原理,尽管尚不清楚该案例研究是否应被视为例外或预期的发现。


新发现的食腐肉的欧亚兀鹫(Gyps fulvus)的肠道细菌中的毒素代谢蛋白酶可能在抵抗食物中毒方面有应用。
这项研究仅仅触及了可以用这个数据集检验的假设的表面。未来方向包括:
01 微生物群如何帮助动物降解有毒的植物化学物质。
02 抵御食物中的病原体。
03 从多种食物来源中提取营养的问题。
四,丰富了许多未知物种的细菌门,并发现某些细菌进化枝相对于同一门中的其他细菌具有独特的功能特性。动物种类和已发现的动物种类(共存细菌的特定簇)之间的细菌状况有所不同。动物中这种未被探索的微生物多样性与被充分研究的人类微生物组形成了对比,而人类微生物组在参考数据库中表现得更好。未描述的物种中的富集度最高的是疣状菌属(Verrucomicrobia),这是一个存在于水、土壤和人类肠道中的门,但培养物种相对较少。
确定了多种途径和直系同源物,这些途径和直系同源物在特定的动物性状中显着丰富,并表明功能性景观与这些性状相关。这些功能中的一些功能提示了野生生物微生物群的新角色和特性。绘制野生动物的微生物群落图也可能有助于野生微生物的保护工作。
五,重述了首次通过扩增子测序发现的结果,包括食草动物微生物组比食肉动物微生物组更加多样化。这是一个比较重要的发现,是否说明植物性饮食可以提高微生物多样性?此外,不同动物群体的微生物组编码的遗传途径因宿主饮食、体型和其他特征而不同。
六,野生动物的微生物区系也是动物和人类病原体的天然库,如当前的COVID-19大流行一样,通过对野生生物微生物景观的广泛的基因组集合可以阐明其传播到人群中的时间和途径。
一,使用MAGs(宏基因组组装的基因组)限制了研究其中一些目标的拓展,即保护医学和生态上重要的细菌菌株。有些微生物物种将需要分析未组装的读取、培养或富集技术,而不是本研究中使用的全基因组测序。
二,即使是那些基因组被MAG组装捕获的细菌,这些组装体也倾向于排除“辅助”基因组,即在不同菌株间存在的基因。然而,这些基因往往在适应特定的宿主和环境中发挥作用。例如,抗菌素抗性、致病性和能量收集是经常由移动元件或其他辅助基因编码的性状。
三,Levin等人检测到的大多数非特征微生物在宿主体内的活动,以及它们是稳定地定殖在动物体内还是短暂地通过它们的胃肠道的问题仍然存在。在这个庞大的数据集上测试每个假设需要大量的计算、解释和实验验证。
参考文献:
Levin D, Raab N, Pinto Y, et al. Diversity and functional landscapes in the microbiota of animals in the wild[J]. Science, 2021, 372(6539).
Coleman M. Diagnosing nutritional stress in the oceans[J]. Science, 2021, 372(6539): 239-240.
L.-X. Chen, K. Anantharaman, A. Shaiber, A. M. Eren, J. F. Banfield, Genome Res. 30, 315 (2020)
E. C. Lindsay, N. B. Metcalfe, M. S. Llewellyn, J. Anim. Ecol. 89, 2415 (2020)