-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康

在过去的几十年里,生物技术和计算技术的巨大进步彻底改变了我们对微生物群落的理解。特别是,基于16S rRNA和宏基因组测序的研究进一步验证了一个事实,即微生物群不是静态的实体,而是动态的生态系统,身在其中的组成成员之间,以及成员与环境之间,都在以各种方式进行相互作用。
在这些研究过程中产生了大量的关于微生物相互作用的机制和环境依赖性的数据,也出现了许多专门储存单一类型或进行了部分分类整合的数据库。但由于是以不同的标准去整理的数据,所以很难共享。
//
本文研究人员提出了一套体系,用于微生物组数据管理,简称FAIR (findable, accessible, interoperable, and reusable),即可查询、可访问、可交互和可重复使用。这些原则于2016年首次正式提出。
首先基于可查询和可访问的原则,需要数据库,它能存储现有数据也能继续转化新的数据。这个平台可以是自己从零开始设计,可以是使用现成的数据库构建工具作为辅助(比如mako软件),也可以是直接使用已有的数据库,比如GloBI。
关于数据获取,文中列出了一些资源,都是目前广泛使用或者比较独特的,如下表:

其次基于交互性和可重复使用的原则,需要将大数据拆分为一个个子集,每一个子集都应该是能满足大量用户的第一需求或者说是第一想了解的信息点。基于此,研究人员提出了四类元数据作为起点:
1. 微生物实体
应提供参与相互作用的每种微生物的物种(和菌株,如果相关)名称。
示例:
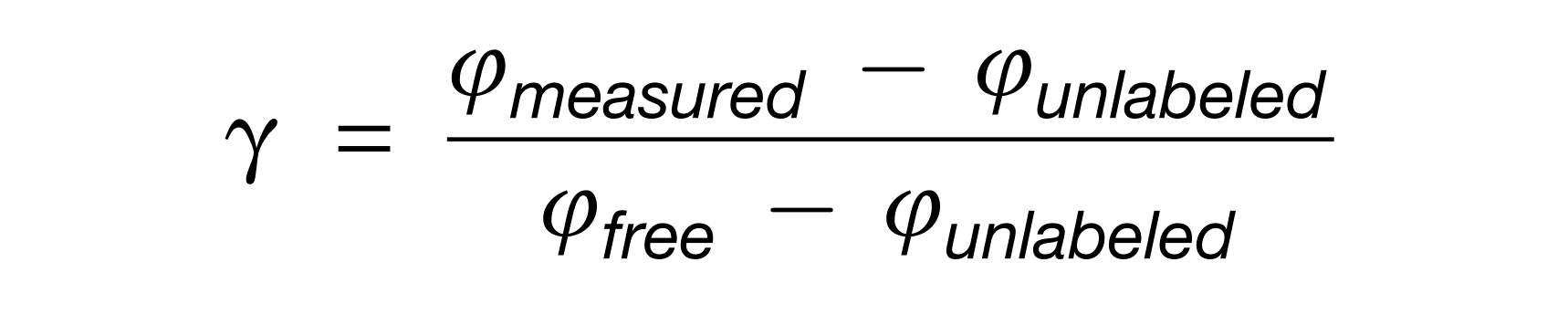
2. 相互作用的推理方法
使用不同的方法来指示高度相关的元数据。
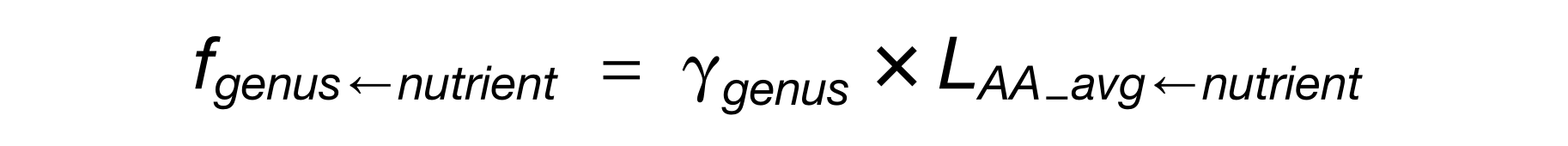
示例:

3. 相互作用的上下文语境
一些环境背景,如生物群落(例如,宿主相关的、合成的)。

示例:

4. 属性,用于定义互作关系的类型
如合作、对抗、关联、成对或高阶等。
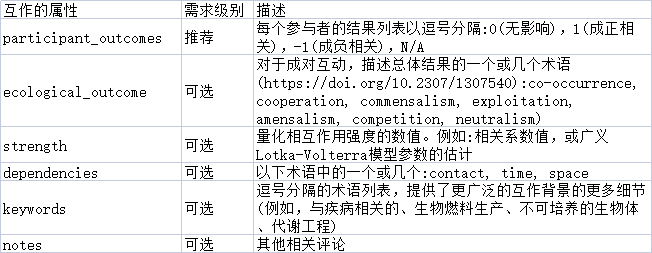
示例:

最后,文章中给出了依照FAIR体系研究微生物互作关系的可拓展性。
如下图,通过研究微生物相互作用和相关性(A部分)而产生的多种类型的元数据(B部分),这些元数据(DT),例如真菌细菌或噬菌体细菌的培养实验数据(DT1),扩增子序列或OTU计数的相关性(DT2),两个或两个以上物种基因组尺度代谢网络的通量平衡模型(DT3),直接比对到特定数据库(DT4)。
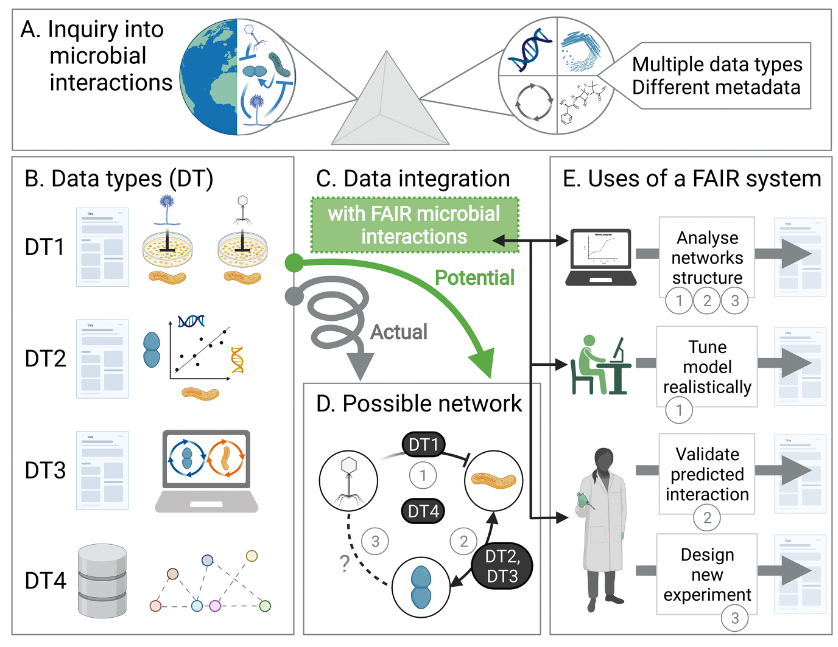
遵循FAIR里的四个原则来解析微生物互作内容(C部分),解析结果如D部分,得到一个可能的相互作用网络关系图谱。
同时,C部分的内容可以横跨不同领域,为不同领域的专家提供信息,例如E部分,建模者可以更容易确定具体的互作以便真实的模拟生态动力学,而实验人员可以评估是否有新的互作在其他宿主或上下文中被报告。
以上内容,是研究人员基于自身经验归纳总结的,研究人员也指出在此概述的实践、标准和示例并不是详尽无遗的,文章旨在促进进一步讨论如何改进微生物相互作用及其属性数据的访问和可用性。希望能进一步的激励各位科学家,共同创建一个共享开放的,拥有统一标准的微生物相互作用资源站。
参考文献:
Pacheco AR, Pauvert C, Kishore D, Segrè D. Toward FAIR Representations of Microbial Interactions. mSystems. 2022 Aug 25:e0065922. doi: 10.1128/msystems.00659-22. Epub ahead of print. PMID: 36005399.