-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com

做过16s测序的小伙伴们都知道
测完之后会拿到一份结果报告
但这并不代表可以开始写文章了
看似一大堆数据图表却不知如何下手
这是很多人头疼的地方
那么怎样给报告中的数据赋予灵魂
让它真正成为对你有帮助的分析呢?
今天我们来详细解读下。
一文扫除困惑
首先什么是16S rRNA?
16S rRNA 基因是编码原核生物核糖体小亚基的基因,长度约为1542bp,其分子大小适中,突变率小,是细菌系统分类学研究中最常用和最有用的标志。
16S rRNA基因序列包括9个可变区和10个保守区,保守区序列反映了物种间的亲缘关系, 而可变区序列则能体现物种间的差异。
16S rRNA基因测序以细菌16S rRNA基因测序为主,核心是研究样品中的物种分类、物种丰度以及系统进化。
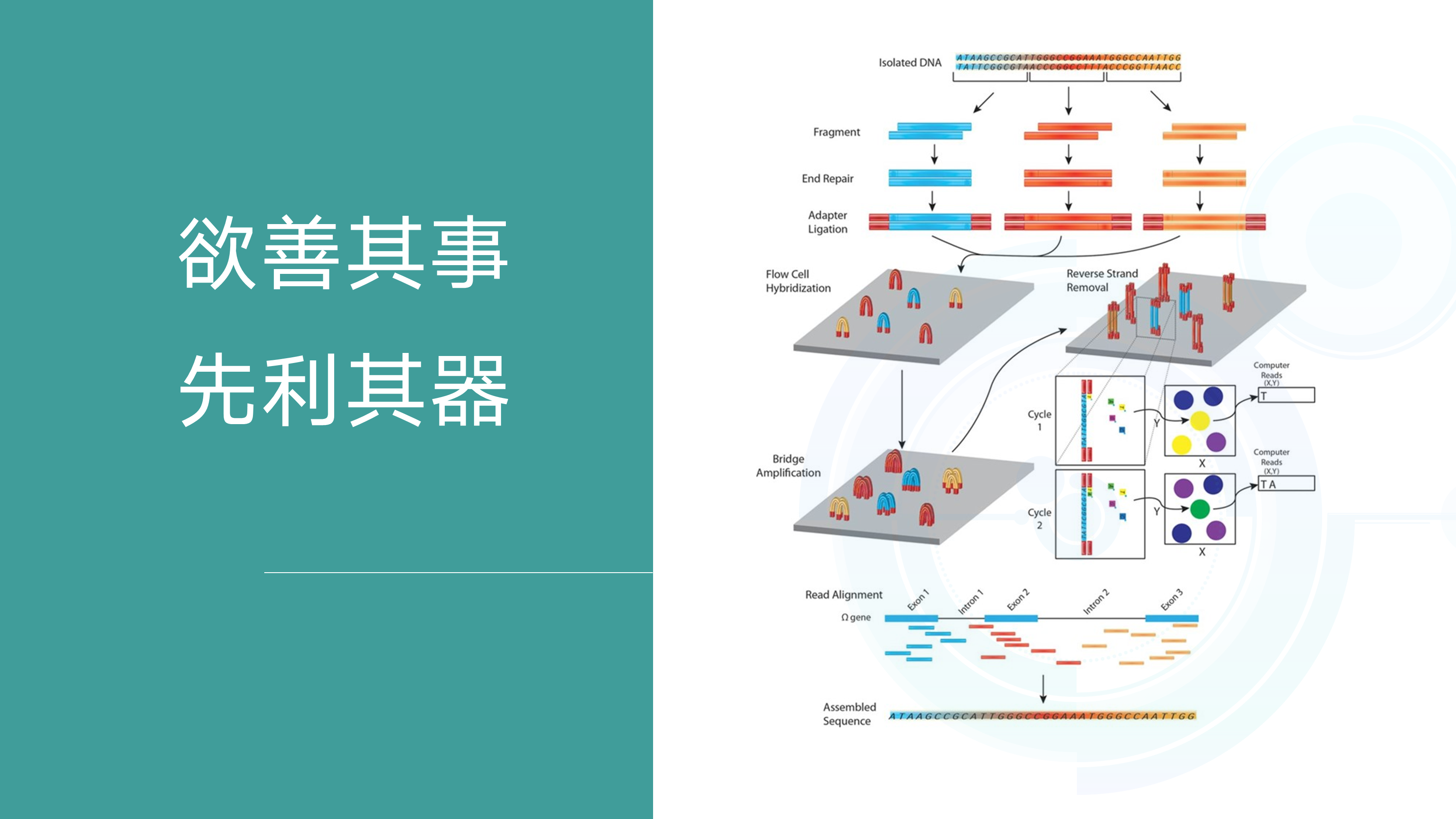
二代高通量测序原理
目前二代测序是一个边合成边测序的过程,使用的是荧光可逆终止子。每个可逆终止子的碱基3’端都有一个阻断基团,而在侧边带有一种荧光。由于有4种不同的碱基(ATCG),因此也会有对应4种不同颜色的荧光。开始扩增每次结合上一个碱基,DNA的扩增便会停止,此时能收到一种荧光信号。然后放试剂除去阻断基团,进行下一个碱基的结合,以此类推得到一连串的荧光信号组合序列。而根据荧光的颜色我们便可以确定每一个位点的基因型,即可以得到这一段DNA片段的序列。
环境样品高通量分析需要重复么?
在进行实验设计前,这是有些小伙伴面临的一个问题。环境样本由于来源和条件不完全可控,每个样品之间会存在很大的差异,即便是相同样本的不同取样时间和部位也会存在一定的差异。
基于高通量测序主要是为了了解样品的菌群构成和功能分析,以及寻找不同环境之间的差异,包括菌和功能基因以及代谢。如果仅做单一样本,很可能结论只能代表这个单一取样样本的信息,无法排除不同样本重复之间的差异,也就可能得不到真正代表环境差异的结果。
所以环境样品不仅要重复而且还应该以分组方式取尽量多的样本以全面的代表一个环境条件下的各种变异情况。
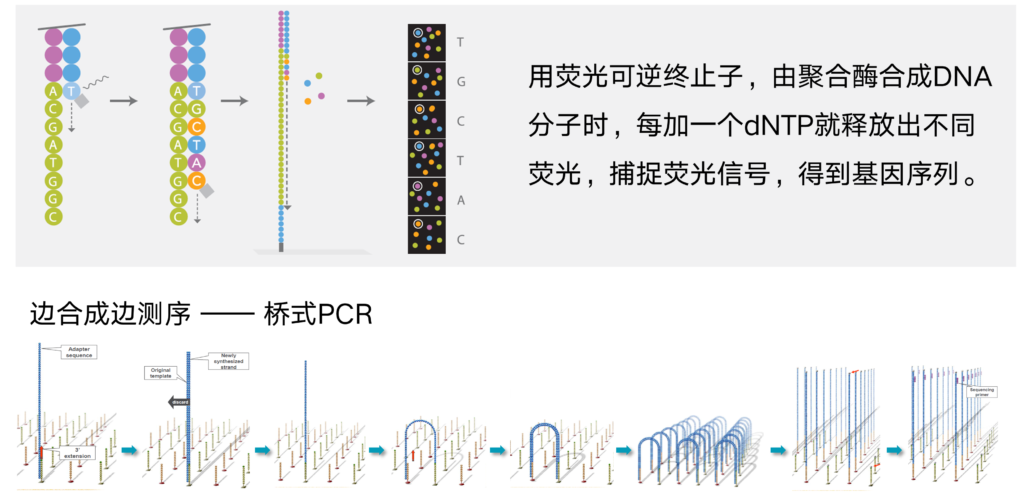
测序区段如何选择
确定做重复后,又面临该怎么选择测序区段的问题。目前市面上有v1-v3区/v3-v4区/v4区等可供选择。
16S rRNA编码基因序列共有9个保守区和9个高可变区。其中,V4区其特异性好,数据库信息全,我们通过大量的测序试验证明用v4区扩增出菌群结果的可以很好的反应样本的菌群结构用于后续的数据建模分析,是细菌多样性分析注释的最佳选择。
基本确定好后,就要着手开始实验,实验完送样又是个问题,以往给测序公司送样往往是低温运输,且不说麻烦,还要提心吊胆怕运输过程会不会有什么问题。为此我们免费提供常温保存取样盒,就不用有这样的顾虑,取样及运输全程都只需要常温即可。
样品到公司之后就更不用操心,全套服务等着呢!

16s分析结果详解
很多小伙伴有过这样的经历,在拿到公司出具的报告之后,仍然一头雾水,几十页的报告内容看着丰富却不知该怎么运用。我们一起来理一下关键图表的含义。
OTU是我们要搞清的一个重要概念,可以说是后续分析的基石。
OTU(operational taxonomic units) 是在系统发生学研究或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系,种,属,分组等)设置的同一标志。通常按照 97% 的相似性阈值将序列划分为不同的 OTU,每一个 OTU 通常被视为一个微生物物种。相似性小于97%就可以认为属于不同的种,相似性小于93%-95%,可以认为属于不同的属。样品中的微生物多样性和不同微生物的丰度都是基于对OTU的分析。
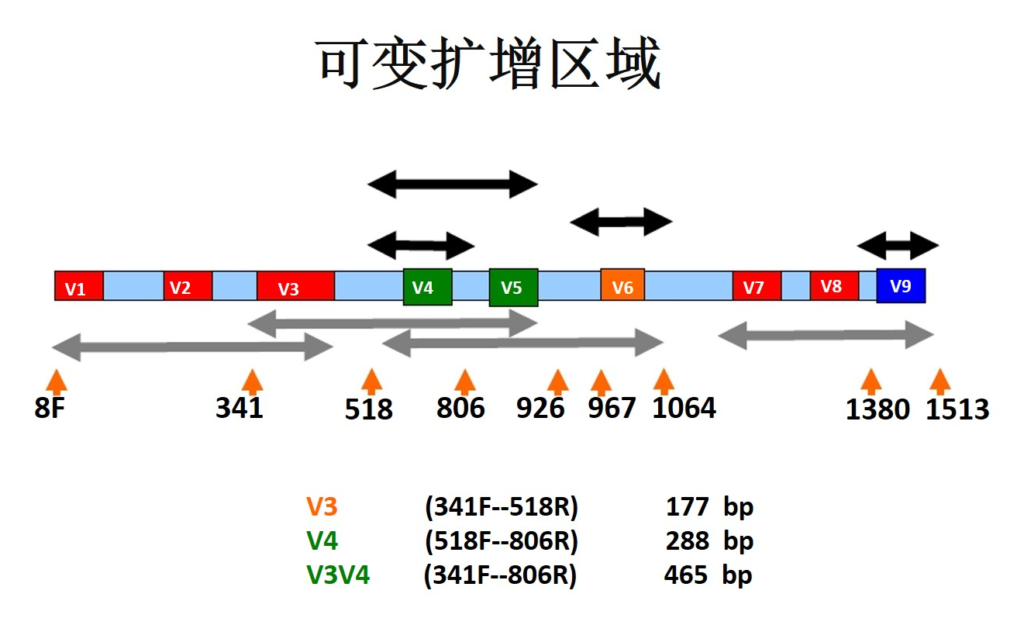
有了OTU这个概念之后,就不难理解下表。对每个样本的测序数量和OTU数目进行统计,并且在表栺中列出了测序覆盖的完整度。
其中 SampleName表示样本名称;SampleSize表示样本序列总数;OTUsNumber表示注释上的OTU数目;OTUsSeq表示注释上OTU的样本序列总数。
Coverage是指各样品文库的覆盖率,其数值越高,则样本中序列没有被测出的概率越低。该指数实际反映了本次测序结果是否代表样本的真实情况。计算公式为:C=1-n1/N 其中n1 = 只含有一条序列的OTU的数目;N = 抽样中出现的总的序列数目。
下表是对每个样本在分类字水平上的数量进行统计,并且在表栺中列出了在每个分类字水平上的物种数目
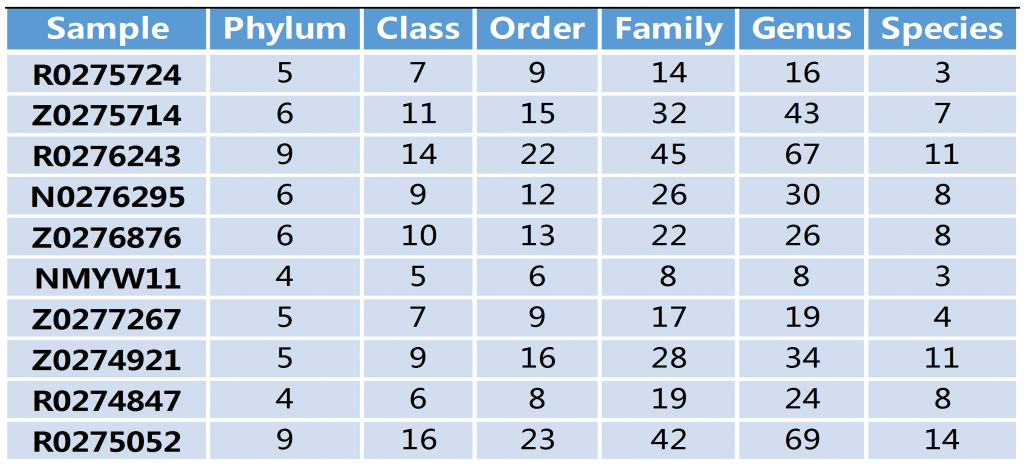
其中SampleName表示样本名称;Phylum表示分类到门的OTU数量;Class表示分类到纲的OTU数量;Order表示分类到目的OTU数量;Family表示分类到科的OTU数量;Genus表示分类到属的OTU数量;Species表示分类到种的OTU数量。
我们可以看到绝大部分的OTU都分类到了属(Genus),也有很多分类到了种(Species)。但是仍然有很多无法完全分类到种一级,这是由于环境微生物本身存在非常丰富的多样性,还有大量的菌仍然没有被测序和发现。
当然,对这些种属的构成还可以进行柱状图展示:
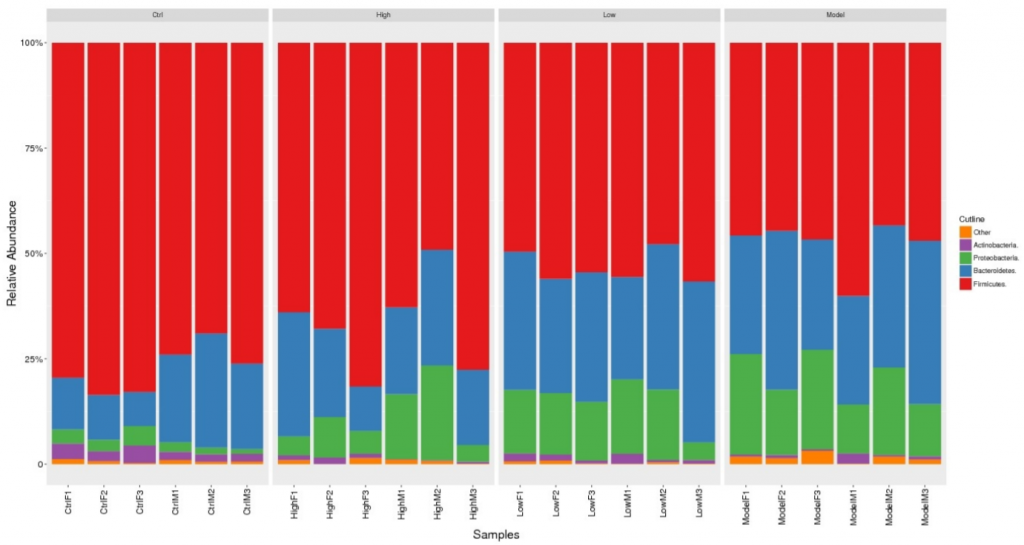
横坐标中每一个条形图代表一个样本,纵坐标代表该分类层级的序列数目或比例。同一种颜色代表相同的分类级别。图中的每根柱子中的颜色表示该样本在不同级别(门、纲、目等)的序列数目,序列数目只计算级别最低的分类,例如在属中计算过了,则在科中则不重复计算。
我们还需要对样本之间或分组之间的OTU进行比较获得韦恩图:
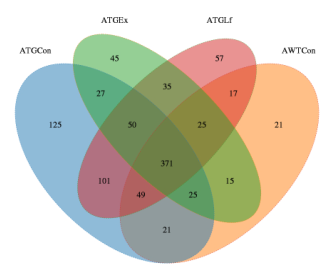
样品构成丰度
稀释曲线
微生物多样性分析中如何验证测序数据量是否足以反映样品中的物种多样性?
稀释曲线(丰富度曲线)可以派上用场。它是用来评价测序量是否足以覆盖所有类群,并间接反映样品中物种的丰富程度。
不免有同学有疑惑,稀释曲线怎么来的?
它是利用已测得16S rDNA序列中已知的各种OTU的相对比例,来计算抽取n个(n小于测得reads序列总数)reads时出现OTU数量的期望值,然后根据一组n值(一般为一组小于总序列数的等差数列)与其相对应的OTU数量的期望值做出曲线来。
至此,我们虽然知道了稀释曲线的由来,那么这个五彩缤纷的稀释曲线该怎么看呢?
当曲线趋于平缓或者达到平台期时也就可以认为测序深度已经基本覆盖到样品中所有的物种,增加测序数据无法再找到更多的OTU;
反之,则表示样品中物种多样性较高,还存在较多未被测序检测到的物种。
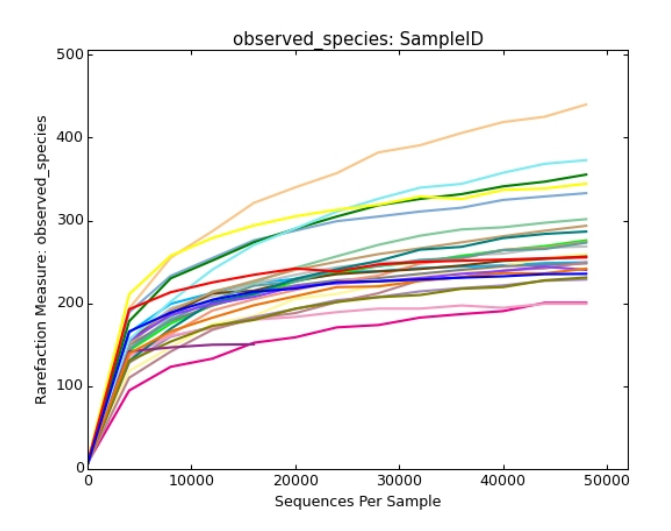
横坐标代表随机抽取的序列数量;纵坐标代表观测到的OTU数量。样本曲线的延伸终点的横坐标位置为该样本的测序数量。
Shannon-Winner曲线
Shannon-Wiener 曲线,是利用shannon指数来进行绘制的,反映样品中微生物多样性的指数,利用各样品的测序量在不同测序深度时的微生物多样性指数构建曲线,以此反映各样本在不同测序数量时的微生物多样性。
当曲线趋向平坦时,说明测序数据量足够大,可以反映样品中绝大多数的微生物物种信息。
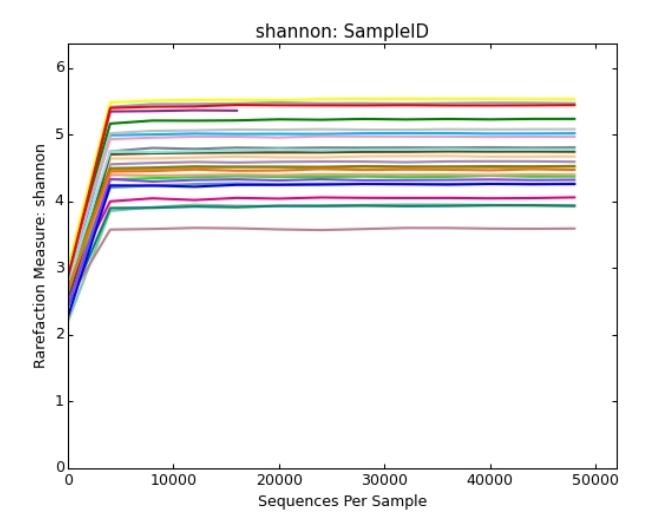
横坐标代表随机抽取的序列数量;纵坐标代表的是反映物种多样性的Shannon指数,样本曲线的延伸终点的横坐标位置为该样本的测序数量。
其中曲线的最高点也就是该样本的Shannon指数,指数越高表明样品的物种多样性越高。
好奇的同学又有疑问,Shannon指数怎么算的?
这里有Shannon指数的公式:
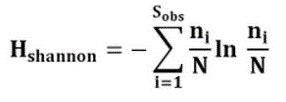
其中,Sobs= 实际测量出的OTU数目;
ni= 含有i 条序列的OTU数目;N = 所有的序列数。
Rank-Abundance曲线
该曲线用于同时解释样品多样性的两个方面,即样品所含物种的丰富程度和均匀程度。
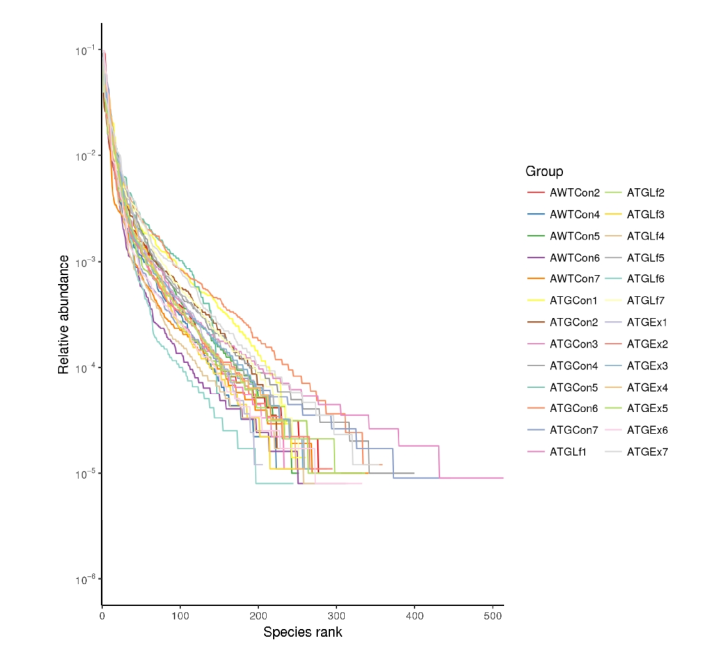
横坐标代表物种排序的数量;纵坐标代表观测到的相对丰度。
样本曲线的延伸终点的横坐标位置为该样本的物种数量
物种的丰富程度由曲线在横轴上的长度来反映,曲线越宽,表示物种的组成越丰富;
物种组成的均匀程度由曲线的形状来反映,曲线越平坦,表示物种组成的均匀程度越高。
如果曲线越平滑下降表明样本的物种多样性越高,而曲线快速陡然下降表明样本中的优势菌群所占比例很高,多样性较低。
但一般超过20个样本图就会变得非常复杂而且不美观!所以假如没超过20个样可以考虑该图哦~
Alpha多样性(样本内多样性)
Alpha多样性是指一个特定区域或者生态系统内的多样性,常用的度量指标有Chao1 丰富度估计量(Chao1 richness estimator) 、香农 – 威纳多样性指数(Shannon-wiener diversity index)、辛普森多样性指数(Simpson diversity index)等。
计算菌群丰度:Chao、ace;
计算菌群多样性:Shannon、Simpson。
Simpson指数值越大,说明群落多样性越高;Shannon指数越大,说明群落多样性越高。
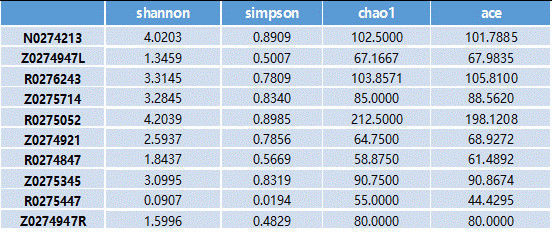
看了那么多指数,可能觉得有点晕,到底每个指数是什么意思呢?
当然要解释下咯:
Chao1:是用chao1 算法计算群落中只检测到1次和2次的OTU数估计群落中实际存在的物种数。Chao1 在生态学中常用来估计物种总数,由Chao (1984) 最早提出。Chao1值越大代表物种总数越多。
Schao1=Sobs+n1(n1-1)/2(n2+1)
其中Schao1为估计的OTU数,Sobs为观测到的OTU数,n1为只有一条序列的OTU数目,n2为只有两条序列的OTU数目。
Shannon:用来估算样品中微生物的多样性指数之一。它与 Simpson 多样性指数均为常用的反映 alpha 多样性的指数。Shannon值越大,说明群落多样性越高。
Ace:用来估计群落中含有OTU 数目的指数,由Chao 提出,是生态学中估计物种总数的常用指数之一,与Chao1 的算法不同。
Simpson:用来估算样品中微生物的多样性指数之一,由Edward Hugh Simpson ( 1949) 提出,在生态学中常用来定量的描述一个区域的生物多样性。Simpson 指数值越大,说明群落多样性越高。

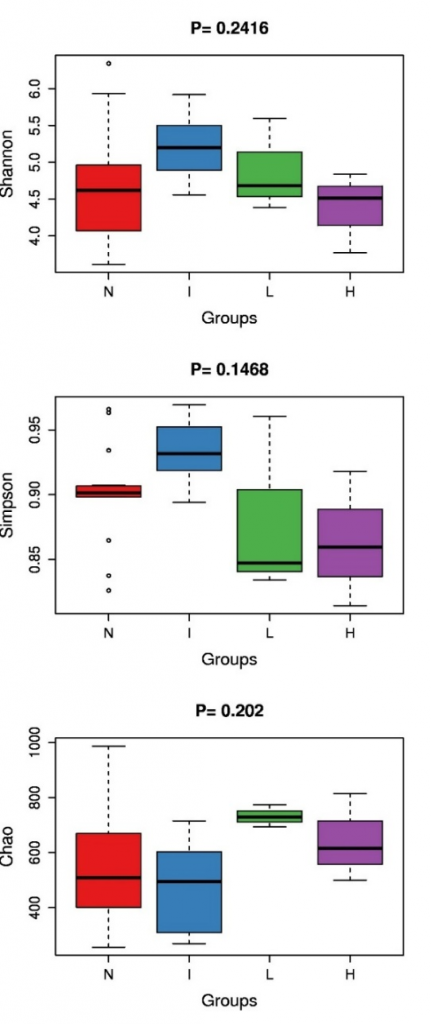
Alpha多样性指数差异箱形图
分别对 Alpha diversity 的各个指数进行秩和检验分析(若两组样品比较则使用 R 中的wilcox.test 函数,若两组以上的样品比较则使用 R 中的 kruskal.test 函数),通过秩和检验筛选不同条件下的显著差异的 Alpha Diversity指数。
Beta多样性分析(样品间差异分析)
也许我们有听说Beta多样性在最近10年间成为生物多样性研究的热点问题之一。具体解释下:
Beta多样性度量时空尺度上物种组成的变化, 是生物多样性的重要组成部分, 与许多生态学和进化生物学问题密切相关!
PCoA分析
PCoA(principal co-ordinates analysis)是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,PCoA 可以找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。
重要的是,它是可以用来观察个体或群体间的差异的。
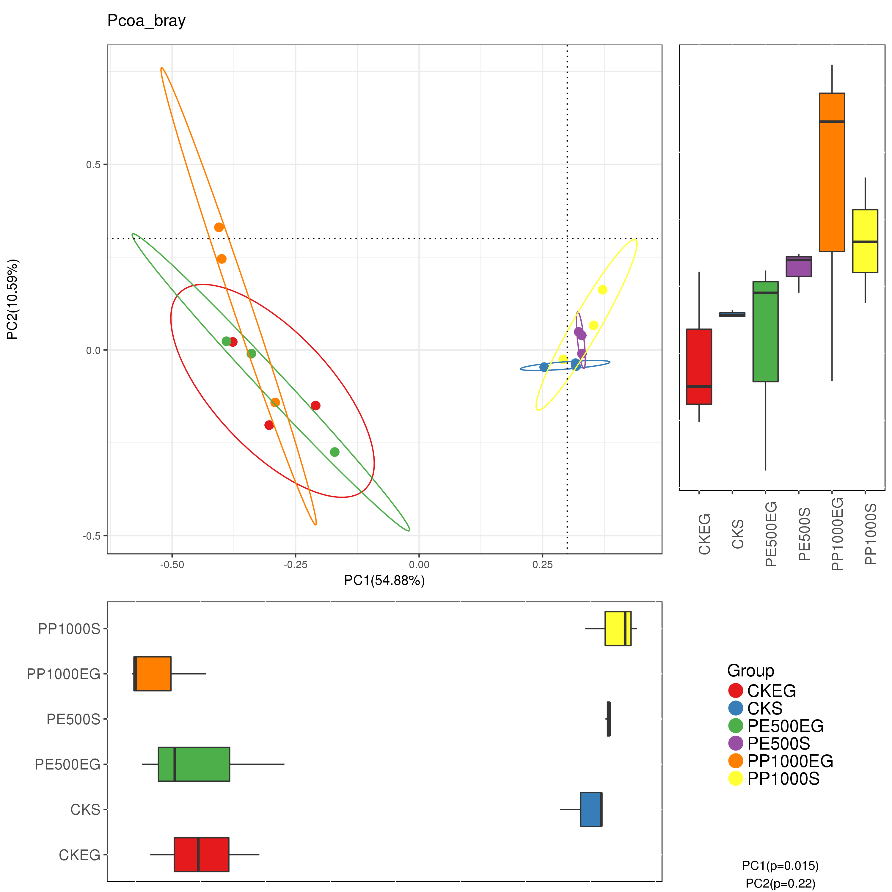
每一个点代表一个样本,相同颜色的点来自同一个分组,两点之间距离越近表明两者的群落构成差异越小。
另一种相似的是PCA分析
主成分分析(Principal component analysis)PCA 是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要的前几位特征值,采取降维的思想,PCA 可以找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。
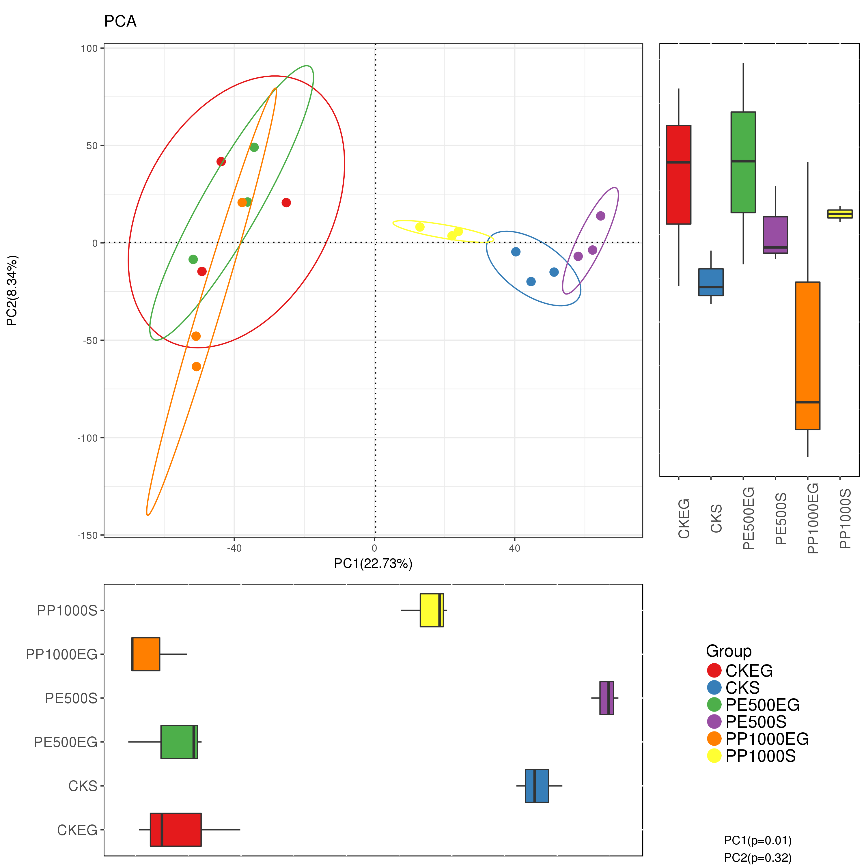
详细关于主成分分析的解释推荐大家看一篇文章,http://blog.csdn.net/aywhehe/article/details/5736659
一起来看看包含PCoA研究的文章
案例解析



研究背景:全球塑料产量飞速增长,而且呈持续上升的趋势,因此导致大量塑料废物排放到环境中,从沿海河口到大洋环流,从东大西洋到南太平洋海域。塑料废弃物具有化学稳定性和生物利用率低的特点,可长期存在于海洋中,从而影响海洋环境包括海洋生物的生存。
作为一个独特的底物,塑料碎片可以吸附海洋中的微生物并形成个“塑性球”。以生物膜形式存在于塑料碎片上的微生物群落。许多研究表明,无论是在海洋还是淡水生态系统中,附着在塑料碎片上微生物群落的组成明显不同于周围环境(水和沉积物),而且易受位置、时间和塑料类型的影响。
主要图表
两两群落差异指数的PCoA图
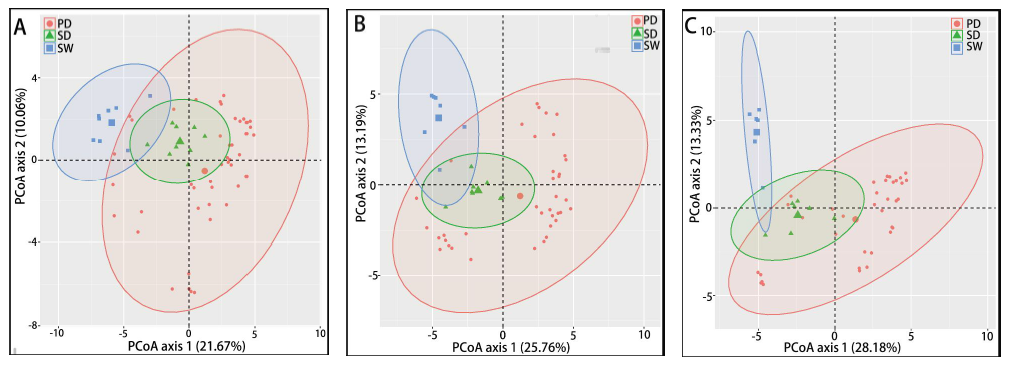
PCoA 图可以清楚地看到,SW区细菌群落的置信椭圆与pd和sd的置信椭圆有显著的偏差(p<0.05),而sd上细菌群落的置信椭圆几乎覆盖了pd的置信椭圆(p>0.05),这表明pd和sd上的细菌群落有相似之处。
不同样本和处理下的细菌群落( 前 10 位)丰度分布
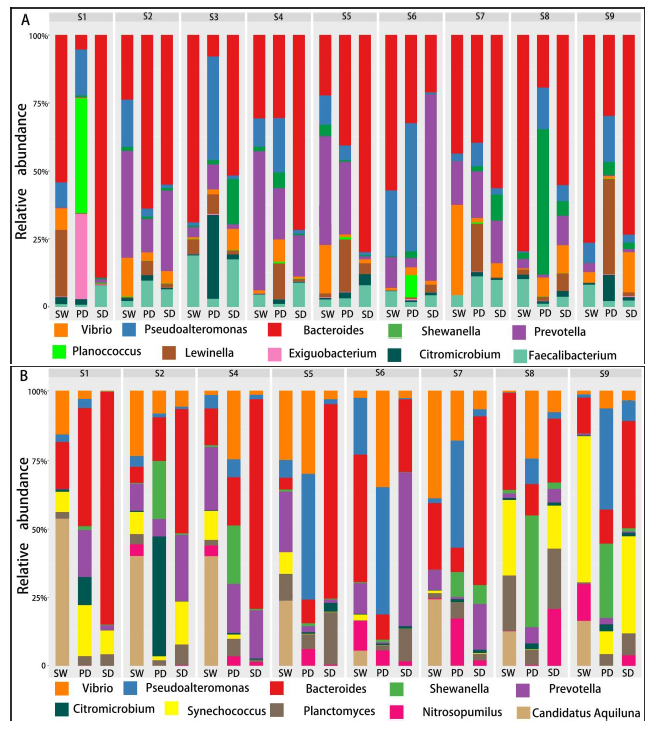
底物(SW、SD和Pd)上的主要属为细菌和假互斥单胞菌,暴露两周后,这些菌可能是分布广泛和适应性强的三种底物(SW、SD和PD)。暴露4周后,弧菌相对丰度增加.此外,暴露6周后,自养细菌(如扁平菌和硝酸菌)的数量增加。这三种底物上个细菌群落的生长模式也与3.2的结果一致。图5还显示,在6个星期内,在429个原位点中,假单胞菌在pd上的相对丰度高于sw和sd(anova,p<0.05)。
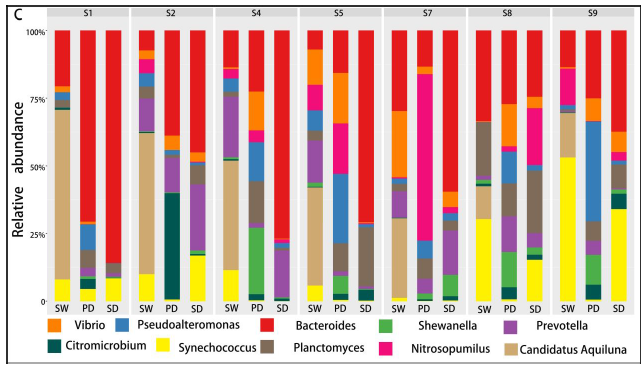
研究结论:首先,营养物质 (TN 和 TP) 与生物膜的平均生长速率呈正相关,而盐度与生物膜的平均生长速率呈负相关。盐度是影响PD的个细菌多样性的主要因素,而温度、溶解氧和养分(TN和TP)在类似的盐度条件下可能具有二次效应。尽管种聚合物类型对PD上的细菌群落的多样性具有较少的影响,但是在细菌群落中的一些属显示对PD的聚合物类型的选择性,并且倾向于将其优选的基质定殖。大的相对丰度SW、PD、SD间属显著差异。盐度是改变河口地区Pd条件致病菌富集的主要因素。另外,在种病原物种丰富的基础上,PD具有较高的致病性。
NMDS分析(非度量多维尺度分析)
NMDS(Nonmetric Multidimensional Scaling)常用于比对样本组之间的差异,可以基于进化关系或数量距离矩阵。
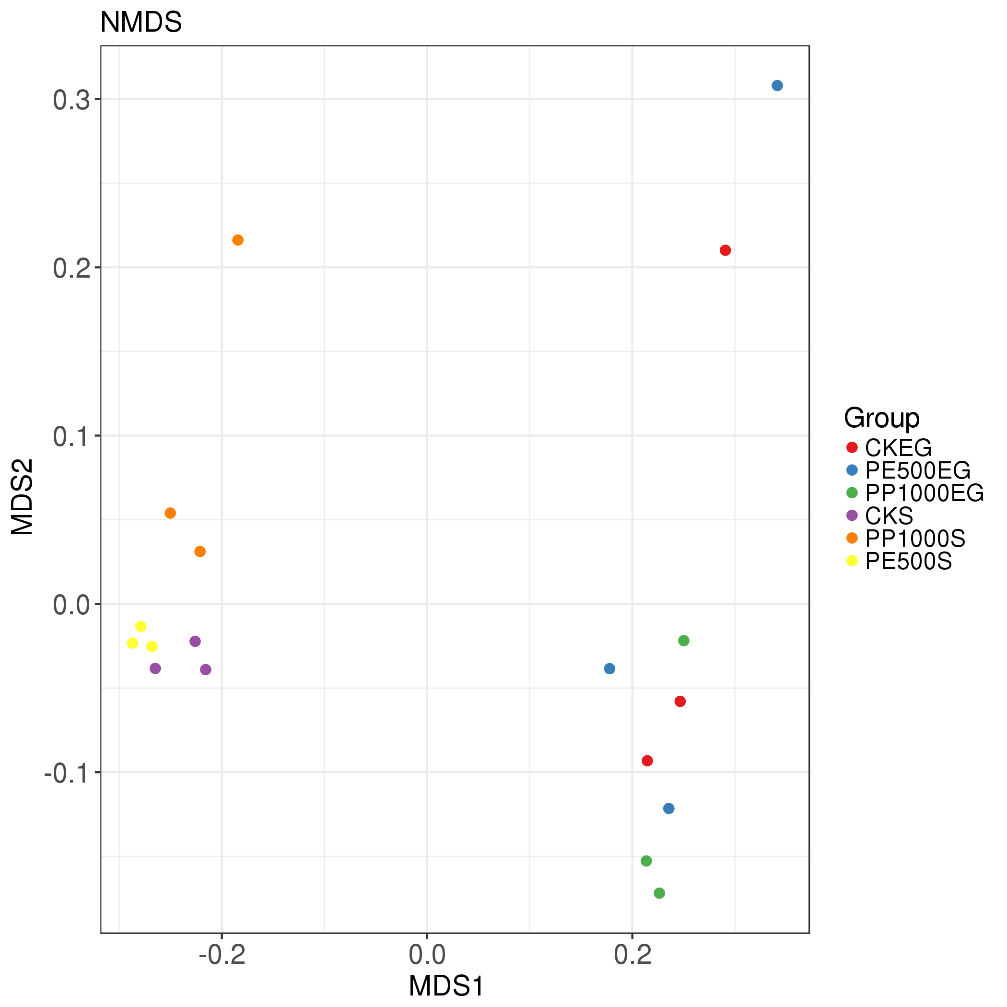
横轴和纵轴:表示基于进化或者数量距离矩阵的数值在二维表中成图。与PCA分析的主要差异在于考量了进化上的信息。
每一个点代表一个样本,相同颜色的点来自同一个分组,两点之间距离越近表明两者的群落构成差异越小。
排序分析
PCA,PcoA,NMDS分析都属于排序分析(Ordination analysis)。
排序(ordination)的过程就是在一个可视化的低维空间或平面重新排列这些样本。
目的:使得样本之间的距离最大程度地反映出平面散点图内样本之间的关系信息。
排序又分两种:非限制性排序和限制性排序。
1、非限制性排序(unconstrained ordination)
——只使用物种组成数据的排序
(1) 主成分分析(principal components analysis,PCA)
(2) 对应分析(correspondence analysis, CA)
(3) 去趋势对应分析(Detrended correspondence analysis, DCA)
(4) 主坐标分析(principal coordinate analysis, PCoA)
(5) 非度量多维尺度分析(non-metric multi-dimensional scaling, NMDS)
2、限制性排序(constrained ordination)
——同时使用物种和环境因子组成数据的排序
(1) 冗余分析(redundancy analysis,RDA)
(2) 典范对应分析(canonical correspondence analysis, CCA)
比较PCA和PCoA
在非限制性排序中,16S和宏基因组数据分析通常用到的是PCA分析和PCoA分析,两者的区别在于:
PCA分析是基于原始的物种组成矩阵所做的排序分析,而PCoA分析则是基于由物种组成计算得到的距离矩阵得出的。
在PCoA分析中,计算距离矩阵的方法有很多种,包括如:Euclidean, Bray-Curtis, and Jaccard,以及(un)weighted Unifrac (利用各样品序列间的进化信息来计算样品间距离,其中weighted考虑物种的丰度,unweighted没有对物种丰度进行加权处理)。
LDA差异贡献分析
如果说 PCA,它所作的只是将整组数据整体映射到最方便表示这组数据的坐标轴上,映射时没有利用任何数据内部的分类信息,是无监督的。
那么LDA是有监督的,增加了种属之间的信息关系后,结合显著性差异标准测试(克鲁斯卡尔-沃利斯检验和两两Wilcoxon测试)和线性判别分析的方法进行特征选择。
两者相同点:
差异:
1)LDA是有监督学习的降维方法,而PCA是无监督的降维方法。(注:监督学习是从标记的训练数据来推断一个功能的机器学习任务。)
2)LDA选择分类性能最好的投影方向,而PCA选择样本点投影具有最大方差的方向。
除了可以检测重要特征,他还可以根据效应值进行功能特性排序,这些功能特性可以解释大部分生物学差异。这部分希望能详细了解的同学可以参考这篇文章http://blog.csdn.net/sunmenggmail/article/details/8071502 。
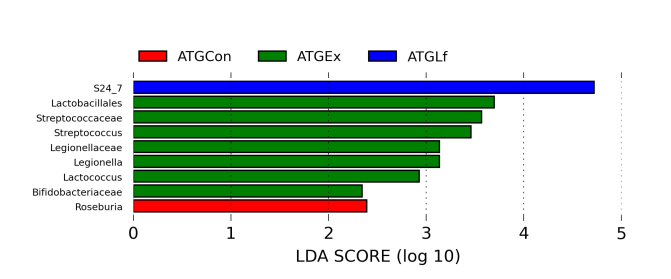
LDA分析究竟能做什么
组间差异显著物种又可以称作生物标记物(biomarkers),这个LDA分析主要是想找到组间在丰度上有显著差异的物种。
案例解析

研究背景:研究表明遗传和环境影响都在I型糖尿病的发展中起作用,增加的遗传风险不足以引起疾病,环境因素也是需要的,而且起着至关重要的作用。肠道菌群也许就是这个重要的环境因素,肠道菌群在免疫系统的成熟中起重要作用,此外还影响自身免疫疾病发展。

不同遗传风险儿童的LDA差异菌群
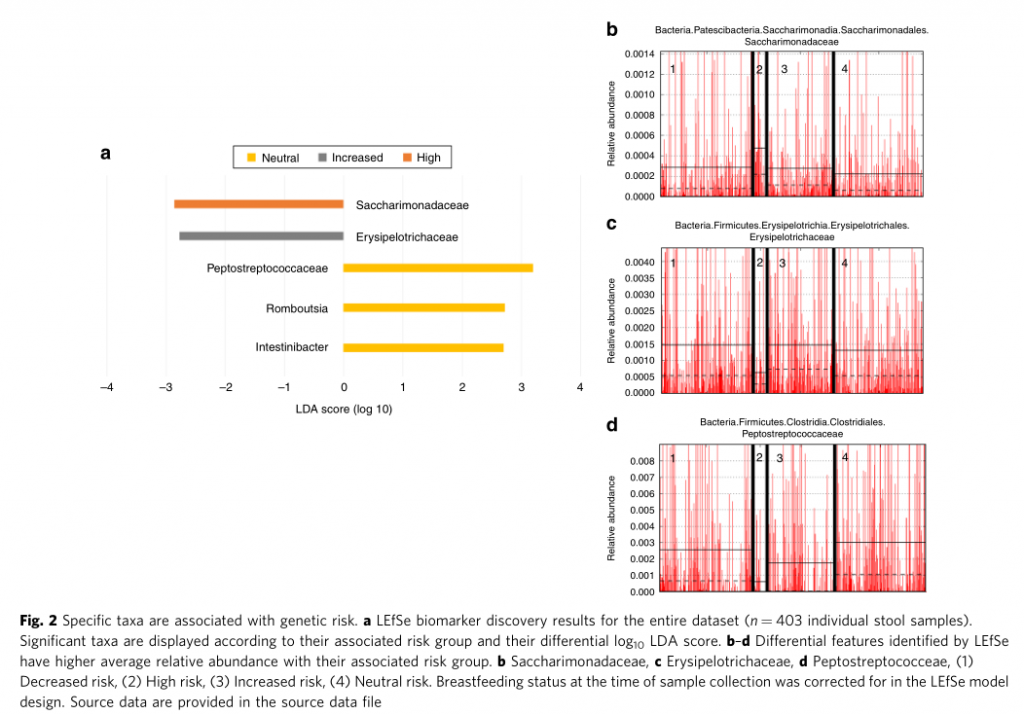
不同遗传风险分组中包含的常见菌属,部分存在特定分组中
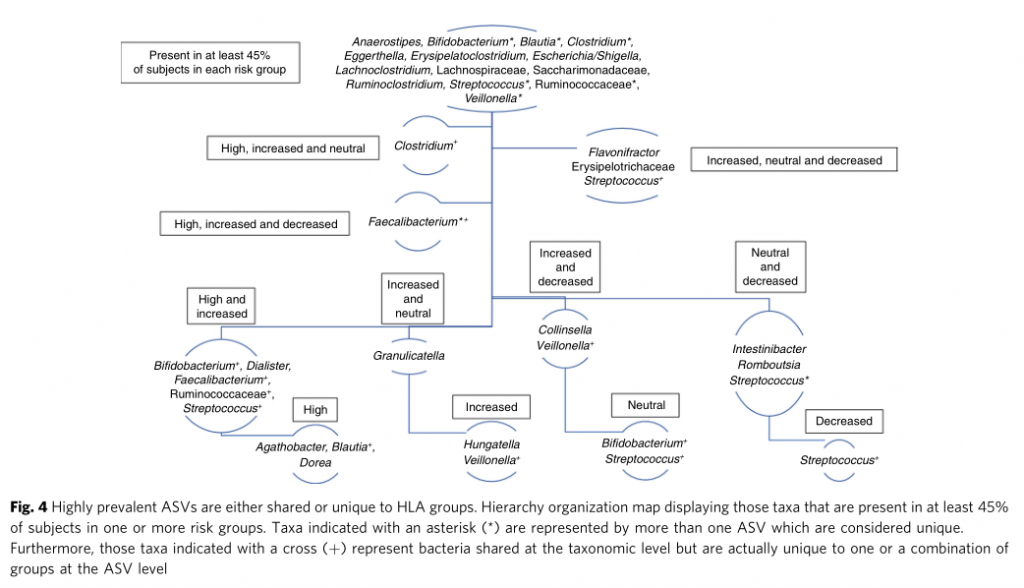
PCoA分析揭示不同遗传风险儿童肠道菌群的在不同地域样本中均存在显著差异
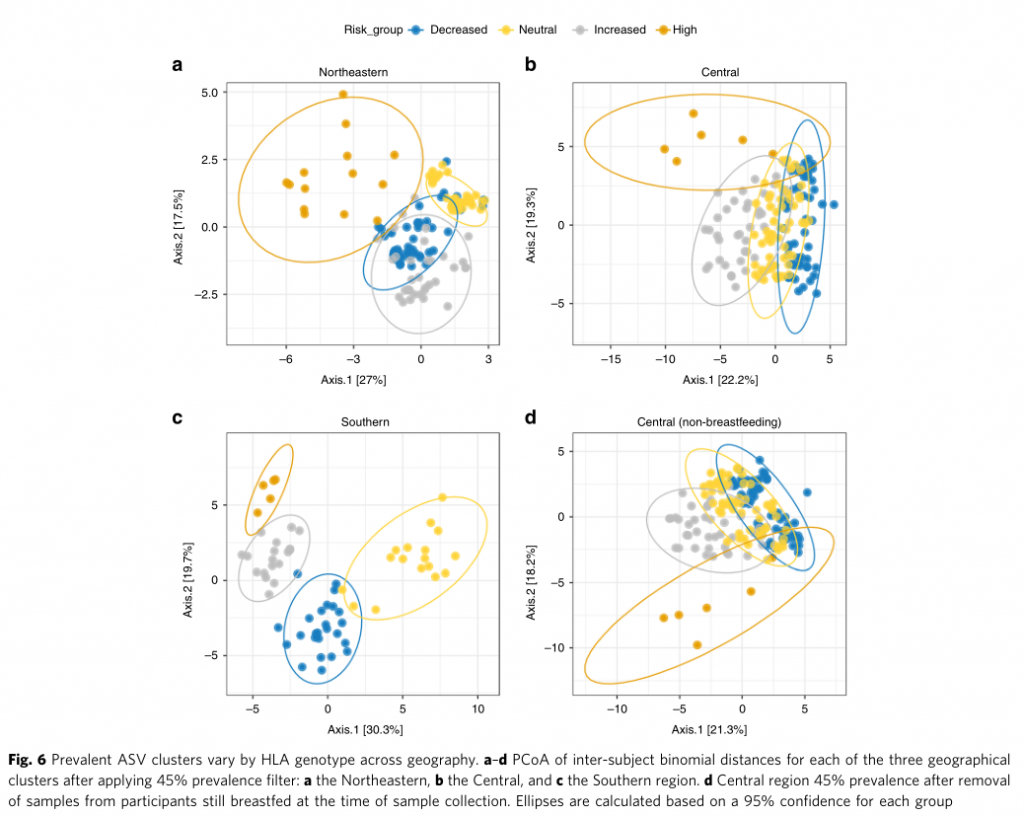
点评:针对I型糖尿病疾病发生过程中遗传HLA分型风险和对应肠道菌群菌的关联分析,揭示了特定肠道菌群与宿主特定遗传风险共同作用推进疾病发生。某些特定菌属可能无法在遗传高风险儿童肠道内定植,可能对疾病发生存在特定作用。此外对于其他遗传风险的自身免疫疾病也具有重要提示意义,例如乳糜泻和类风湿性关节炎。
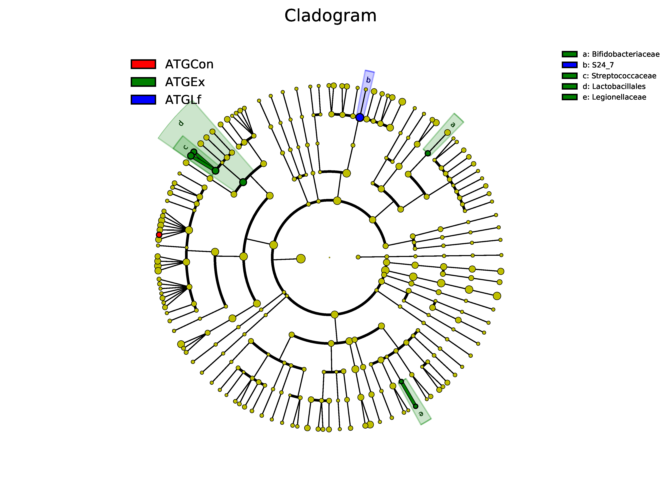
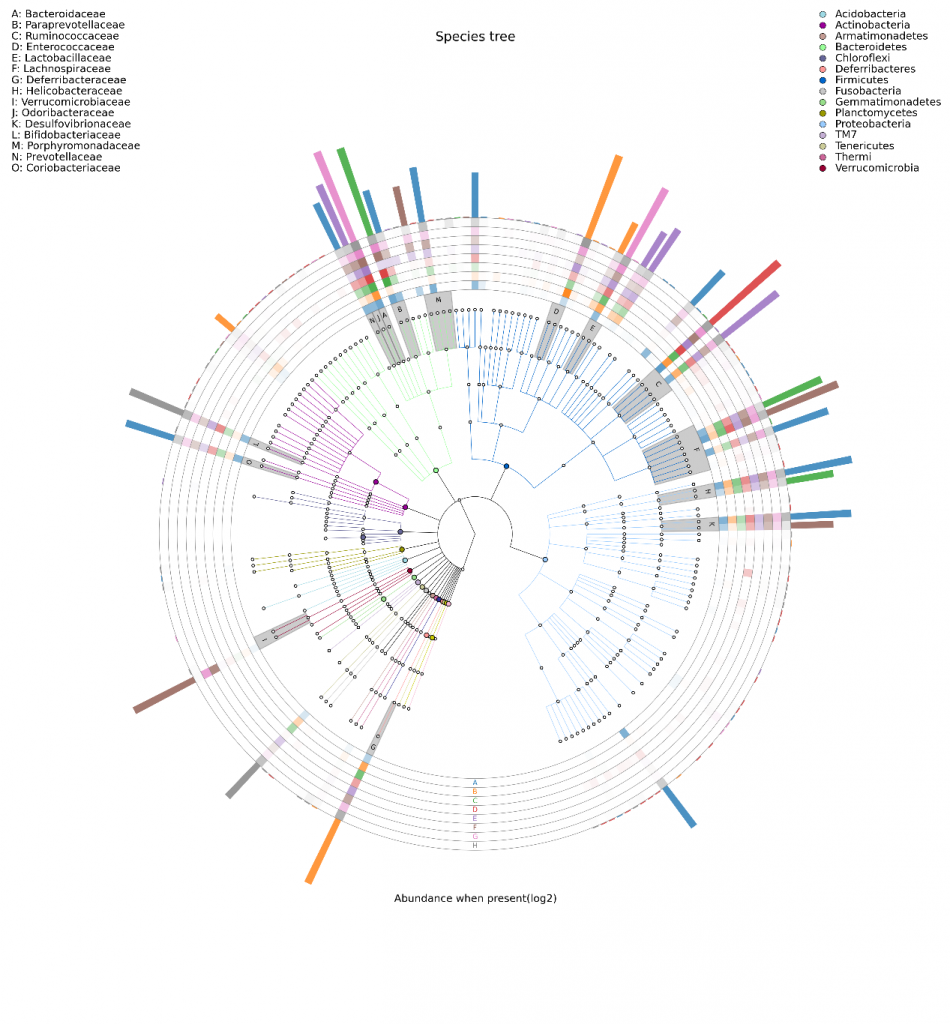
物种进化树的样本群落分布图
这是另一款和LDA长得有点像的图,当然功能可完全不一样。它是将不同样本的群落构成及分布以物种分类树的形式在一个环图中展示。数据经过分析后,将物种分类树和分类丰度信息通过这款软件GraPhlAn进行绘制
(http://huttenhower.sph.harvard.edu/GraPhlAn )。
其目的是将物种之间的进化关系以及不同样本的物种分布丰度和最高分布样本的信息在一个视觉集中的环图中一次展示,其提供的信息量较其他图最为丰富。
物种相关性分析
根据各个物种在各个样品中的丰度以及变化情况,计算物种之间的相关性,包括正相关和负相关。
相关性分析使用CCREPE算法
怎么画的?
首先对原始16s测序数据的种属数量进行标准化,然后进行Spearman和Pearson秩相关分析并进行统计检验,计算出各个物种之间的相关性,之后在所有物种中根据simscore绝对值的大小,挑选出相关性最高的前100组数据,基于Cytoscap绘制共表达分析网络图。
网络图采用两种不同的形式表现出来。
物种相关性网络图A
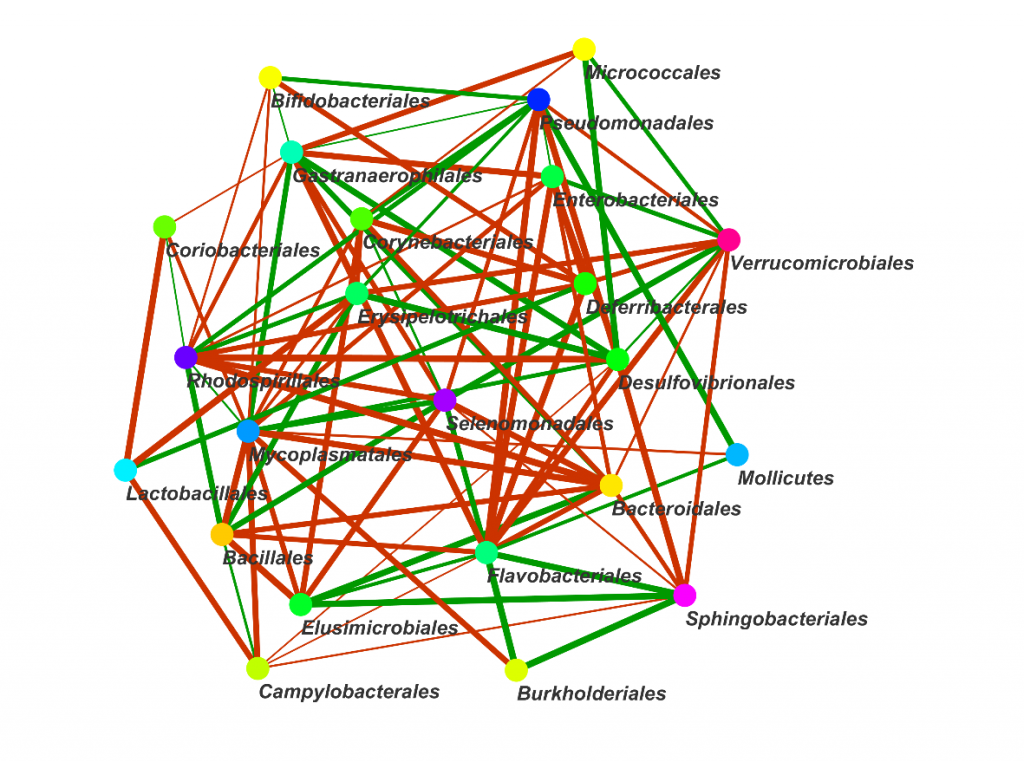
○ 图中每一个点代表一个物种,存在相关性的物种用连线连接。
○ 红色的连线代表负相关,绿色的先代表正相关。
○ 连线颜色的深浅代表相关性的高低。
物种相关性网络图B
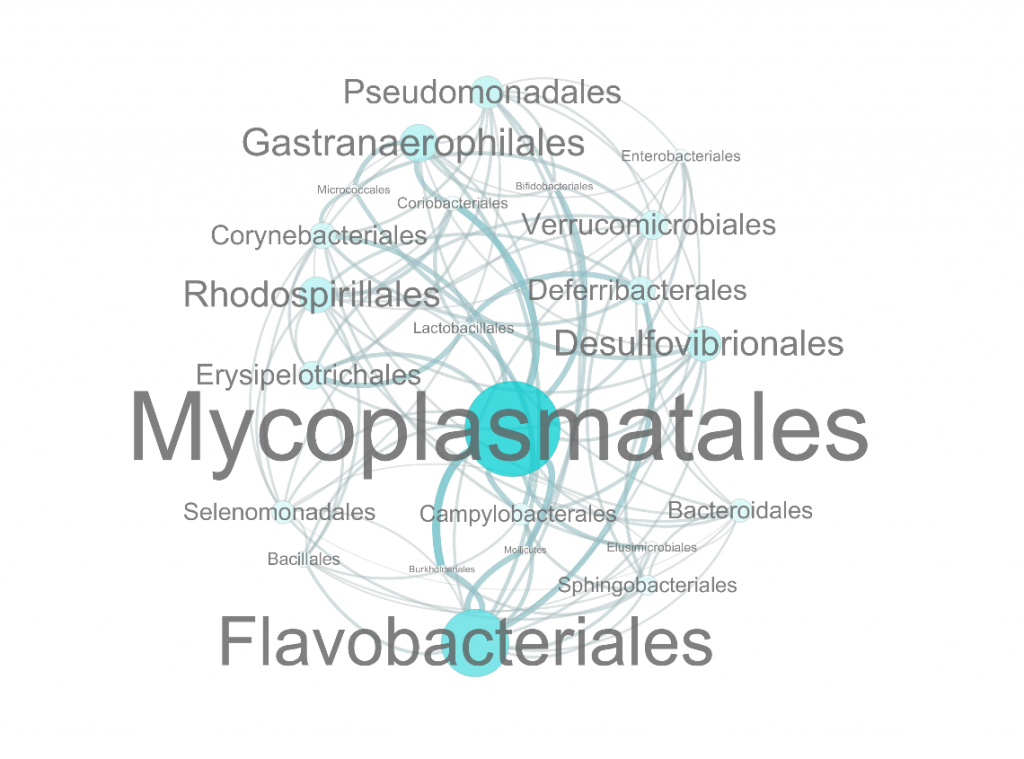
○ 图中每一个点代表一个物种
○ 点的大小表示与其他物种的关联关系的多少
○ 其中与之有相关性的物种数越多,点的半径和字体越大
○ 连线的粗细代表两物种之间相关性的大小
连线越粗,相关性越高。
案例解析


研究背景:气候变化导致美国中部草原的降水模式发生变化,对土壤微生物群落构成及代谢影响很大。
研究希望明确土壤微生物群落对土壤水分变化的反应,并确定响应的特定代谢特征。
主要图表
同一样本在不同水分含量孵化处理下土壤菌群的变化
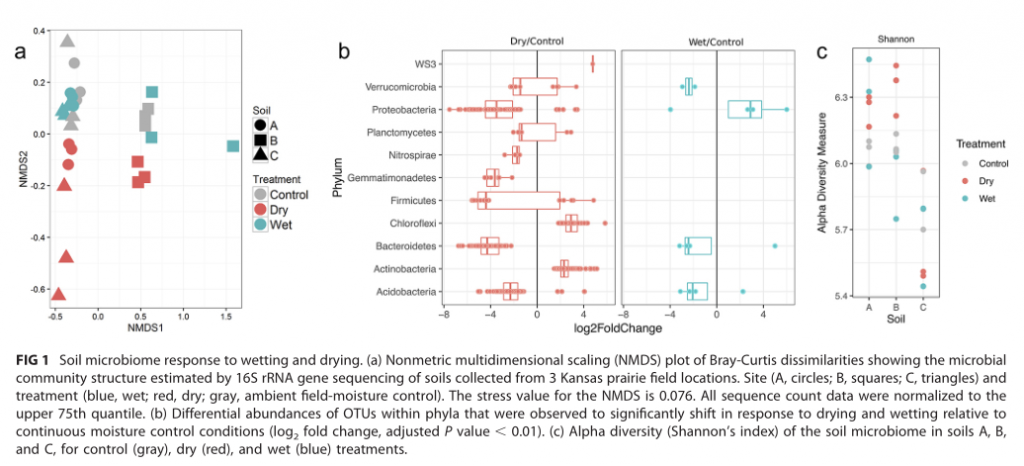

受到水分条件影响的土壤菌群代谢途径和网络分布
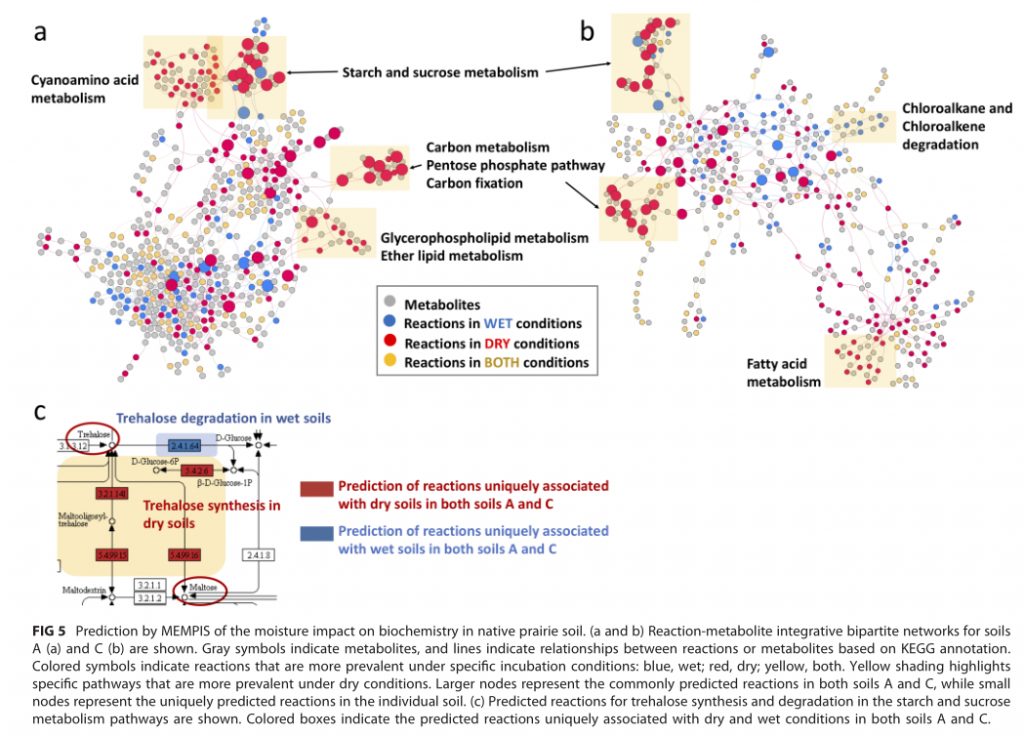

研究结论:土壤干燥导致土壤微生物组的组成和功能发生显着变化。相反,润湿后几乎没有变化。由于干旱导致的土壤水分减少对土壤碳循环和土壤微生物组进行的其他关键生物地球化学循环的影响很大。导致渗透保护剂化合物产生的代谢途径受到较大影响。
点评:
相对简单的样本和实验设计,但是从多个维度探寻支持土壤微生物群落对湿润和干燥表型的反应。
与常见的环境采样检测不同,针对同一样本在对照环境下进行环境控制孵化,然后比较菌群变化可以更为有效的控制背景差异。
聚类分析
根据OTU数据进行标准化处理(1wlog10)之后,选取数目最多的前60个物种,基于R heatmap进行作图
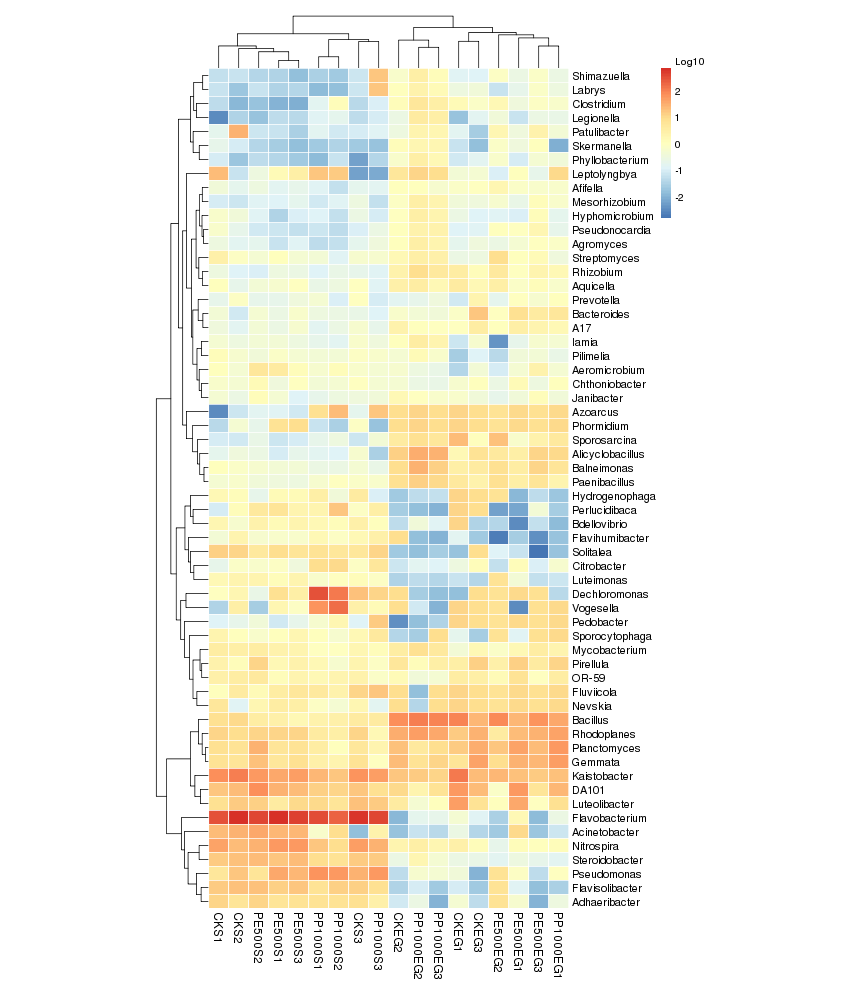

○ 热图中的每一个色块代表一个样品的一个属的丰度
○ 样品横向排列,属纵向排列
○ 差异是是否对样品进行聚类,从聚类中可以了解样品之间的相似性以及属水平上的群落构成相似性。
Tips:
如果聚类结果中出现大面积的白或黑是因为大量的菌含量非常低,导致都没有数值,可以在绘制之前进行标准化操作,对每一类菌单独自身进行Z标准化。
案例解析






研究背景:妊娠期糖尿病(GDM)的患病率在全球范围内迅速增加,构成一个重要的健康问题和产科实践的重大挑战(Ferrara,2007)。高脂血症是妊娠常见的合并症。在GDM患者中,血脂的生理变化可能导致怀孕期间潜在的代谢紊乱。肠道失调在宿主代谢异常中起着至关重要的作用,最近关于2型糖尿病(T2D)和肥胖的研究就证明了这一点。这些研究表明,妊娠期间肠道微生物ME的主要变化可能在GDM的发展中起着至关重要的作用。

GDM加高脂血症(M队列)妊娠期间与显著改变的脂质相关的肠道微生物群(属)
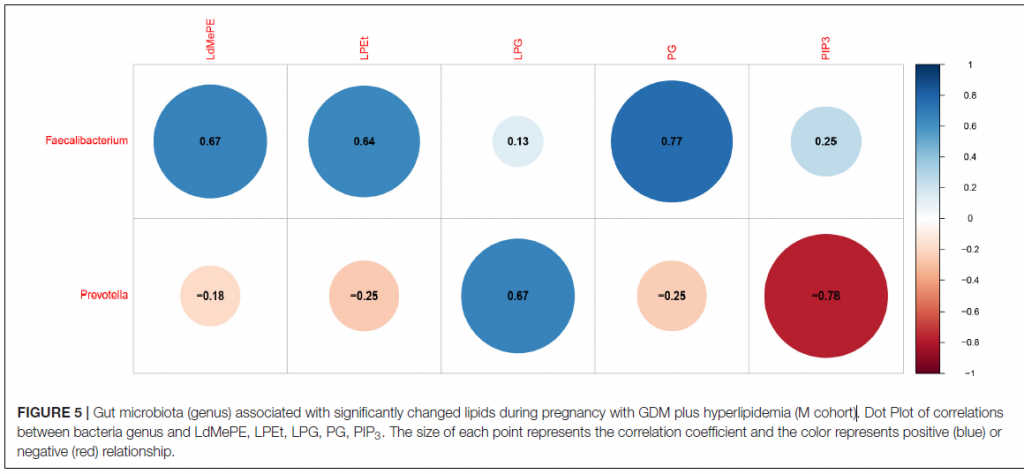


研究结论:我们的结果表明,血脂水平可能反映了GDM发展过程中的一些异常变化。所鉴定的多种生物标志物对GDM合并高脂血症的防治有一定的参考价值。

组间物种差异性箱形图
组间物种差异性盒形图描述在不同分组之间具有差异显著的某一物种做盒形图,图中以属水平为例做物种差异性盒形图,展示如下:

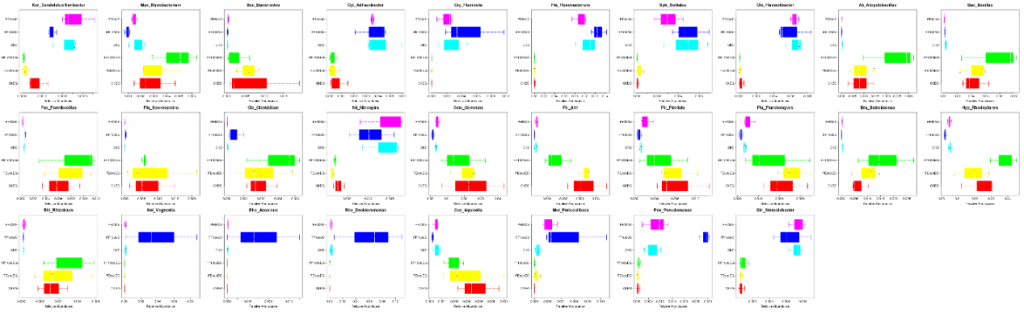

○ 图中不同颜色代表不同的分组,更直观显示组间物种差异
○ 每一个盒形图代表一个物种,图上方是物种名。
Anosim检验
Anosim分析是一种非参数检验,用来检验组间的差异是否显著大于组内差异,从而判断分组是否有意义
展示如下:
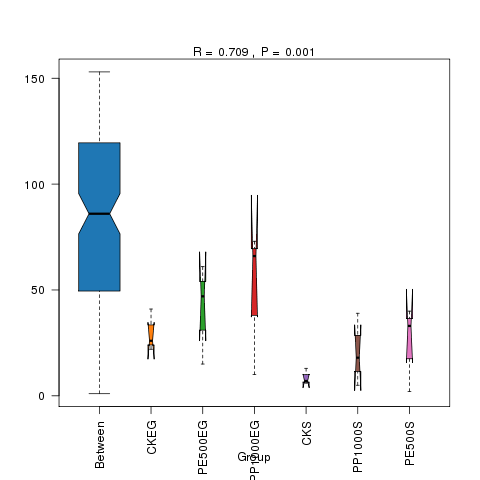


R-value介于(-1,1)之间,R-value大于0,说明组间差异显著。
R-value小于0,说明组内差异大于组间差异。
统计分析的可信度用 P-value 表示,P< 0.05 表示统计具有显著性。
对Anosim的分析结果,基于两两样本之间的距离值排序获得的秩(组间的为between,组内的为within),这样任一两两组的比较可以获得三个分类的数据,并进行箱线图的展示(若两个箱的凹槽互不重叠,则表明它们的中位数有显著差异)
随机森林分类树属分类效果
随机森林是机器学习算法的一种,它可以被看作是一个包含多个决策树的分类器。
其输出的分类结果是由每棵决策树“投票”的结果。由于每棵树在构建过程中都采用了随机变量和随机抽样的方法,因此随机森林的分类结果具有较高的准确度,并且不需要“减枝”来减少过拟合现象。
随机森林可以有效的对分组样品进行分类和预测。
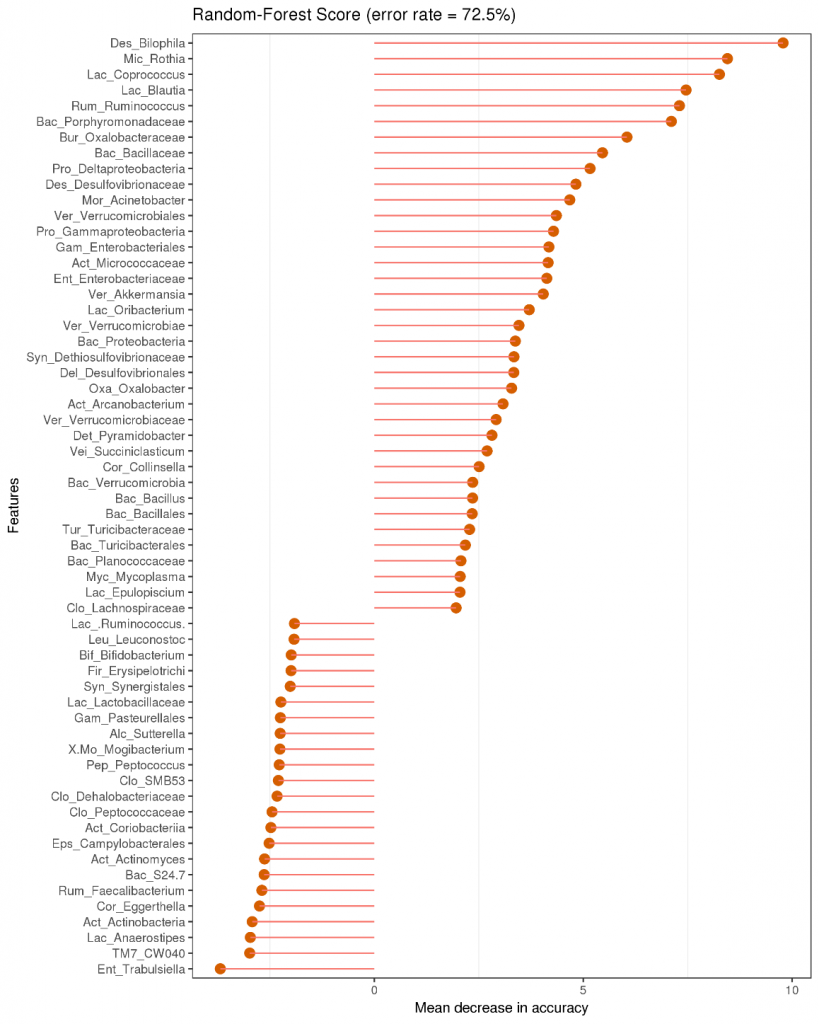

物种重要性点图。横坐标为重要性水平,纵坐标为按照重要性排序后的物种名称。上图反映了分类器中对分类效果起主要作用的菌属,按作用从大到小排列。
Error rate: 表示使用下方的特征进行随机森林方法预测分类的错误率,越高表示基于菌属特征分类准确度不高,可能分组之间菌属特征不明显。图中以所有水平为例,取前60个作图。
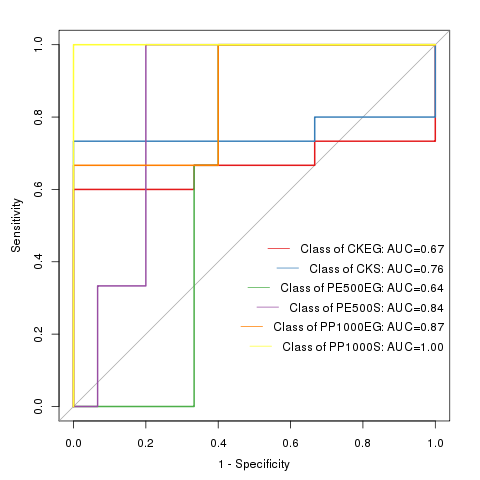
ROC曲线图
ROC 曲线指受试者工作特征曲线(receiver operating characteristic curve), 是反映敏感性和特异性连续变量的综合指标,通过构图法揭示敏感性和特异性的相互关系。
ROC 曲线将连续变量设定出多个不同的临界值,从而计算出一系列敏感性和特异性,再以敏感性为纵坐标、(1-特异性)为横坐标绘制成曲线。
曲线下面积越大,诊断准确性越高。展示如下:

FAPROTAX生态功能预测
FAPROTAX是一款在2016年发表在SCIENCE上的较新的基于16S测序的功能预测软件。它整合了多个已发表的可培养菌文章的手动整理的原核功能数据库,数据库包含超过4600个物种的7600多个功能注释信息,这些信息共分为80多个功能分组,其中包括如硝酸盐呼吸、产甲烷、发酵、植物病原等。
FAPROTAX对环境样本更友好
如果说PICRUSt(后续会介绍)在肠道微生物研究更为适合,那么FAPROTAX尤其适用于生态环境研究,特别是地球化学物质循环分析。
FAPROTAX适用于对环境样本(如海洋、湖泊等)的生物地球化学循环过程(特别是碳、氢、氮、磷、硫等元素循环)进行功能注释预测。因其基于已发表验证的可培养菌文献,其预测准确度可能较好,但相比于上述PICRUSt和Tax4Fun来说预测的覆盖度可能会降低。
FAPROTAX可根据16S序列的分类注释结果对微生物群落功能(特别是生物地化循环相关)进行注释预测。
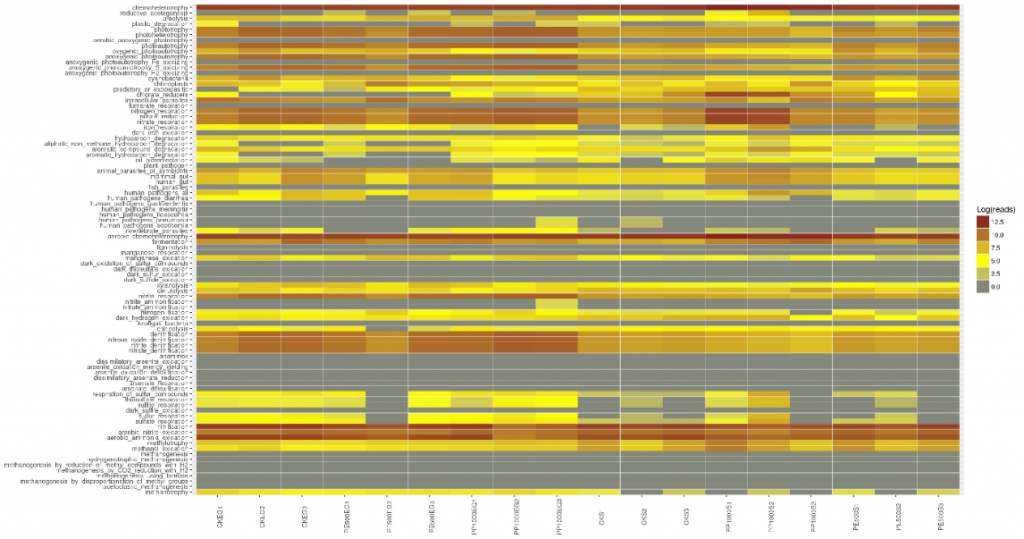

图中横坐标代表样本,纵坐标表示包括碳、氢、氮、硫等元素循环相关及其他诸多功能分组。可快速用于评估样品来源或特征。
基于BugBase的表型分类比较
Bugbase也是16年所提供服务的一款免费在线16S功能预测工具,到今年才发表文章公布其软件原理。该工具主要进行表型预测,其中表型类型包括革兰氏阳性、革兰氏阴性、生物膜形成、致病性、移动元件、氧需求,包括厌氧菌、好氧菌、兼性菌)及氧化胁迫耐受等7类。
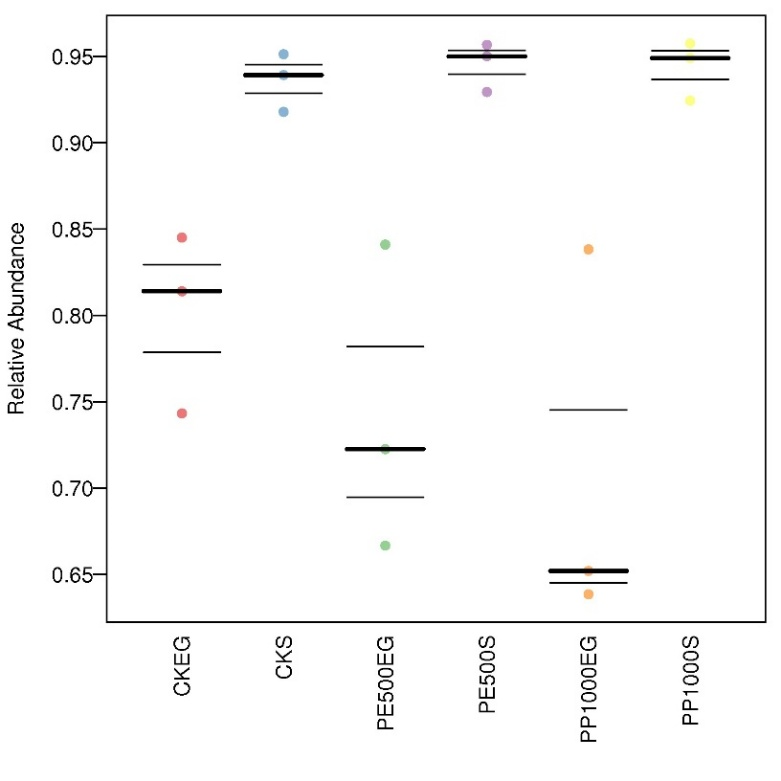

Gram Negative 革兰氏阴性菌
Picrust群落功能差异分析
通过对已有测序微生物基因组的基因功能的构成进行分析后,我们可以通过16s测序获得的物种构成推测样本中的功能基因的构成,从而分析不同样本和分组之间在功能上的差异(PICRUSt Nature Biotechnology, 1-10. 8 2013)。
Picrust对肠道菌群样本更友好
通过对宏基因组测序数据功能分析和对应16s预测功能分析结果的比较发现,此方法的准确性在84%-95%,对肠道微生物菌群和土壤菌群的功能分析接近95%,能非常好的反映样品中的功能基因构成。
怎么做出来的?
为了能够通过16s测序数据来准确的预测出功能构成,首先需要对原始16s测序数据的种属数量进行标准化,因为不同的种属菌包含的16s拷贝数不相同。
然后将16s的种属构成信息通过构建好的已测序基因组的种属功能基因构成表映射获得预测的功能结果。(根据属这个水平,对不同样本间的物种丰度进行显著性差异两两检验,我们这里的检验方法使用STAMP中的two-sample中T-TEST方法,Pvalue值过滤为0.05,作Extent error bar图。)
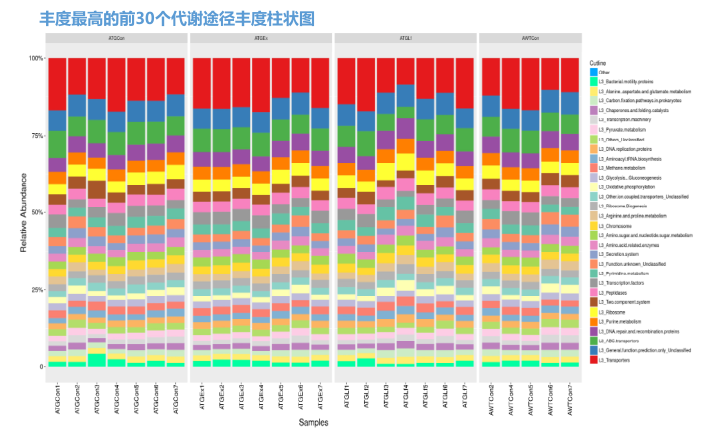

此处提供COG,KO基因预测以及KEGG代谢途径预测。当然,跃跃欲试的小伙伴也可自行使用我们提供的文件和软件(STAMP)对不同层级以及不同分组之间进行统计分析和制图,以及选择不同的统计方法和显著性水平。
这里提到的STAMP有些小伙伴说不太了解,别急,后面会有更多介绍。
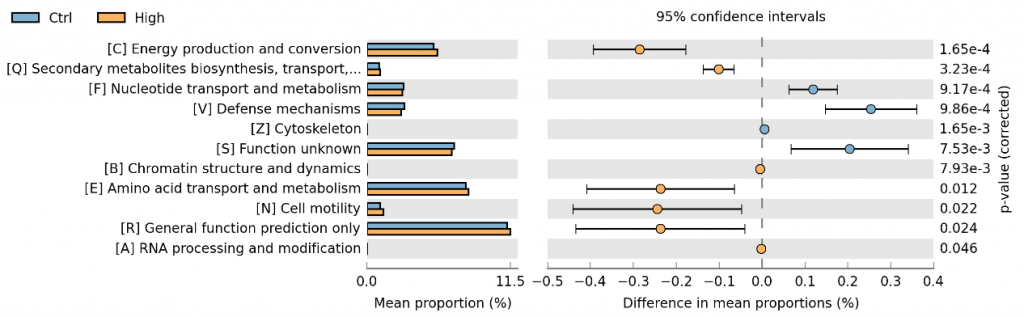
COG构成差异分析图
图中不同颜色代表不同的分组,列出了COG构成在组间存在显著差异的功能分类以及在各组的比例,此外右侧还给出了差异的比例和置信区间以及P-value。

KEGG代谢途径差异分析图
通过KEGG代谢途径的预测差异分析,我们可以了解到不同分组的样品之间在微生物群落的功能基因在代谢途径上的差异,以及变化的高低。为我们了解群落样本的环境适应变化的代谢过程提供一种简便快捷的方法。
本例图所显示的是第三层级的KEGG代谢途径的差异分析,也可以针对第二或第一层的分级进行分析。
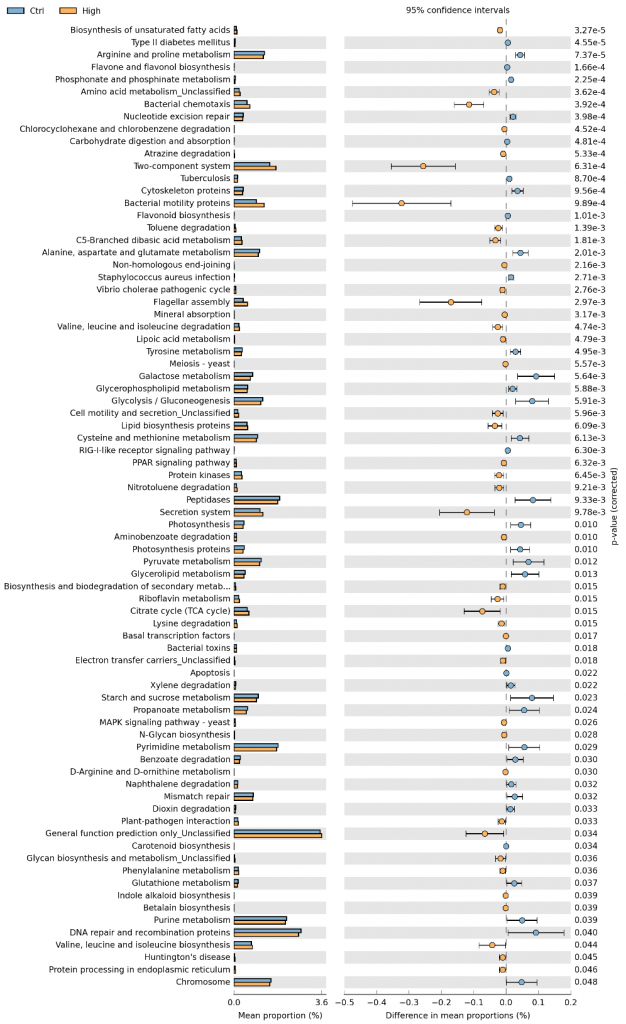

图中不同颜色代表不同的分组,列出了在第三层级的构成在组间存在显著差异的KEGG代谢途径第三层分类以及在各组的比例,此外右侧还给出了差异的比例和置信区间以及P-value。
案例解析




研究背景:尽管普遍认为肠道微生物组的生态多样性和分类组成在肥胖和T2D中发生改变,但与单个微生物或微生物产物的关联在研究之间不一致。缺乏大样本群体研究,从而确定肠道微生物组,血浆代谢组,肥胖和糖尿病表型以及环境因素之间的几种关联。
主要图表:
按照肥胖和糖尿病对人群分为三组,同时进行了16S,代谢和宏基因组的检测。

与肥胖相关的菌属以及代谢途径
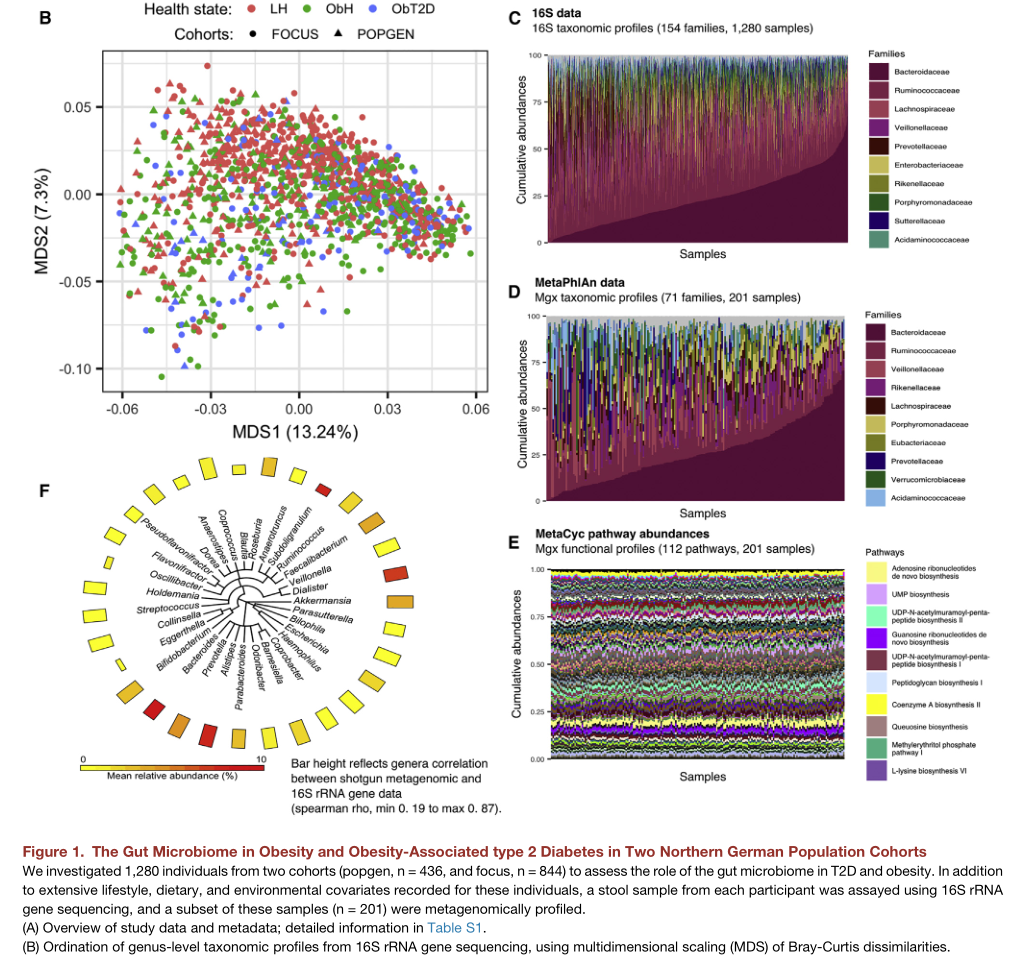

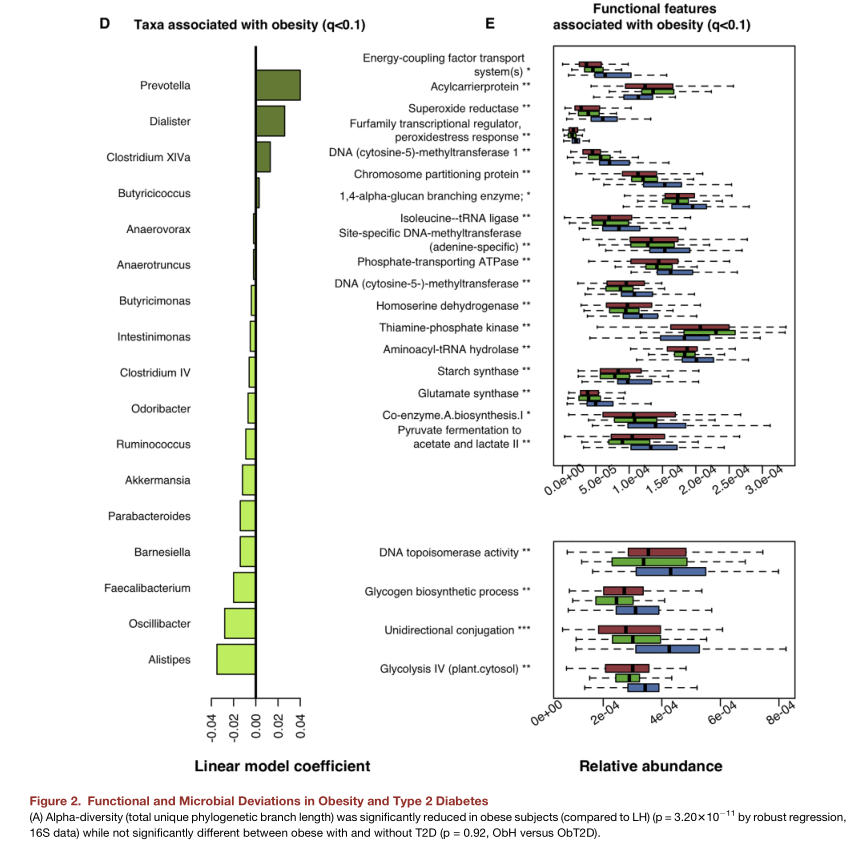

研究结论:确定了肠道微生物组,血浆代谢组,肥胖和糖尿病表型以及环境因素之间的几种关联。与肠道微生物组变异相关的主要是肥胖,不是2型糖尿病。存在与肠道微生物组变异相关的药物和膳食补充剂。高铁摄入量影响小鼠的肠道微生物组成。微生物组变异也反映在血清代谢物谱中。
点评:
相对大人群的队列研究,同时涵盖了菌群、代谢和疾病表型以及膳食补充调查的数据。
从结果看菌属和血浆代谢存在关联,但是贡献度都较低,如果样本数量不足很可能找不到显著的联系,这也是这类大样本队列研究的意义。
本研究在人群分组时针对性的研究了肥胖-II型糖尿病和菌群的关联,因而构建了三个主要分组人群,结果显示肥胖与菌群的关联度更大,解释了大部分的菌群差异,而糖尿病的菌群变化较小。
本研究其中较为重要的是发现了不同膳食补充对菌群的影响,并在小鼠实验中得到证实。

基因的差异分析图
除了能对大的基因功能分类和代谢途径进行预测外,我们还能提供精细的功能基因的数量和构成的预测,以及进行样本间以及组间的差异分析,并给出具有统计意义和置信区间的分析结果。
这一分析将我们对于样本群落的差异进一步深入到了每一类基因的层面。


图中不同颜色代表不同的分组,列出了在组间/样本间存在显著差异的每一个功能基因(酶)以及在各组的比例,此外右侧还给出了差异的比例和置信区间以及P-value。
很多小伙伴总希望能亲自上手做点分析,机会来了!
在获得标准报告后如果希望单独修改分组或对某些组之间进行显著性差异分析,可以使用STAMP软件在自己的电脑上进行数据分析。STAMP提供了丰富的统计检验方法和图形化结果的输出。
在使用STAMP之前需要首先准备需要的spf格式文件和样品分组信息表,但是如果数据不会处理,那也很不便。
而在我们的报告中已经将KEGG和KO以及COG的结果文件后经过转换生成了适用于STAMP软件打开的spf格式文件,还有对应的分组信息表文件groupfile.txt。
使用STAMP时的一些相关问题
1、STAMP作图用的原始数据的来源?
STAMP 可以直接使用来自QIIME的biom文件和PICUST的KEGG和ko 文件,groupfile.txt文件的格式为tab-saperated value (tab键隔开的数据)
2、分组问题?
导入数据之后,viewàgroup legend ,在窗口右侧会出现分组栏,根据需要进行分组。
3、Unclassiffied选项中,remain Unclassiffied reads、remove Unclassiffied reads、和use only for calculating frequency profiles 方法的区别?
remain Unclassiffied reads和use only for calculating frequency profiles方法会保留所有的数据,而remove Unclassiffied reads仅仅保留有确定分组信息的数据。
4、Statistical test 中,Welch’s t-test、t-test、white’s non-parametric t-test的区别,各自优缺点?
为了确保统计学意义和准确度和精确性,需要足够多的样本数目,t-test检验可以在最少样本数为4的时候确保高的准确度和精确性。
当两个样本之间具有相同方差的时候,用t-test更为准确,当两个样本没有相同方差,Welch’s t-test更为准确。
当样本数目少于8的时候,可以使用white’s non-parametric t-test,该计算时间较长,当样本数目过多的时候不宜使用该方法。
5、Two-group 中 type: one side和two side的区别?
One side 只会显示前一个group与后一个group差异的比例,而two side 两者之间的比例均会显示。
6、STAMP在使用时首先打开了一个分析文件,如果新打开一个可能会导致显示错误?
目前版本的STAMP存在一些小问题,一次分析只能使用一个数据文件,如果要打开新的需要关闭软件后再打开。
详细的STAMP使用教程可以参考我们提供的STAMP使用教程。
环境因子分析
冗余分析(redundancy analysis, RDA)或者
典范对应分析(canonical correspondence analysis, CCA)都是基于对应分析发展的一种排序方法,将对应分析与多元回归分析相结合,每一步计算均与环境因子进行回归,又称多元直接梯度分析。主要用来反映菌群与环境因子之间的关系。
RDA 是基于线性模型,CCA是基于单峰模型。分析可以检测环境因子、样品、菌群三者之间的关系或者两两之间的关系。
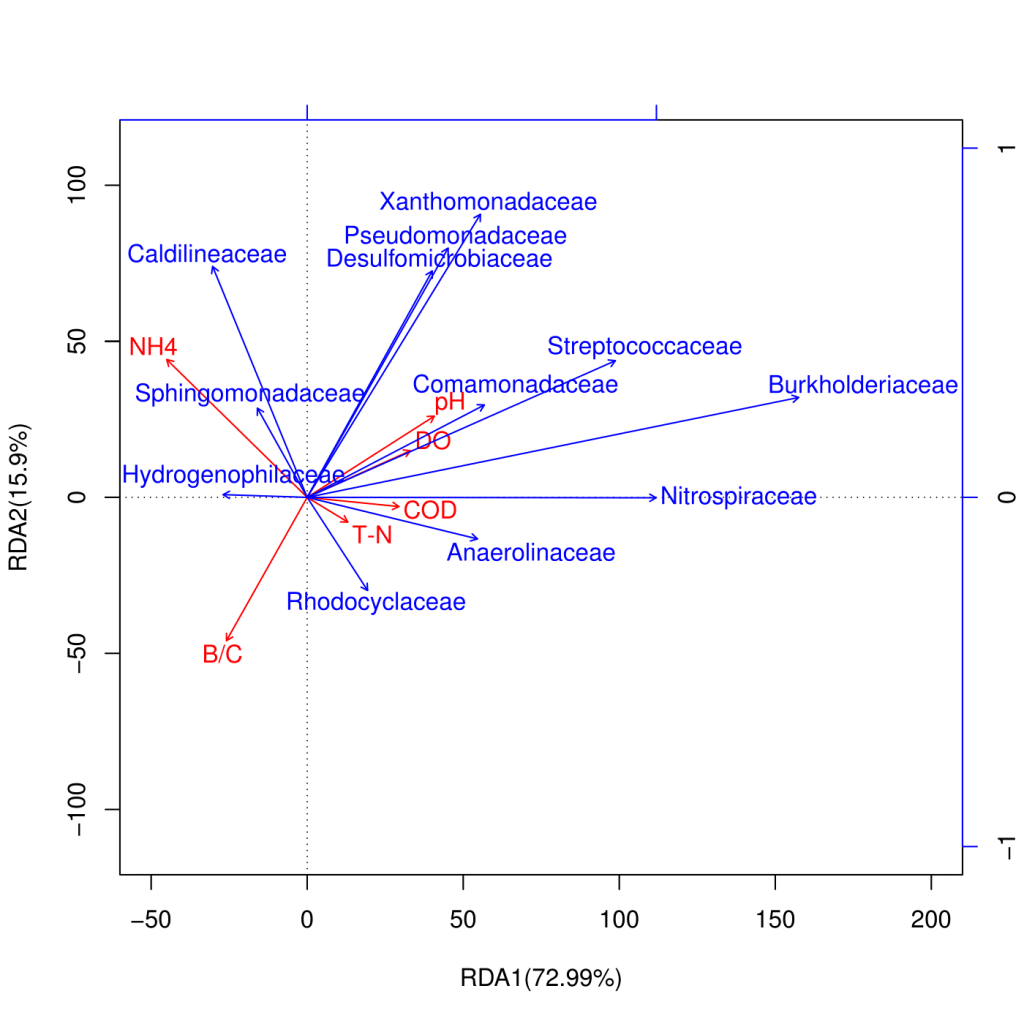

○ 冗余分析可以基于所有样品的OTU作图,也可以基于样品中优势物种作图;
○ 箭头射线:箭头分别代表不同的环境因子;
○ 夹角:环境因子之间的夹角为锐角时表示两个环境因子之间呈正相关关系,钝角时呈负相关关系。环境因子的射线越长,说明该影响因子的影响程度越大;
○ 不同颜色的点表示不同组别的样品或者同一组别不同时期的样品,图中的拉丁文代表物种名称,可以将关注的优势物种也纳入图中;
○ 环境因子数量要少于样本数量,同时在分析时,需要提供环境因子的数据,比如 pH值,测定的温度值等。
个性化图表

除以上部分,还可以进行个性化图表定制,像下面这样:

看完以上内容,也许还有不明白的地方,没关系,我们罗列了一些常见的问题。看看有没有你想问的。
答疑小课堂
Q1
原始数据形式以及数据如何上传?
原始fastq格式是一个文本格式用于存储生物序列(通常是核酸序列)和其测序对应的质量值。这些序列以及质量信息用ASCII字符标识。通常fastq文件中一个序列有4行信息:如
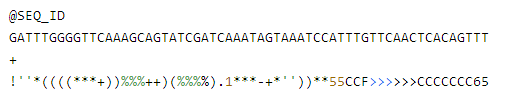

第一行:序列标识,以 @开头。格式自由,允许添加描述信息,描述信息以空格分开。
第二行:序列信息,不允许出现空格或制表符。一般是明确的DNA或RNA字符,通常大写
第三行:用于将序列信息和质量值分隔开。以 +开头,后边是描述信息或者不加。
第四行:质量值, 每个字符与第二行的碱基一一对应,按照一定规则转换为碱基质量得分。进而反映该碱基的错误率,因此字符数必须和第二行保持一致。
Fasta格式
fasta是一种基于文本用于表示核苷酸序列或氨基酸序列的格式。在这种格式中碱基对或氨基酸用单个字母来编码,且允许在序列前添加序列名及注释。由两部分信息组成:如
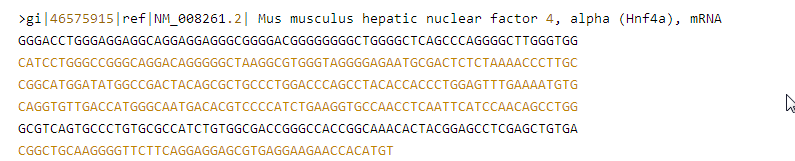

第一行:序列标记,以 >开头,接序列的标识符,序列标识符以空格结束,后接描述信息。为保证分析软件能区分每条序列,每个序列的标识必须具有唯一性。
第二行:序列信息,使用既定的核苷酸或氨基酸编码符号。
数据提交
原始数据(Raw data),常见的是illumina机器产生的fastq文件,这一类文件需要向NCBI的SRA数据库进行提交,SRA是NCBI为了并行测序的高通量数据(massively parallel sequencing)提供的存储平台。完整提交SRA需要一些独立项目的分步提交,包括BioProject、BioSample、Experiment、Run等,每一部分用以描述数据的不同属性。
Q2
如何判断测序质量是否合格?
原始的Tags数据会经过质控、过滤、去嵌合体,最终得到有效数据(Effective Tags)。所以在判断测序质量是否合格时应该从几个方面去判断。
打开文件01_sequence_statistic/sumOTUPerSample.txt
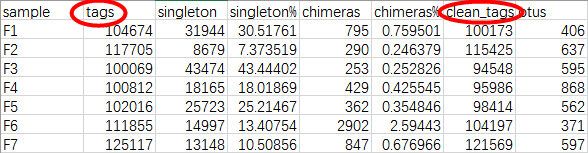


报告里所有的txt打开如果格式不对的话,可以用excel表打开。
其中tags为经质量过滤后能正确overlap包含正确barcode和高质量序列的数据。
Singleton为非完全相同的序列,只要有1个碱基的差异即为不同序列,该值的高低与OUT数量并无直接关系,OTU是以97%的相似度聚类,测序质量较低导致的碱基错误、PCR扩增过程中的碱基错误、菌种内部的多样性以及OTU数量均会影响该数量。
Chimeras为通过与RDP等标准数据库比对分析判断可能由于PCR过程错误扩增导致的嵌合体比例,chimeras%为百分比,一般低于1。
首先判断下机数据tags和有效数据 clean tags 的数据量是否满足测序要求,一般下机数据量达到3万条reads以上满足测序需要,谷禾16s样本的测序深度可以达到10万条reads左右。如果数据量不够则需要重新补测样本。通过观察嵌合体数chimras 和嵌合体所占百分比chimeras%,可以反应出有效序列的转化率,嵌合体的比例越小序列的利用转化率就越高。
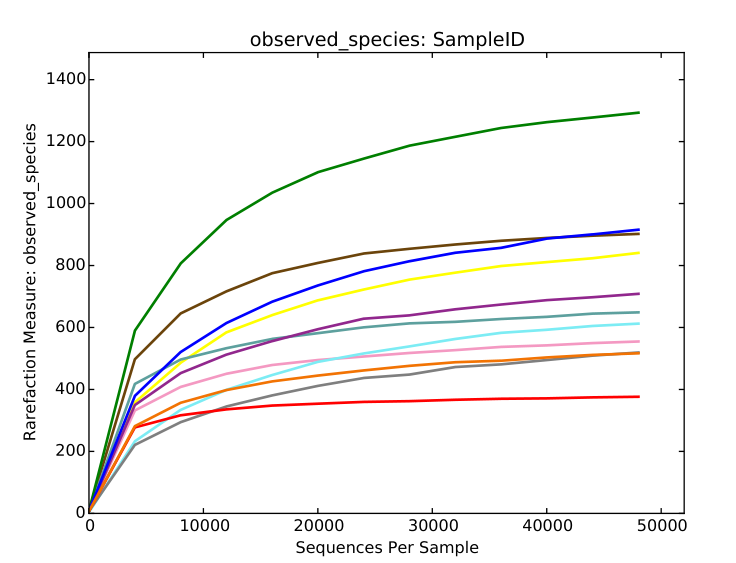

根据稀释曲线可以判断测序深度是否达到饱和,如图中曲线都逐渐趋于平缓,就证明样本的测序深度较好,测序深度基本覆盖能测到的该样本所有的物种,测序深度比较好。同时曲线趋于水平纵坐标的高低也能够反映各样本的微生物多样性情况,曲线越高,证明测到的物种种类越多,样本的微生物多样性就越高。
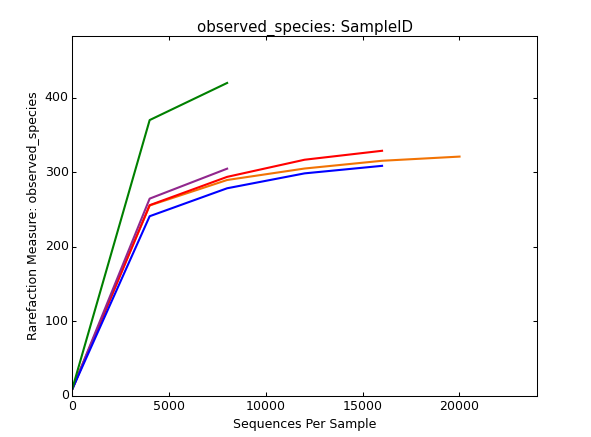

而从该图可以看出,个别样本的曲线未趋于平缓,证明该样本测序深度不够,测序深度未能很好的反映出该样本的完整菌群构成。如果测序数据量更大的的话会检测到更多物种。
Q3
如何了解分组内部的多个样本的重复性以及多样性情况?
观察分组内部多个样本的重复性如何可以从以下几个方面考虑。
首先在各分类水平的柱状图的菌属构成来看
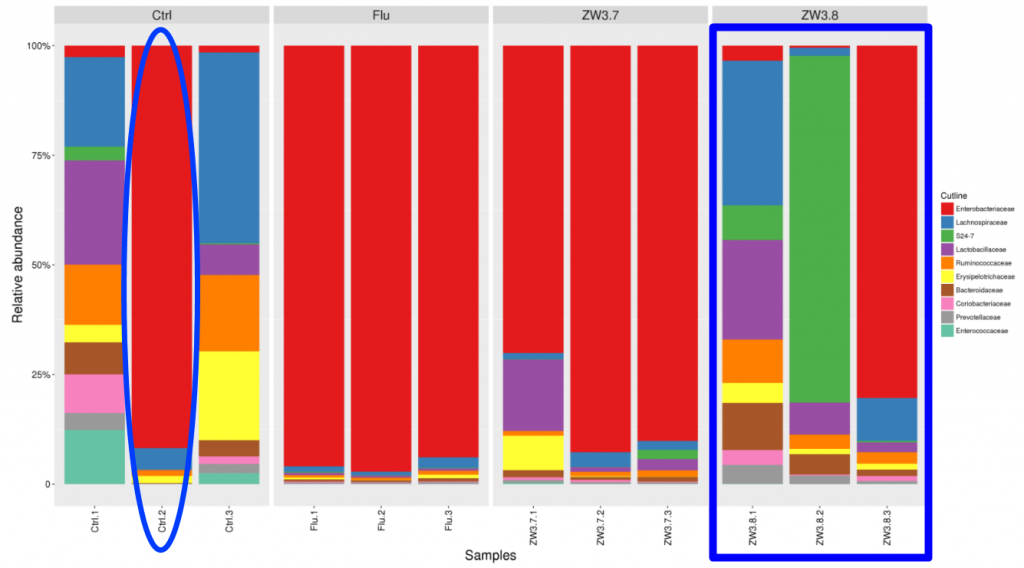

从构成图来看,Flu组和ZW3.7组,组内样本重复性较好。Ctrl组中Ctrl.2明显区别于组内另外两个样本,可以去掉该样本。而ZW3.8组内样本间差异性较大。
比如人体肠道或小鼠肠道样本本身个体差异性较大,菌群结构组成复杂,即便通过不同疾病的分类的样本,但营养饮食、代谢以及环境的影响都会改变肠道菌群的构成,所以有可能组内样本间差异性会比较大。而经过单因素处理的样本组内差异会比较小。
所以在前期实验设计时,尽量选择同一批次相同处理的小鼠或其他样本,避免组内差异的影响。并且要预留好多余的样本,比如组内只有3个样本,如果去掉一个差异性较大的样本,一个分组内只有2个样本,会影响后续组间差异比较,组间差异性比较分析每组要至少要3个样本。
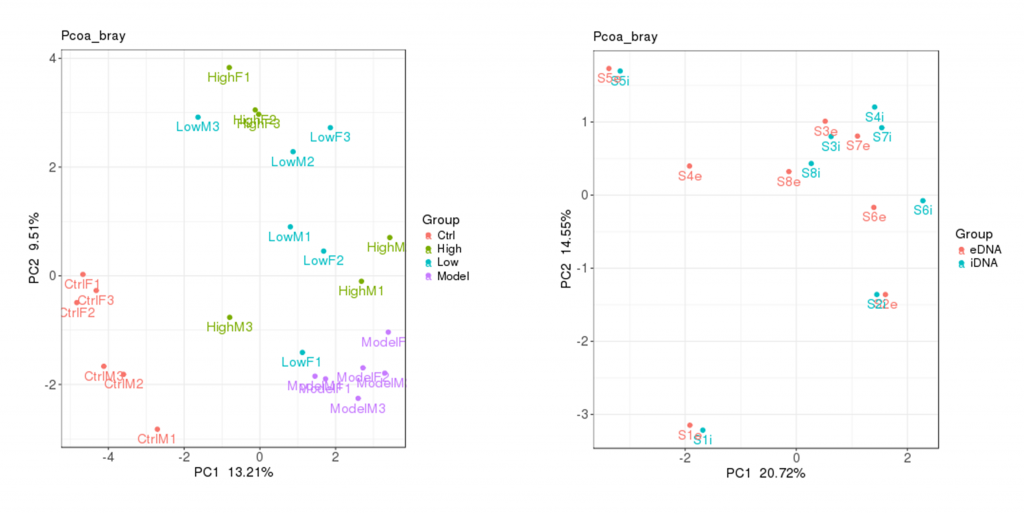

通过beta多样性分析PCA,PCoA,MNDS 也可以大致观察组内样本重复性情况,左图组内样本重复性较好,右图组内样本间差异性较大,两组间的区割不是很明显。
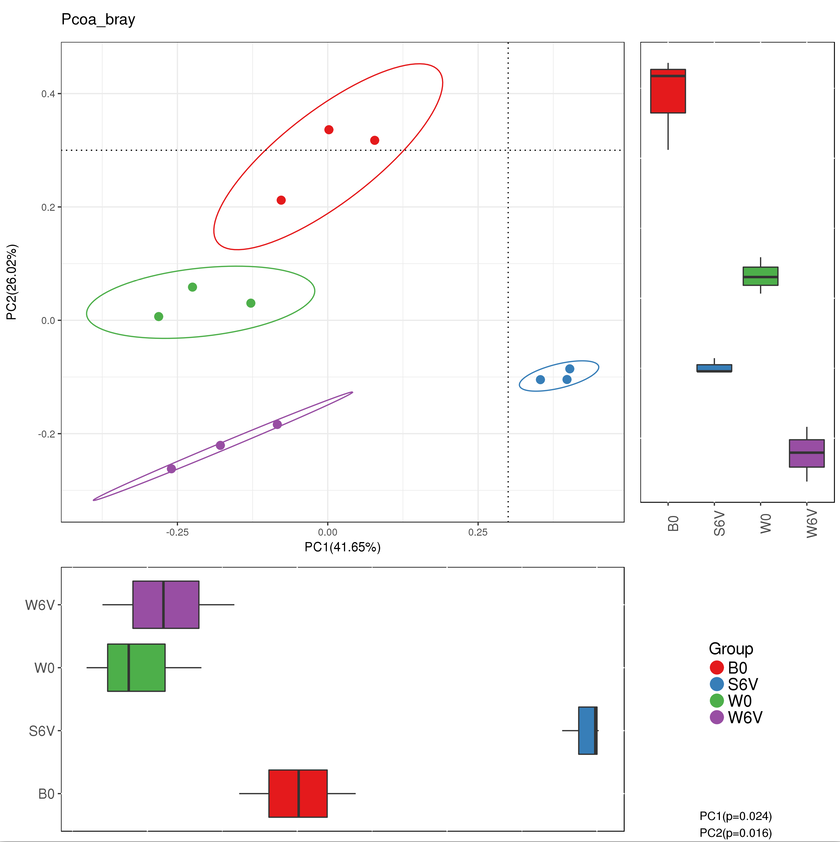


在加圈图的beta多样性分析中,右下角有给出PC1和PC2的P值,小于0.05则差异显著。
Alpha多样性是针对单个样品中物种多样性的分析,包括chao1指数、ace指数,shannon指数以及simpson指数等。前面4个指数越大,最后一个指数越小,说明样品中的物种越丰富。
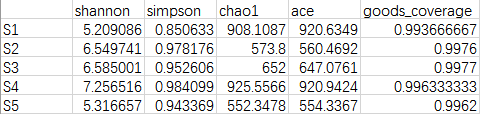

其中chao指数和ACE指数反映样品中群落的丰富度(species richness),即简单指群落中物种的数量,而不考虑群落中每个物种的丰度情况。指数对应的稀释曲线还可以反映样品测序量是否足够。如果曲线趋于平缓或者达到平台期时也就可以认为测序深度已经基本覆盖到样品中所有的物种;反之,则表示样品中物种多样性较高,还存在较多未被测序检测到的物种。
而shannon指数以及simpson指数反映群落的多样性(species diversity),受样品群落中物种丰富度(species richness)和物种均匀度(species evenness)的影响。相同物种丰富度的情况下,群落中各物种具有越大的均匀度,则认为群落具有越大的多样性。
稀释曲线是利用已测得序列中已知的各种OTU的相对比例,来计算抽取n个(n小于测得Reads序列总数)Tags时各Alpha指数的期望值,然后根据一组n值(一般为一组小于总序列数的等差数列,本项目公差为500 )与其相对应的Alpha指数的期望值绘制曲线。
Q4
不同的样本之间差异大吗?不同分组之间能否用菌群差异来区分?
观察不同分组间差异的大小可以观察随机森林分类效果图。
路径在07_diff_analysis/RF
图中以该分类水平下选取用于区分不同分组间的差异性起到关键性影响因素的物种作为标志物作图。标志物按重要性从大到小排列,图中随机森林值error rate 表示用随机森林方法预测分组之间的错误率,分值越高代表所选取的标志物准确度不高,并不能很好的用于区分各分组,分组差异不显著。分值越低证明分组效果比较好。
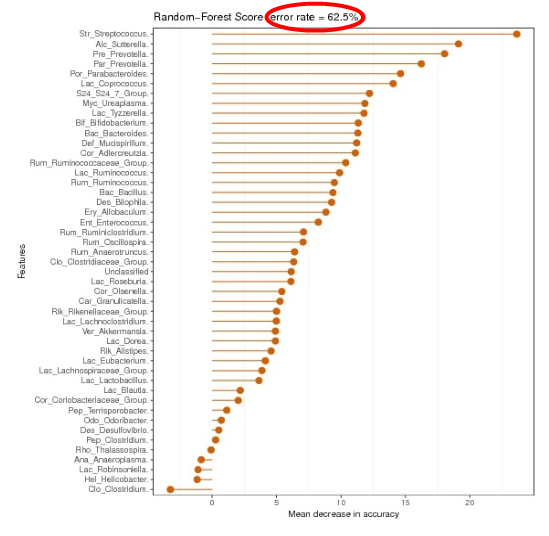

上图中的随机森林按照门和属以及代谢途径分别进行分析作图,各自都有单独文件,报告中仅给出了一个图,其他文件需要到目录中查看。可能存在门或属区分效果不佳,但是代谢途径区分效果较好。
随机森林筛选出来的物种是用于区分所有分组的重要标志。分值越高代表该物种用于区分所有组之间的重要性越大。
Q5
二代测序16s 能用普通酶扩增吗?
16s测序主要为了鉴定菌种,通常在做鉴定的时候区分标准是97%,区分亚种和菌株的时候相似度更高。
普通TAQ酶的复制错误率较高,可能在扩增过程中引入错误,这些错配可能导致相似度下降从而分类错误。
一般我们不建议使用普通TAQ酶进行扩增,都选择高保真酶。
Q6
利用16s rRNA鉴定细菌能确定到种上吗?
16s rRNA长度为1.5k多,作为菌种鉴定一般选择相似度97%的标准,相似度超过97%一般定义为同一种菌。
如果是sanger测序获得16s全长的都可以鉴定到种,甚至能区分亚种。有些细菌并不只有1个16s序列,会包含有1-15拷贝的16s序列,所以单一的16s序列鉴定可能会出现偏差。
利用高通量如454或miseq测序一般由于读长的缘故,通常只有300-500多个碱基被测序,所以在物种鉴定上一般比较可靠的是能分类到属,部分能分类到种。
根据我们的经验,不同的样品会有大约10-50的菌能分类到种。利用新的分析方法,我们现在也可以利用16s rRNA的群落多样性高通测序数据进行亚种级别的分析。主要是利用16s中共同变化的SNP位点进行分型。这样可以大大提高菌种的分类精度,尤其是在有些菌株之间表型差异巨大的时候。
Q7
听说光测16s就可能预测基因和功能,是真的吗?
16s序列能够区分菌的种属,但是并不包含这些菌的基因和代谢功能的信息。不过由于我们已经对大量的细菌基因组进行了测序,所以可以根据16s的菌种信息,利用这个菌属已经测序的细菌基因组的基因信息和代谢功能信息来估计每类基因的上限和下限。
所以答案是可以利用16s序列测序来预测菌群的功能基因分布和代谢途径分布情况。
目前主要使用的软件是PICRUSt和新发表的Tax4Fun。
从我们实际分析和实验结果来看,预测的准确性还是很高的,不过和样品有很大关系。像肠道菌群和土壤以及一些致病菌的测序较多,所以预测的准确度较高可以到85-90%以上。一些海洋的菌由于测序的菌较少,预测准确性要差一些。目前发表的文献基本都是用PICRUSt,新的软件还有待验证。
Q8
测16s rRNA能分到亚种吗?不同菌株都有致病性差异光到种不解决问题啊!
16s rRNA如果是使用sanger测序可以细分到亚种甚至有些可以精确区分菌株,但是要看菌种。
如果是高通量测序,目前的常见分析一般以97%为标准,大部分情况只能到属,少部分能区分到种。如果要进一步细分到亚种甚至更小的区分目前是有可能的,我们在使用oligotype一类的方法时可以将相同变化模式的SNP归类,并对原来的OTU进行进一步细分,理论上可以区分到菌株。
不过这种区分不同菌属差异很大,有些可以很理想的区分,主要用来了解在更细分化尺度上菌株构成的地理和时间变化。
仅通过16s高通量测序恐怕不能完全解决菌株致病性差异这种问题,但是通过对常见OTU的进一步深入分析可以提供可能的解释或方向。如果明确了某一特定类型菌株的变化有关,可以采用比如毒力基因或菌株特异性标记等方法详细了解不同菌株的比例和差异。

多项合作成果发表于Nature communications、PNAS、Plant biotechnology journal、DNA Research、Environmental Science & Technology、Plant、cell & environment、Science of The Total Environment 、Gut Microbes 、Frontiers in microbiologyt、Journal of environmental management 等国际著名学术期刊。
近期发表文章目录