-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康

生态学家在分析微生物组和感兴趣的协变量(如临床结果或环境因素)之间的关联时,经常以两种方式查看物种分类计数数据。
一种是将计数视为定量的(即作为相对丰度数据进行分析);另一种是将计数数据离散化,只表明一个分类单元在样本中是否存在。
虽然第一种方法在医学文献中可能更常见,但这种关联也可能是由于样本中存在或不存在的分类群的变化所驱动的。例如,在人类肠道中,物种丰富度的增加与更稳定的生态系统有关,生态系统往往对饮食、抗生素使用和病原体入侵等环境压力更具有抵抗力。
相比之下,健康的阴道微生物群的特点往往是以Lactobacilli为主的低多样性。研究人员认为当常见的分类群占主导地位时,基于相对丰度的分析可能更合理,而当稀有分类群占主导地位时,基于存在或不存在的分析可能表现更好。
目前最常用的分析方法是基于未加权的UniFrac和Jaccard距离的PERMANOVA统计检验,但是它只给出了整个群落的关联,没有给出与单个分类群的关联。Fisher’s精确检验可以检验协变量与单个分类群是否存在关联,对于更复杂的情况,可以使用精确的逻辑回归,但计算成本较高。
LDM本身是一种线性模型,研究人员对它进行了扩展,使得该方法可以基于稀疏计数数据,来分析协变量与群落或单个分类群之间的关联,同时控制混杂协变量(例如实验中出现的提取和扩增偏差),协变量可以是离散的也可以是连续的。
在LDM中,元数据被放入矩阵X中(行对应N个样本,列对应协变量)。将X的列分为K组,这里可以理解为”子模型“,每个子模型代表一组想要联合检验的变量。
LDM使X的列正交。设Y为N×J(原始)分类群计数表,共有J个分类群。
Hk为子模型的帽子矩阵
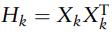
H0为完整模型X的帽子矩阵
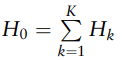
通过计算完整模型的帽子矩阵可以得到在整个模型下的预测值。帽子矩阵可以用来计算残差,即实际观测值与模型预测值之间的差异。
同时,帽子矩阵也可以用来评估模型的拟合优度,例如通过计算决定系数(R-squared)来衡量模型对数据的解释程度。

研究人员表示由于LDM使用排列法评估显著性,在计算中没有考虑通常涉及自由度的乘法因子,并对通常的F-statisc加1。因此,LDM的检验统计量是在排除子模型k的模型中计算的残差平方和(RSS)与包含子模型k的模型中计算的RSS之比。
Fkj指所有分类群上特定于分类群的检验统计量的比值之和。总而言之,LDM使用残差来衡量子模型的效应,然后计算F-statisc来判断子模型是否对分类群产生显著影响,计算公式:
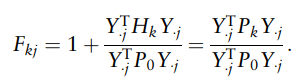

其中B的元素根据Y是否大于0来确定,I是指示函数。然后使用上面的公式,将矩阵Y替换为矩阵B,计算F-statisc。
最后作者给出来两种方式来结合稀疏的数据,选择取平均值或比值,并通过对协变量进行排列来进行分类群水平和整个群落的检验。但是作者也表示选择平均值较为稳妥。
模拟数据由50个病例和50个对照构成,根据上呼吸道微生物组(URT)的856个物种的相对丰度进行了模拟。
为了模拟在病例中存在而在对照中不存在的物种,从中均匀的选择了100个物种,并与病例-对照状态相关联。同时,单独选择了另外100个物种与混杂因子相关联,这是一个二元变量,在对照组与病例组中的分布为7:3。其中与病例-对照状态、混杂因子相关联的物种可能会有重叠。
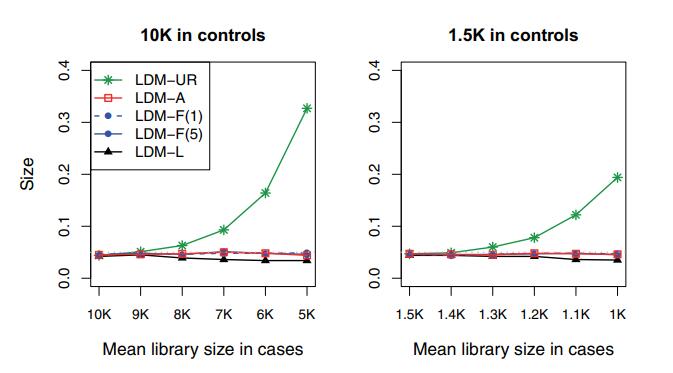
在模拟数据集中,作者比较了不同LDM使用方式的性能。
LDM-A作为基准,也是本篇文章的中心—一种扩展的LDM方法,它代表对所有稀释后的数据计算残差平方和(RSS)的平均值。
LDM-F(R),指通过对R次稀释后的数据计算平均F-statisc,这里的R在文中等于1或5。
LDM-UR,指对未稀释的矩阵(方法中的矩阵B)进行分析。
LDM-L,指在未稀释的数据中将库大小调整为一个协变量,并在LDM中应用。比较的结果基于10000次模拟重复实验。显著性水平定义为0.05。
结果如上图所示,LDM-UR的性能随着病例和对照之间的文库大小差异增加而变差,而其他方法都可以控制文库大小差异带来的影响。其中LDM-L的成功可能是因为文库大小本质上是一个二元变量,如果不是这个数据类型,LDM-L在更复杂的情况下可能表现不佳。
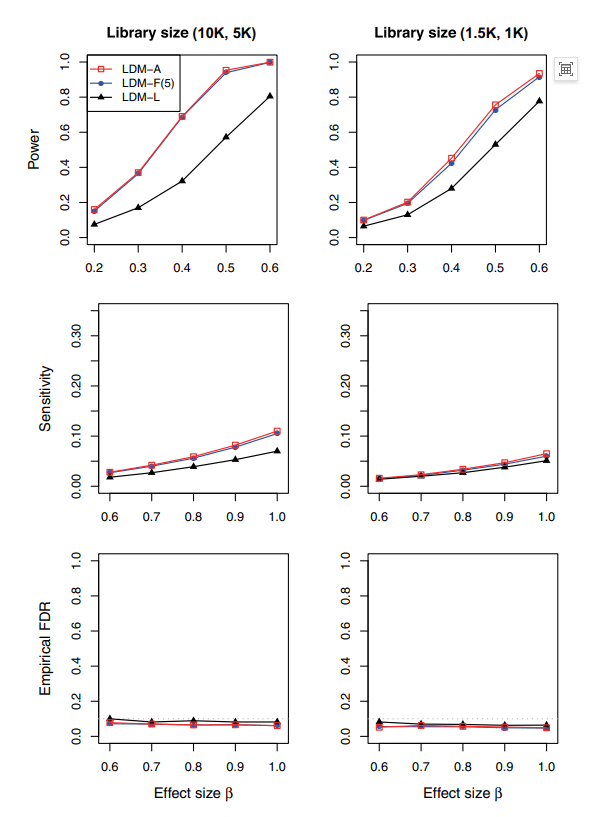
接着作者将平均文库大小统一固定为10k、5k和1.5k、1k,在此基础上比较LDM-A、LDM-F(5)和LDM-L的性能。
结果如下图所示,纵坐标为Power的图表示在整个群落中的表现能力,其中LDM-A表现最出色。
文中的Power指的是统计功效(statistical power)值,指在进行假设检验时,能够正确拒绝一个错误的零假设的概率。
纵坐标为sensitivity的图表示在分类群中,检验差异存在的敏感性,其中LDM-A具有最高的敏感性,相比之下,LDM-F(5)和LDM-L较低。
纵坐标为empirical FDR表示假阳性发现率,所有方法都在可接受范围内(<10%)。
编辑
虽然LDM-UR方法在控制文库大小差异影响表现最差,但作者发现LDM-UR对未稀释的数据进行分析是有效的,并且可以期望达它达到最佳的功效,因为它使用了全部reads。
★ 使用适当的稀释深度,功效损失将很小
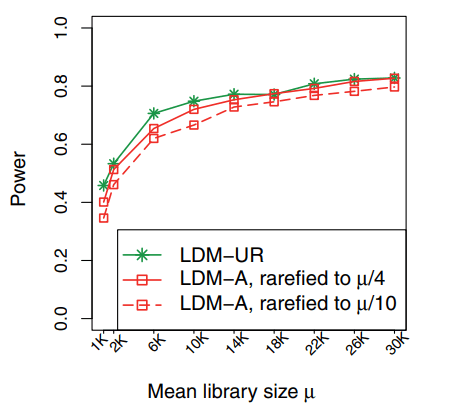
于是作者将LDM-A和LDM-UR进行了比较,给定两个稀释深度水平,即均值库大小的25%和10%(稀释深度也是数据中生成的最小库大小),观察当没有文库大小这类系统差异时,这两种LDM方法在不同稀释比例数据中的表现。
结果如下图所示,与分析完整数据的相比,稀释确实会导致功效损失,但随着均值库大小的增加,功效损失逐渐减小。
在较高的稀释深度下,当均值库大小超过一定阈值时,LDM-A的功效与LDM-UR相当。因此,考虑到现代测序技术产生的大型文库的大小,只要使用适当的稀释深度,预计功效损失将很小。
对来自RISK队列的数据子集进行了分析,该队列研究了新发炎性肠病(IBD)的儿童患者以及非IBD对照组。
作者选择了该数据子集中的来自直肠黏膜组织活检的数据。过滤掉了文库大小<10000的样本,相当于丢失了10%的样本。此外还过滤掉了在少于5个样本中存在的分类群。
最终留下267个样本的2565个分类群,共169个病例和98个对照。由于数据中男性比例存在不平衡(病例组为62%,对照组为44%),作者便将性别和抗生素使用作为需要控制的混杂因子,使用LDM检验直肠微生物组与IBD状态的存在-缺失关联。重点关注在群落水平上进行的关联分析,并检测对群落水平关联有显著贡献的个别分类群。
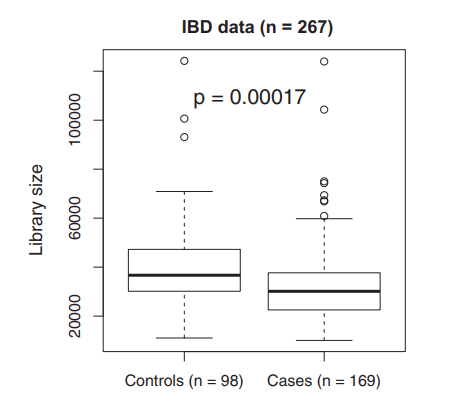
首先调查了所选数据中病例和对照组的文库大小分布,结果如下图所示,发现文库大小分布确实存在系统差异(同模拟数据)。因此对所有样本的读取计数数据进行了稀释,将测序深度稀释到最小值10081。
作者在文中提到在补充数据中,对去除性别和抗生素使用影响后的数据利用Jaccard距离绘制排序图,分别进行了无稀释和一次稀释的分析。
结果显示病例组与对照组在稀释前后都有明显的差异。且在没有稀释的情况下,两组差异更明显,这证实了文库大小的混杂效应。
接着作者对数据集应用LDM-A和LDM-F(R)方法,其中稀释次数R在1到20之间变化。分析结果如下图。
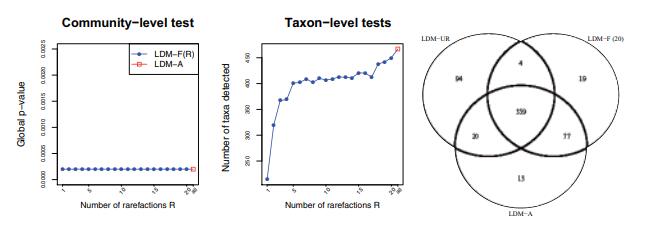
最左的图展示了在群落水平上,直肠微生物组与炎症性肠病状态存在-缺失关联分析的P值,这些P值表明,在群落水平上,炎症性肠病状态与直肠微生物组之间存在非常强的存在-缺失关联。
中间的图展示了在FDR为10%时,在病例和对照之间检测到差异的分类群数量,数据表明随着稀释次数从1增加到5,LDMF(R)检测到的分类群数量急剧增加,并在5次稀释后趋于稳定,这进一步证实数据至少需要进行5次稀释。
最右的韦恩图展示了LDM-UR、LDM-F(20)和LDM-A检测到的唯一或共同存在的分类群数目,数据显示,LDM-F(20)和LDM-A检测到的分类群集合有很大的重叠,而LDM-UR检测到的分类群集合中有很多(94个)与其他任何集合都不重叠,作者认为这些可能是由于文库大小混杂导致的假阳性结果。
编辑
作者在文中提出了两种有效的LDM扩展方法,LDM-A和LDM-F(R),它们分别使用不同的F-statisc计算方法。
LDM-A将分子和分母的残差平方和分别进行平均,取比值。而LDM-F(R)则对R次稀释重复的F-statisc进行平均。
根据测试结果,作者建议使用LDM-A而不是LDM-F(R),因为它有以下优点:(i) 不需要选择稀释重复次数;(ii) 在功效以及对于检测个别分类群的敏感性方面比LDM-F(R)更强;(iii) 计算效率更高。
注:作者已将该方法构建为R包,可使用’devtools::install_github(“yijuanhu/LDM”, build_vignettes=TRUE)’安装。
// Tips
在比这篇文章迟些发表的”A comprehensive evaluation of microbial differential abundance analysis methods: current status and potential solutions.”文章中提到,LDM方法虽然在统计方面有最好的功效,但在强成分效应存在的情况下,其对假阳性控制并不好。
该文章对多种差异分析方法做了比较评估。虽然它引用的文章是”Testing hypotheses about the microbiome using the linear decomposition model (LDM).”,这时的LDM方法还未进行上述的扩展。但我们仍然推荐阅读,以提供不同的视角。
参考文献:
Hu YJ, Lane A, Satten GA. A rarefaction-based extension of the LDM for testing presence-absence associations in the microbiome. Bioinformatics. 2021 Jul 19;37(12):1652-1657.

谷禾健康

病毒-细菌合并或继发感染
引起呼吸道感染的病毒是导致全世界高发病率和死亡率的原因,数十年来通常发生在冬季。在冬天,空气干燥,那些可能含有病毒的飞沫可以在空气中停留更长时间,并可以进一步传播。此外人的免疫力在冬季的表现也会不佳。因为纤毛是上呼吸道中微小的毛发状突起,通常将异物从鼻子中移出,但在较低的温度和较低的湿度下,纤毛的功能无法有效发挥作用。
在过去 20 年中,世界经历了六次重大传染病爆发(SARS-CoV:2002-2004 年;H1N1 流感:2009-2010 年;中东呼吸综合征冠状病毒:2012-2020 年;埃博拉病毒:2013-2016 年;寨卡病毒: 2015-2016 年;SARS-CoV-2:2019 年至今),其中四种病毒(SARS-CoV、H1N1 流感、MERS-CoV、SARS-CoV-2)感染会导致呼吸道感染。
病毒感染(尤其是肺部病毒)的一项主要并发症是细菌在病毒感染的器官中定植,这与高发病率和死亡率有关。虽然继发细菌感染或重复感染很大程度上是原发病毒感染引起的免疫易感性的结果,但合并感染是同时发生的多种感染(病毒/细菌/酵母)。病毒大流行期间,合并感染、继发感染或“重复感染”很常见。1918 年西班牙流感大流行导致约 5000 万人死于细菌合并感染,其中许多人是由于细菌重复感染导致继发性肺炎。即使在抗生素时代,在 1957 年 H2N2 和 1968 年 H3N2 大流行中,超过一半的严重感染患者出现细菌并发症。而在 2009 年 H1N1 流感大流行期间,高达 34% 的死亡是细菌合并感染造成的。这些并发症在临床记录中常常被忽视。
因此,我们需要更好地了解病毒感染后继发细菌性肺炎的发病机制,以便在治疗这种破坏性并发症方面取得进展。
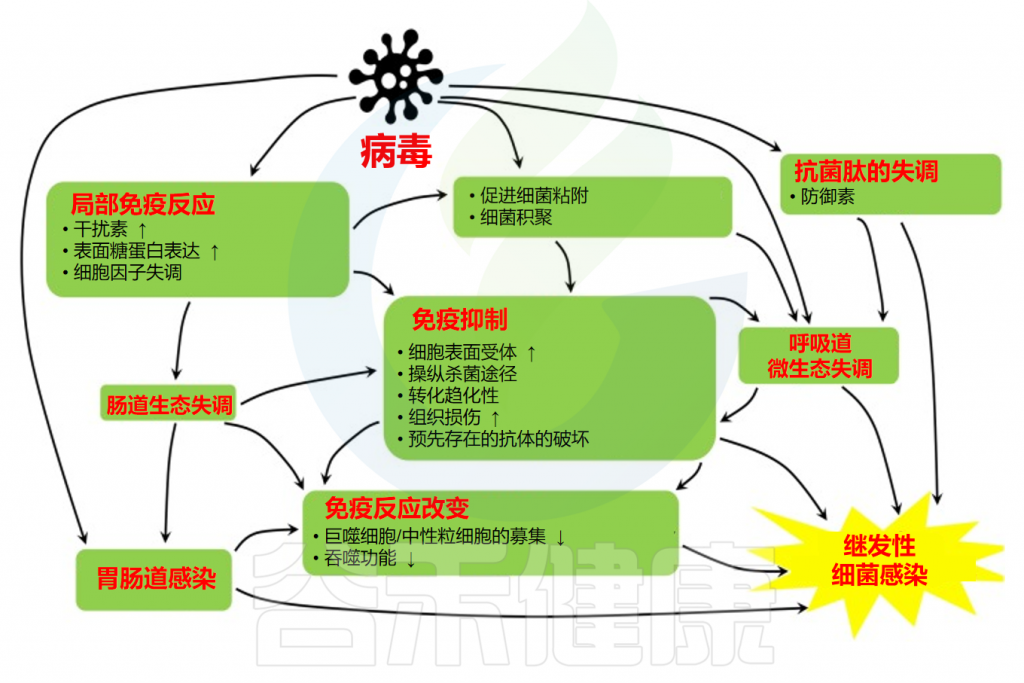
病毒后细菌感染的潜在机制很复杂,包括由病毒、细菌和宿主免疫系统之间的相互作用介导的多因素过程。过去十几年的研究表明,呼吸道病毒常常通过补充机会性细菌病原体的生长而促进继发性细菌感染。病毒感染从组织学和功能上损害呼吸道。
细胞损失、杯状细胞增生、粘液分泌改变、纤毛跳动频率降低、粘液纤毛清除功能不协调以及氧交换减少是病毒感染的特征。这些影响与不同的分子机制有关,通过这些机制,病毒在呼吸道中发生易感性,从而促进细菌感染。
同时病毒感染可能用来驱动急性加重的另一种机制是诱导血管舒张或紧密连接开放因子,这可能会增加浸润率。这也增加了过敏原与气道免疫细胞接触的机会。
呼吸道是一个复杂的器官系统,分为不同的区域;上呼吸道包括鼻腔、咽和喉,而下呼吸道则由传导气道(气管和支气管)、小气道(细支气管)和呼吸区(肺泡)组成。总的来说,这个巨大的表面积蕴藏着一系列不同的微生物群落,其中细菌负荷最高的位于上呼吸道。
呼吸道微生物组的发现始于 2008 年的人类微生物组计划,这要归功于使用 16S rRNA 基因的高通量测序的发展。从那时起,各种研究证明了上呼吸道(URT)和下呼吸道(LRT)中微生物组的存在及其对肺稳态的重要性。自过去十年以来,进行了大量研究来了解病毒感染对微生物组的影响。
各种 NGS 研究表明,呼吸道合胞病毒(Respiratory Syncytial Virus,RSV)和流感病毒通过直接或间接过程改变呼吸道微生物组,导致生态失调。对呼吸道病毒感染的人类样本进行的研究表明,从拟杆菌门到变形菌门的转变,包括许多革兰氏阴性致病菌。
迄今为止,URT的细菌定植被认为是原发性病毒感染后细菌侵入肺部的第一阶段。流感病毒诱导的信号,例如 ATP 的释放,导致生物膜中分散的细菌增加,与致病表型相关。
上呼吸道病毒感染后可出现细菌性肺炎,并使下呼吸道病毒感染复杂化。继发性细菌性肺炎是流感相关死亡的主要原因。
我们评估了以下假设:
i)呼吸道病毒通过改变上呼吸道(URT)的细菌群落结构来影响肺炎的病因;
ii)呼吸道病毒促进或抑制下呼吸道(LRT)的定植)由 URT 中的某些细菌种类引起。
通过对文献的系统回顾,发现病毒既改变了URT中的细菌群落,又促进了LRT中的细菌定植。
流感和细菌性肺炎是全世界传染病发病和死亡的主要原因。多种病毒、细菌和真菌病原体吸入肺部后可引起肺炎。几乎所有这些严重肺炎病例都用抗生素治疗,因为继发细菌感染会使下呼吸道病毒感染复杂化。因此,即使在确定仅由病毒病因引起的肺炎的病例中,病毒和细菌的细菌相互作用也可能发挥一定作用。
确定肺炎病因的挑战
即使在美国等定期进行肺炎监测的国家,大约 65-85% 的住院肺炎病例也没有记录微生物病原学信息。重症患者通常不被纳入监测范围,在患者临床出现时或检测之前,由于怀疑肺炎时需要快速使用抗生素治疗,致病途径上的微生物可能已被清除,并且很少对老年人进行尸检。为了最佳地确定病因,需要通过支气管肺泡灌洗直接取样,但由于易于收集、道德问题和成本,通常对血液、痰液和尿液进行致病因子检测。成人肺炎病例中仅 7%–13% 观察到菌血症,儿童肺炎病例中观察到菌血症为 1–5%,痰液可能被 URT 中的细菌污染,且很难从儿童身上获取,血液和尿液抗原检测需要进一步检测验证或仅限于成人,并且仅针对少数病原体(例如肺炎链球菌和军团菌)。尽管现代分子生物学技术使得对所有存在的细菌、病毒和真菌物种进行非针对性筛查成为可能,但仍然很难区分感染、定植或污染。需要继续努力开发更准确的方法来确定肺炎的病因,从而最大限度地发挥治疗和预防作用。
本文主要探讨了常见的病毒性呼吸道感染以及其可能引起的继发性细菌感染。比较了上呼吸道和下呼吸道微生物组的组成差异,并分析了病毒感染后细菌性肺炎发生的潜在机制。接下来着重讨论了肠道微生物组与呼吸道感染之间的相互作用,以及这种相互作用如何影响宿主的健康和疾病进程。通过这些分析,本文旨在为预防和干预病毒感染后的继发细菌感染提供重要的见解和警示。
病毒感染是一个常见的问题,尤其是在年轻人和老年人中。呼吸道是微生物暴露的主要门户,病毒感染可导致无症状、轻度和自限性或严重且有时致命的感染。尽管已经确定病毒特异性特性(例如寿命和复制动力学)会影响临床表现,但人们不太清楚为什么在感染同一病毒株的人群中可能会出现不同的感染结果。新出现的证据表明肺部和胃肠道微生物组的组成存在人际差异,特别是乳酸菌的成员属,作为定义呼吸道病毒感染结果的关键组成部分。
甲流病毒变异与疫苗挑战
甲型流感病毒 (IAV) 是一种由负义片段化 RNA 病毒引起的人类和动物高度传染性上呼吸道 (URT) 疾病。它被认为是一个主要的公共卫生问题,每年都会造成严重的疾病和经济负担。
注:负义片段化RNA病毒是指RNA病毒的基因组由负义片段组成。在这种病毒中,基因组的信息被存储在负义链上,而不是通常的正义链。负义链的信息需要通过反向转录过程转化为正义链,以便进行蛋白质合成。
频繁的核苷酸替换会导致甲流病毒颗粒表面的血凝素和神经氨酸酶糖蛋白发生变化(也称为抗原漂移),从而需要持续更新疫苗。在疫苗设计和病毒突变之间的这种进化“军备竞赛”下,平均每年导致 3-500 万例重症病例和高达 291,000 ~ 646,000 人死亡。
甲型H1N1流感病毒的传播
分段甲流病毒基因组的模块化结构允许与其他不同的甲流病毒进行遗传重配(抗原转移),从而导致能够引起大规模流行病或大流行的新型病毒的零星出现。过去,新的甲流病毒在未接触过的人群中传播曾引起过大流行,导致严重的发病率和死亡率,其中最引人注目的是 1918 年和 1919 年,当时的西班牙流感导致全球约 20 至 5000 万人死亡。
上呼吸道合并感染
对 1918 年大流行病尸检标本的回顾性分析揭示了由上呼吸道细菌引起的继发性重复感染的流行。然而,细菌合并感染在疾病预后中的作用不仅限于流行病;季节性流感流行期间细菌和病毒的双重感染通常会导致住院人数、重症和死亡人数增加。
微生物组与甲流病毒感染
尽管非患病个体的微生物组相对稳定,但甲流病毒感染已被证明会增加上呼吸道中存在的细菌类群的多样性。具体而言,甲流病毒可导致葡萄球菌属和拟杆菌属以及嗜血杆菌属、梭杆菌属和其他类群的相对丰度发生变化。
在急性或慢性疾病期间,由于局部上皮细胞的变化而导致微生物群的暂时紊乱也是感染的诱发因素。上呼吸道微生物群也可能在调节甲流病毒感染期间诱导的炎症反应中发挥有益作用。
此外,肠道微生物组组成在甲流病毒感染后可正向调节 Toll 样受体 7 信号通路。不过,甲流病毒复制和诱导先天免疫反应对人类或动物上呼吸道微生物组组成的影响,仍有待在社区范围内进行深入阐明和分析。
人类 URT 病毒感染期间假单胞菌大量繁殖
对感染者和健康患者菌群分析结果发现,在感染者队列中最常见的类群是γ变形菌,而在健康患者队列中最常见的类群是放线菌。除此之外,所有其他的分类单元都被归类到一个名为“其他”的附加分类单元中。在流感感染个体的所有样本中,假单胞菌是最丰富的分类群。系统发育推断将该分类单元可靠地定位为假单胞菌属,但无法进一步确定到物种水平。
其他数量较少的门包括拟杆菌门、厚壁菌门、放线菌门和变形菌门的一些其他科,如红杆菌科和巴斯德氏菌科(Gammaproteobacteria)和根瘤菌目的布鲁氏菌科(Alphaproteobacteria)。在对个体进行时间动态分析时,假单胞菌也被确认为主要的分类群。对于未感染的受试者,放线菌是最主要的分类群,而假单胞菌则是存在数量最少的分类群。这些结果在个体受试者的分析中也得到了验证。其他较少丰富的门包括疣微菌门和变形菌门、α变形菌门和ε变形菌门。
未感染 IAV(左)和感染 IAV(右)的人类受试者的综合分类分类
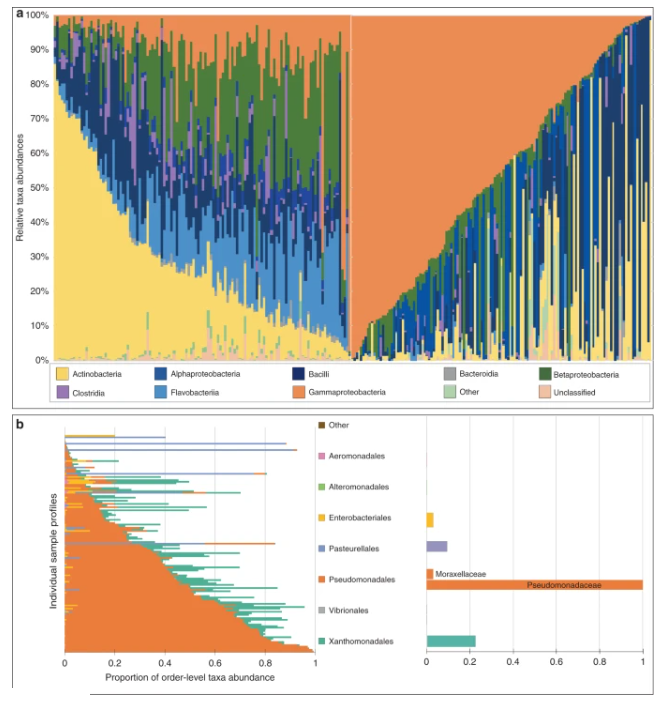
每列都是一个特定样本。该图总结了 >5% 的样本中存在的分类群在类水平上的相对分类丰度(参见下面的图例),按每个队列中最丰富的分类群按降序排序。γ 变形菌(假单胞菌,橙色)在感染患者中普遍存在(右),而放线菌在健康患者中最为丰富。
b在感染患者中观察到的 γ 变形菌目(左)和科(右)水平细分。
流感后细菌性肺炎的严重程度
流感后细菌性肺炎在季节性和大流行性流感病毒疾病相关的发病率和死亡率中发挥着重要作用。大流行预防和管理的一个重要部分是了解流感感染与继发细菌感染之间的关系。
在美国 2003-2004 年流感季节的一项全国调查中,传染病专家观察到成人和儿童患者细菌并发症的发生率分别为 1.6% 和 2%,我国的这个调查数据没有查到,应该也差不多。
——死亡人数
1918-1919 年流感大流行中的大多数死亡可能直接由常见上呼吸道细菌引起的继发性肺炎造成。肺组织样本表明,估计的 20-6000 万人死亡中,大部分是由于细菌重复感染,而不是病毒的直接影响。
——住院情况
在季节性流行期间,流感细菌合并感染与住院人数增加有关。入住 ICU 的四分之一的严重甲型流感感染患者出现细菌或病毒合并感染。系统回顾和荟萃分析显示,细菌合并感染的频率变化很大,范围从 2% 到 65%。
最常见的共感染细菌是:肺炎链球菌和金黄色葡萄球菌
这两种细菌分别占 35%(95% CI,14-56%)和 28%(95% CI,16-40%)。其他多种病原体,例如肺炎嗜血杆菌或肺炎克雷伯菌,引起了其余的感染。
有个研究在2010年至2018年的八个流感季节进行了一项回顾性、观察性研究。在209名流感相关肺炎入院患者中,41名(19.6%)被确定为社区获得性细菌合并感染,主要是沙门氏菌感染和金黄色葡萄球菌。这种现象在流感相关肺炎中经常观察到,但迄今为止尚未发现危险因素。
细菌合并感染可能预测严重程度,并且是院内死亡的独立危险因素。此外,金黄色葡萄球菌和流感的混合感染经常导致致命的协同作用。
呼吸道合胞病毒(Respiratory Syncytial Virus,RSV)是 1 岁以下儿童细支气管炎的最常见原因。RSV 还导致老年人和免疫功能低下的成年人发生急性下呼吸道感染。该疾病通常与同时或继发的细菌感染有关。
RSV 和细菌的共同感染
对医院内 RSV 感染患者进行的多项研究显示,RSV 感染患者与下呼吸道细菌合并感染有关,17.5% 至 44% 的患者 RSV 和细菌合并感染均呈阳性。
最常见的分离细菌是肺炎链球菌和流感嗜血杆菌。然而,最近的研究表明 RSV 和金黄色葡萄球菌之间存在最普遍的关联,尤其是耐甲氧西林金黄色葡萄球菌(MRSA) 。与单纯 RSV 感染相比,细菌合并感染与 RSV 感染通常与易感人群中更严重的疾病相关。
SARS-CoV-2是一种单股正链RNA病毒,属于β冠状病毒属。它通过飞沫传播和接触传播,感染人类的呼吸道,引起了全球范围内的疫情。
截至 发现3年内,SARS-CoV-2 已迅速成为全球主要病原体,COVID-19 大流行影响了全球超过 2 亿人,并导致超过 超过400万人死亡。
因新冠入院的患者细菌合并感染的频率较低
最近一项大型多中心前瞻性队列研究表明,在英国第一波大流行期间,经微生物学证实的细菌感染(主要是继发性感染)在因 COVID-19 入院的患者中很少见(低于 2.3%)。这一结果与之前的几项观察性研究和荟萃分析一致,这些研究和荟萃分析报告了因 COVID-19 入院的患者细菌合并感染的频率较低。
细菌合并感染在重症患者中更为常见
最近,对 3338 名 COVID-19 患者进行了荟萃分析,以评估细菌合并感染。3.5%的患者出现这种现象(95% CI 0.4-6.7%),但14.3%的患者也观察到继发细菌感染(95% CI 9.6-18.9%)。
一般来说,出现细菌感染的 COVID-19 患者比例为 6.9%(95% CI 4.3-9.5%)。然而,细菌合并感染在重症患者中更为常见(8.1%,95% CI 2.3-13.8%)。
在一般住院患者中观察到细菌合并感染的频率较低,可能不需要抗菌治疗。其他荟萃分析研究中也观察到类似的结果。
与单纯感染患者相比,社区获得性混合感染和医院获得性重复感染的 COVID-19 患者的预后最差。
常见的共感染细菌
主要检出肺炎链球菌、肺炎支原体、金黄色葡萄球菌、铜绿假单胞菌、大肠杆菌、流感嗜血杆菌,尤其是危重患者。
相比之下,其他研究声称重症 COVID-19 患者中存在细菌(和真菌)混合感染,包括鲍曼不动杆菌和肺炎克雷伯菌。
所有临床数据都表明,SARS-CoV-2感染者的细菌或真菌合并感染率低于流感病毒感染者。当然这也可能是由于漏报问题、抗生素的广泛使用或限制几种呼吸道病原体传播的控制措施的实施造成的。迄今为止,对 COVID-19 患者合并感染的重要性及其对发病机制的影响仍知之甚少。
继发性细菌感染及其在病毒感染期间的免疫反应列表

编辑

Manna S et al., J Infect Public Health. 2020,1397-1404.
吸道微生物组的多样性与健康关联
人类呼吸道的微生物群是多样且异质的,与多种疾病和表型相关。
上呼吸道栖息着丰富而复杂的微生物组,以健康个体的口腔共生类群为主。
相比之下,健康个体的下呼吸道微生物组(声带下方)的生物量通常相当低,其定义是通过上呼吸道微抽吸输入的竞争动态,以及通过粘液纤毛活动和先天免疫功能清除的竞争动态,可能存在一些有限的局部微生物复制。
因此,上呼吸道微生物组的变化不仅会影响局部微生物组与宿主的相互作用,还会影响下呼吸道的微生物组与宿主的相互作用。
微生物组变化与疾病状态的联系
在各种疾病状态下,通过增加进入下呼吸道、清除缺陷或下呼吸道微生物生长增加,维持低微生物量肺微生物组的平衡可能被破坏。
上呼吸道微生物群在晚期肺病、HIV 感染、血管炎和流感等情况下以及吸烟等暴露条件下会发生改变。
下呼吸道(肺)微生物组不仅在化脓性肺部疾病(如囊性纤维化和肺炎)中发生改变,而且在通常不被认为是微生物引起的肺部疾病,包括哮喘、慢性阻塞性肺病(COPD)和肺纤维化,以及机械暴露等肺部疾病中也发生改变。
微生物组与病毒感染的关系
上呼吸道微生物组的变化与病毒感染的易感性有关,特别是在流感暴露个体中,并且与 RSV 感染的疾病严重程度有关。例如,一项家庭传播研究发现链球菌属的丰度增加。病毒感染也可以改变细菌微生物组,从而导致潜在的双向相互作用。因此,人们对气道微生物组在 COVID-19 中的潜在作用的研究产生了浓厚的兴趣。
呼吸道微生物组对免疫系统的影响
呼吸道微生物群影响宿主健康的方式之一是通过局部和全身免疫系统的调节。如在 COVID-19 中的免疫反应与疾病进展和结果密切相关。
在健康方面,肺部免疫张力受到通过上呼吸道微抽吸获得的生理微生物群的调节,气道生态失调与炎症增加有关,例如哮喘或接触香烟烟雾。
重要的是,过去五年的研究表明,呼吸道微生物组在调节急性肺损伤患者或有急性肺损伤风险的患者的严重程度方面发挥着作用。肺微生物组负荷和组成与急性肺损伤患者的局部和全身炎症相关,并预测患有急性呼吸窘迫综合征(ARDS)的机械通气危重患者的临床结果。
创伤后,肺微生物组的组成与 ARDS 的发生有关,并且被认为可以介导吸烟对 ARDS 发生风险的影响 。因此,呼吸道微生物组与多种可导致严重肺损伤的情况下的局部和全身机制相关,并可能通过局部和全身机制影响结果。
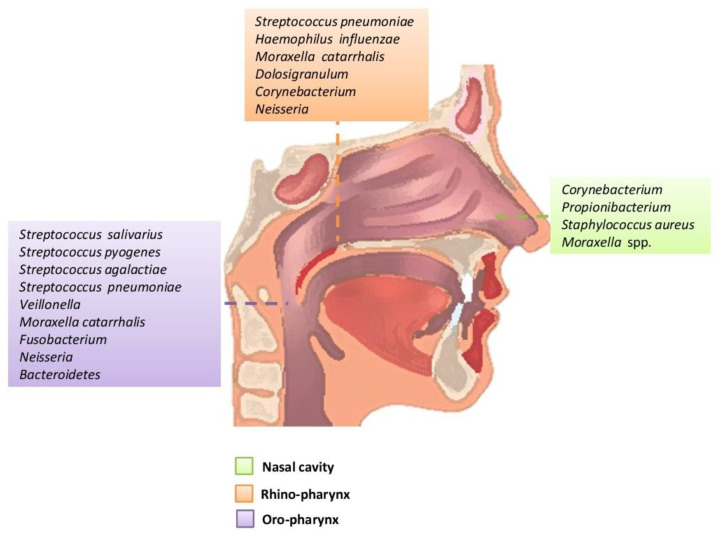
成人上呼吸道的主要细菌属和微生物组种类
doi: 10.3390/biology9100318
在 COVID-19 中,已观察到免疫抑制和失控的炎症,并可能导致更严重的疾病,而保护性免疫涉及诱导体液和细胞介导的反应。感染可诱导多种促炎细胞因子,如 IL-1B、IL-6、TNF、IL1RA、CXCL10/IP10、MIP-1α 和 CCL2。几种 SARS CoV-2 蛋白能够通过延迟 I 型干扰素反应来抑制抗病毒免疫。
上面讲过上呼吸道和下呼吸道具有相似的微生物群落组成,尽管前者在健康宿主中的微生物密度要高得多。已知有几个因素会影响气道微生物组的组成,包括感染史、年龄、遗传和结构性肺疾病。
上呼吸道是一个相互连接的系统,由前鼻孔、鼻腔、鼻咽、鼻窦、咽鼓管、中耳腔、口腔、口咽和喉组成,每个系统都作为独特的生态位,拥有自己的微生物群落。
在健康成年人中,鼻腔中存在的细菌通常是与皮肤相关的细菌,主要是放线菌门(例如棒状杆菌属、丙酸杆菌属),其次是厚壁菌门(例如葡萄球菌属)和变形菌门。
口咽部含有厚壁菌门、变形菌门和拟杆菌门的成员,包括链球菌、奈瑟菌、嗜血杆菌和毛螺旋菌属。皮肤和口腔谱系以鼻咽为代表,例如链球菌、葡萄球菌、棒状杆菌和普雷沃菌。
包括肺炎链球菌、脑膜炎奈瑟菌和流感嗜血杆菌在内的有限数量的病原体是 URT 的共生细菌。
呼吸道微生物组的分布与动态
在健康个体中,LRT 中的微生物群落丰富度(即细菌类群总数)低于 URT 中的微生物群落丰富度(即细菌类群总数)。
与正常健康的肺部是无菌环境相反,可以使用测序技术来识别独特且有些动态的肺部微生物组,微抽吸是微生物从 URT 迁移到 LRT 的主要途径。
健康/慢性气道疾病中肺部微生物组的特征
健康肺部的主要门是拟杆菌门和厚壁菌门,其中主要包括普雷沃氏菌、韦荣球菌和链球菌。患有慢性气道疾病(例如囊性纤维化、慢性阻塞性肺病)的个体肺部细菌数量增加,并且某些物种的相对丰度存在差异。
微生物群落与呼吸道感染
由于内在或外在因素导致的气道清除受损导致可以利用这种生长机会的细菌种类的增殖。URT 的细菌定植通常被认为是侵袭性细菌感染发展的第一步,包括呼吸道病毒感染后的继发性细菌感染。下面将更详细地讨论细菌丰度、物种多样性以及影响后续感染免疫反应的因素。
呼吸道合并感染和重复感染涉及多种机制。长期以来,病毒感染对上皮屏障的影响被认为是细菌二重感染的主要原因。最近,多项研究表明,抗病毒免疫反应也在混合感染中发挥作用。
病毒对继发细菌感染的易感性机制
doi: 10.1016/j.jiph.2020.07.003
尽管在下文单独整理讲述,但是生理学和免疫学机制是同时存在且密切相关的。
病毒感染可破坏上皮完整性,导致紧密连接和细胞骨架功能障碍
上皮细胞在防止吸入病原体和颗粒物的入侵方面具有至关重要的作用。上皮细胞通过紧密连接组装成假复层结构,为病原体创造了不可渗透的屏障。SARS-CoV-2感染后,在气液界面培养的人原代上皮细胞(HAE)中观察到跨上皮阻力急剧下降和细胞形态改变,表明上皮完整性被破坏。
使用类似的体外模型对流感和 RSV 进行了类似的观察。在动物模型中还发现病毒感染后观察到的脱屑、纤毛缺失、免疫细胞浸润和坏死之间存在相关性。
单次病毒感染后经常观察到紧密连接和细胞骨架功能障碍。RSV 和流感病毒可以通过直接或间接靶向相关蛋白质(例如claudin、occludin 或ZO-1)来减少紧密连接,并诱导F-肌动蛋白细胞骨架重排,从而导致细胞形态改变。
呼吸道病毒引起的上皮损伤是导致继发感染的原因之一
原发性流感或RSV感染会引起上皮损伤,导致动物模型对金黄色葡萄球菌或肺炎链球菌的易感性更高。
病毒诱导的细胞凋亡也可能通过多种机制(例如 FasL/TRAIL 途径)导致上皮屏障的丧失。尽管没有经过专门研究,但一些研究表明病毒诱导的细胞凋亡/坏死与对细菌重复感染的较高易感性之间存在相关性,细胞碎片可改善细菌粘附和侵袭。
病毒感染→粘液分泌增加和纤毛损伤→破坏粘液纤毛清除机制
病毒引起的气道功能改变也是呼吸道合并感染的原因。上皮可以通过粘液纤毛清除来阻止感染,粘液纤毛清除重新组合了两种基本机制:
粘液纤毛清除机制受损→气道阻塞→为继发性感染创造条件
众所周知,上皮细胞在感染后会产生粘液,以减少流感、RSV 或 SARS-CoV-2 的感染。然而,这会导致气道阻塞,反映出在患者中观察到的肺活量下降。RSV 和流感病毒在感染后也会增加粘液相关蛋白,例如粘蛋白。
呼吸道病毒主要针对纤毛上皮细胞,诱导细胞死亡,从而导致纤毛损失。到目前为止,还没有研究明确关注合并感染期间粘液纤毛清除的分子机制。
RSV 和流感病毒增强病原体粘附
原发性病毒感染也会增加呼吸道中的细菌粘附。RSV 感染通过 G 糖蛋白与细菌成分的直接结合,增加肺炎链球菌对上皮细胞的粘附和毒力。
G 糖蛋白在感染后锚定在细胞膜上并充当细菌受体。转录组学研究表明,RSV 增加了 RSV 感染细胞表面的粘附分子,例如 CD47,导致肺炎链球菌粘附增加。
病毒蛋白质作为细菌受体
流感病毒还通过增加受感染细胞膜上的纤维蛋白原、糖蛋白修饰和唾液酸,来增强各种模型中金黄色葡萄球菌或肺炎链球菌的粘附。血小板激活因子受体 (PAF-R)也与细菌相互作用,促进重复感染。流感糖蛋白血凝素和神经氨酸酶也促进细菌粘附,充当或暴露细菌受体。
冠状病毒与特定细菌的重复感染
另一种人类冠状病毒 (HCoV-NL63) 增强了LLC-MK2 和 HAE 细胞中肺炎链球菌的重复感染,但对金黄色葡萄球菌、流感嗜血杆菌或铜绿假单胞菌等其他细菌没有增强作用。
重复感染加剧病理严重程度
流感和肺炎链球菌或金黄色葡萄球菌重复感染的各种小鼠模型在流感感染后 7 天接种细菌时表现出致命的协同作用,这表明重复感染在病毒感染后期的修复过程中增强了病理严重程度。
病毒感染干扰细胞修复机制
观察到修复细胞反应受损,尤其是巨噬细胞和上皮细胞的修复细胞反应受损,细胞再生减少和稳态信号通路改变。RSV 可以通过增加 MMP 或生长因子的产生来干扰修复机制,从而导致纤维化加剧。
细胞修复延迟与重复感染相关
流感病毒和肺炎链球菌重复感染小鼠模型的转录谱显示,48hpi 上皮细胞增殖和上皮修复增加。此外,观察到基因上调与疾病严重程度之间存在相关性,表明修复机制的改变与重复感染有关。最近证明,流感病毒诱导的 I 型和 III 型干扰素可延迟修复阶段的上皮细胞增殖。注意到 p53 激活和细胞周期改变导致细胞凋亡增加,导致细胞分化和生长抑制以及上皮修复延迟。
感染流感的Ifnlr1−/−小鼠在重复感染肺炎链球菌后具有更好的存活率,这表明病毒感染后诱导的IFN-λ延迟了上皮修复并导致继发性细菌感染。
共同感染的生理机制总结如下:
与细菌重复感染相关的生理机制的示意图
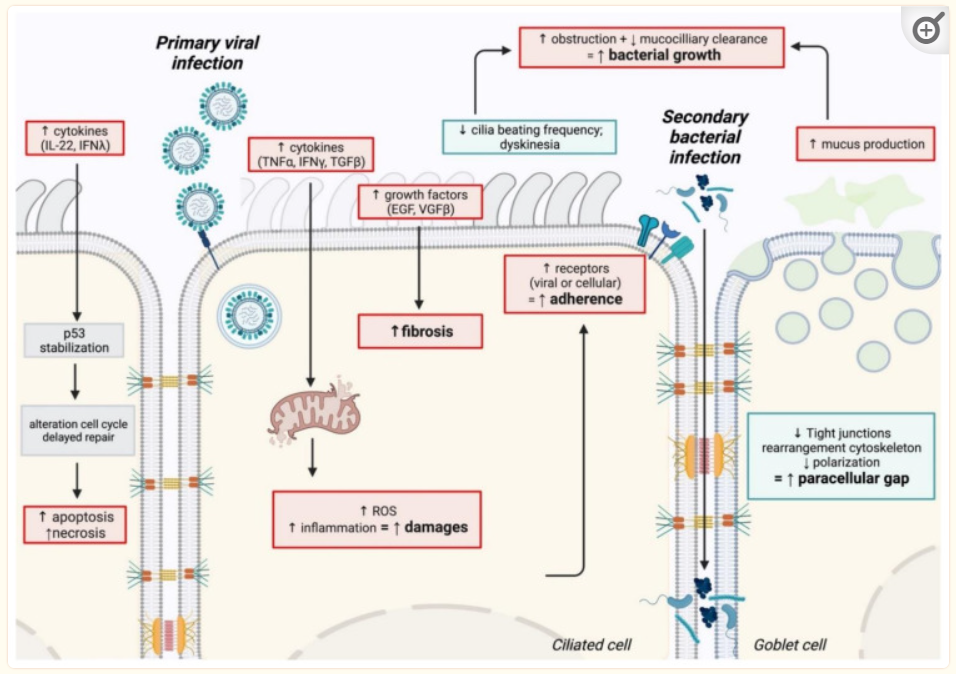
原发性病毒感染对上皮完整性和功能的影响(上皮损伤、修复延迟)有助于为继发性细菌感染的建立创造有利的环境。
呼吸道病毒通过URT进入人体,其一种可能的机制是改变上呼吸道的微生物组成,促进病原体的生长,并促进大量细菌随后进入。
横断面研究
病毒感染与细菌定植的互作
考虑到病毒对增强细菌对上皮细胞粘附的影响,对人类受试者以及动物模型的多项研究表明,病毒感染与潜在致病菌的定植增加有关,这也许并不奇怪。
使用 qPCR 检测感染或未感染甲型流感的成年患者中的特定细菌进行的比较分析显示,感染患者中分别有 12%、24% 和 32% 存在金黄色葡萄球菌、肺炎链球菌和流感嗜血杆菌,而 5% 的感染患者中共同存在金黄色葡萄球菌、肺炎链球菌和流感嗜血杆菌。在体外实验模型中,病毒感染会增加 URT 中各种细菌的定植率 ,包括肺炎链球菌和流感嗜血杆菌。
较高的致病菌负荷与呼吸道病毒合并感染,和继发性细菌感染风险增加相关
在儿童中,流感与鼻咽部肺炎链球菌滴度增加 15 倍相关。动物模型也同样证实,病毒感染,尤其是流感,会增加 URT 中的细菌定植率,从而增加继发细菌感染的风险。
在调整年龄和性别后,较高的肺炎球菌定植密度与呼吸道病毒合并感染和侵袭性肺炎球菌肺炎有关。
另一项比较患有和不患有肺炎的鼻咽细菌的病例对照研究也发现,鼻咽部肺炎链球菌(而非流感嗜血杆菌和卡他莫拉菌)的负荷与病毒合并感染和肺炎之间存在关联。
病毒感染可能会增强细菌的传播
在一项对小鼠定植肺炎链球菌并在 3 天后感染甲型流感病毒的研究中,只有当所有小鼠都感染流感并被流感中和抗体阻断时,才会发生肺炎链球菌传播。然而,虽然特定细菌可能在病毒感染期间获得竞争优势,但这并不普遍适用于所有细菌类群。最近一项针对患有和未患有呼吸道病毒感染的受试者的研究表明,与未感染的对照组相比,病毒感染受试者鼻咽样本中的总体细菌丰度较低。
急性病毒感染和细菌定植之间的关系是双向的
细菌携带或其配体可以增加或降低病毒感染率,从而对随后的宿主对病毒感染的免疫反应产生积极或消极的影响。接触肺炎链球菌可以增强呼吸道中的病毒复制。
与没有定植的患者相比,携带肺炎链球菌的患者更有可能经历随后的急性呼吸道疾病发作。此外,气道中存在的细菌可以调节宿主针对病毒感染的反应。
鼻咽共生体的存在,可以保护小鼠免受 RSV 诱导的气道高反应性
感染 RSV 的小鼠在接受抗生素介导的鼻咽部草绿色链球菌清除后,表现出炎症淋巴细胞数量和气道高反应性增加,以及调节性 T 细胞数量和转化生长因子-β 产生的减少。
特定细菌定植对免疫应答的影响
其他研究表明,金黄色葡萄球菌在上呼吸道的定植可通过将 2 型 CC 趋化因子受体+分化簇 (CD)11b +单核细胞子集募集到肺部并诱导 M2,从而显著降低小鼠中流感引起的急性肺损伤和死亡率。
病毒感染对微生物组多样性的影响不一致,受多因素影响
随着新一代 16S rRNA 测序的出现,基于微生物组的研究试图辨别病毒感染期间每个解剖生态位的细菌群落的整体变化模式,例如多样性的变化。多样性可以使用各种指数来评估,例如微生物组的独特物种总数(即丰富度)或考虑群落成员的丰富度和相对丰度均匀度的其他衡量标准(例如香农指数)。
当将病毒感染受试者与健康对照进行比较时,微生物组分析的结果并未显示出一致的多样性变化。考虑到采样对象的可变性、病毒感染的类型和严重程度、样本收集的类型和时间以及分析方法的差异。
多样性与严重程度有关
法国对因流感入院的儿童进行的一项研究表明,随着流感严重程度的增加,鼻咽微生物群的多样性也随之增加。
多样性与病毒感染的类型有关
我国最近一项针对 H7N9 禽流感患者的研究表明,与健康对照相比,H7N9 感染患者的口咽微生物组多样性显着增加,特别是继发细菌性肺炎的 H7N9 患者。
相反,法国的一项鼻咽样本研究和韩国的一项急性呼吸道病毒感染患者口咽样本研究均显示,与健康对照相比,病毒感染期间多样性指数有所下降 。
这两项研究的受试者范围从婴儿到 80 岁以上的成人,限制了有关年龄相关影响的结论。在接受鼻病毒实验性自我接种的健康志愿者中进行的纵向研究也未能证明 URT 微生物组多样性的显着变化,而对健康成年人注射 LAIV 疫苗导致病毒攻击后多样性指标的增加。
因此,与多样性降低被认为对宿主有害的其他疾病不同,病毒感染对多样性本身的影响是可变的,目前不被认为是并发症风险的良好指标,包括继发性细菌性肺炎。
微生物组测序研究还使研究人员能够同时识别多个细菌类群之间的丰度变化,而不仅仅是可以单独培养的细菌类群。这使得研究人员能够确定哪些细菌群在病毒感染期间一致变化,哪些细菌群相互竞争。
各种病毒感染中,微生物群的变化
法国最近一项基于宏基因组学的研究报告称,与健康对照相比,确诊呼吸道病毒感染受试者的鼻咽样本中金黄色葡萄球菌、肺炎链球菌、流感嗜血杆菌、卡他莫拉菌和肺炎克雷伯菌富集。
对感染和未感染 2009 年甲型 H1N1 流感大流行病毒感染的肺炎患者的口咽微生物组进行的检查显示,厚壁菌门(包括葡萄球菌和链球菌)和变形菌(主要是杏仁假单胞菌、荧光假单胞菌、假单胞菌UK4、鲍曼不动杆菌)——在流感患者中显着富集。
另一项针对 2009 年大流行 H1N1 流感感染患者的研究表明,尽管未包括正常对照,但携带大流行 H1N1 流感的患者上呼吸道(鼻和鼻咽样本)的主要门是放线菌门、厚壁菌门和变形菌门;然而,作者认为流感与变形菌的扩张有关,而变形菌在健康宿主中的数量通常较少。这些发现得到了另一组的支持,他们发现莫拉氏菌和肠杆菌属(被归类为变形菌门)是从大流行性 H1N1 流感患者的鼻咽样本中获得的最具代表性的细菌。然而,这些研究表明,受试者之间存在相当大的变异性,强调需要进行纵向研究来破译病毒感染后的变化。
特定病毒是否始终与某些细菌类群的富集相关
在澳大利亚原住民和非原住民儿童的鼻咽腔中,检测到 HRV 与肺炎链球菌、流感嗜血杆菌和卡他莫拉氏菌携带之间以及腺病毒与卡他莫拉氏菌之间存在正相关性。另一项研究通过 PCR 检测 20 种呼吸道病毒的存在以及儿童鼻咽部细菌携带的流行率,发现金黄色葡萄球菌定植与流感病毒之间存在很强的正相关性。
此外,肺炎链球菌定植与 HRV 和肠道病毒的存在呈正相关。流感嗜血杆菌与 HRV 和 RSV 呈正相关;卡他莫拉氏菌定植与冠状病毒和腺病毒呈正相关。
一项针对患有急性 RSV 或 HRV 呼吸道感染的婴儿进行的基于 16s rRNA 测序的研究报告称,患有 RSV 的婴儿的葡萄球菌丰度显著更高。
上呼吸道细菌群特征与宿主年龄相关
通过不依赖于培养物的焦磷酸测序对 57 名健康无症状个体和 59 名流感病毒、副流感、hRV、RSV、冠状病毒、腺病毒或偏肺病毒患者的 URT 细菌含量进行分析,揭示了六种不同的细菌谱,即链球菌+普雷沃氏菌+韦荣氏菌、链球菌+嗜血杆菌+奈瑟菌、链球菌、莫拉氏菌、嗜血杆菌和克雷伯氏菌。然而,这些特征与病毒类型无关,但与受试者的年龄有关。
病毒后细菌性肺炎成因复杂,需深入研究
鉴于许多人类研究本质上是横断面的,目前尚不清楚病毒后细菌性肺炎是否可能是病毒感染增强细菌定植或获得、定植细菌影响宿主对呼吸道病毒感染的易感性或两者结合的结果。另一个复杂的因素,特别是在病毒感染期间检查微生物组的横断面研究中,是组群控制不佳,而且考虑到可能影响呼吸道微生物组的变量数量(例如年龄、性别、口腔卫生和挖鼻孔习惯、基于医疗保健的就业状况、吸烟状况、药物使用、接触幼儿等。
病毒感染的潜在类型、感染发生后的采样时间点、感染的严重程度以及伴随的抗菌药物使用情况其他混杂因素。这可能是病毒感染患者微生物组研究中高度可变且有时不一致的观察结果的基础。
纵向研究
由于感染前采样困难,很少有临床研究比较患有急性病毒感染的其他健康个体感染前和感染后微生物组的基线。然而,现有的研究相对较少,可以深入了解细菌定植模式随时间的动态和稳定性,以及急性病毒感染引起的扰动是否以及如何改变这些模式。
在健康儿童中,鼻咽部微生物群的主要门是变形菌门、厚壁菌门、拟杆菌门、放线菌门和梭杆菌门,其中莫拉菌、嗜血杆菌、链球菌、黄杆菌、多洛西颗粒菌、棒状杆菌和奈瑟菌为主要属。
不同季节鼻咽微生物组变化的研究不一致
不同季节观察到鼻咽微生物组多样性的变化,秋季冬季以变形菌门和梭杆菌门为主,春季以拟杆菌门和厚壁菌门为主;这些差异与最近的抗生素和病毒合并感染无关。
然而,对 40 名哮喘儿童和青少年相隔 5.5-6.5 个月收集的两次鼻咽冲洗液进行的另一项分析表明,不同季节鼻咽微生物组多样性没有显着差异,尽管嗜血杆菌、莫拉氏菌、葡萄球菌和棒状杆菌的平均相对丰度在夏季和秋季样本之间以及不同年龄组之间存在显著差异。
此外,在 87.5% 的患者中,患者微生物丰度在不同时间点之间存在显着差异。对冬季和夏季无症状医疗人员的细菌和病毒载量的频率和季节变化进行的一项调查显示,在冬季接受测试的 100 名受试者中,34 人被至少一种细菌定植,11 人至少检测出一种病毒的阳性。
最常检测到的病原体是耐甲氧西林金黄色葡萄球菌(MRSA)、卡他莫拉菌和冠状病毒。相比之下,在夏季测试的 100 名受试者中,37 名受试者至少携带一种细菌(主要是 MRSA 和肺炎克雷伯菌),四名受试者检测出一种病毒呈阳性。
另一项对 31 个有小孩的家庭进行纵向鼻拭子采样的研究表明,鼻病毒感染与社区中肺炎链球菌感染的增加以及肺炎链球菌在家庭内传播的增加有关。
其他小组研究了将 hRV 实验接种到 URT(鼻孔)中的效果。这些研究报告称,随着时间的推移,鼻咽样本或咽拭子的总读数或主要门(例如放线菌门、厚壁菌门和变形菌门)没有显著变化。在口咽部,鼻病毒感染与副流感嗜血杆菌、微黄奈瑟菌(Neisseria subflava)相对丰度短暂增加的强烈趋势以及金黄色葡萄球菌相对丰度增加的微弱趋势相关。到 60 天时,这些细菌的丰度已恢复到基线。
鼻咽取样显示完全相反的结果,在 hRV 感染后的受试者中,嗜血杆菌和奈瑟菌属的相对丰度降低,但正常鼻共生丙酸杆菌增加。没有观察到葡萄球菌的差异。然而,这两项研究的受试者数量都很少,限制了检测随时间变化的能力。
与 URT 微生物组的研究相比,由于难以从肺部获取未污染的样本,因此对病毒感染后的 LRT 微生物组的研究相对较少。方便的样本(例如痰液)会受到口腔污染,但支气管镜样本是侵入性的,并且定期获取的成本很高。
此外,目前还不清楚,除了慢性肺病(例如慢性阻塞性肺病)患者之外,肺部微生物负荷是否足以对免疫反应和病毒感染期间继发细菌感染的风险产生强大影响。
来自流感感染小鼠模型的数据似乎表明,流感感染对肺部微生物组的细菌计数、多样性和组成仅产生适度的影响。
在 hRV 感染后患有慢性阻塞性肺疾病(COPD) 的受试者中,但在健康个体中则不然,基线时细菌负荷和细菌生长有所增加,特别是流感嗜血杆菌。
研究人员观察到,细菌的生长似乎源于现有的群落。将肺炎链球菌鼻内接种到预先感染流感病毒的小鼠体内,首先在鼻子中定植,几天后气管和肺部出现化脓性炎症。然而,在未感染的动物中没有观察到这种效应。这表明,在流感病毒感染者中,肺炎球菌感染可能从URT顺序发展到LRT。因此,一些流感感染者的肺部微生物组可能会因 URT 微生物群落的变化而发生变化。
微生物间相互作用
细菌中已经进化出竞争优势的机制,使它们能够在原本恶劣的宿主环境中生存。然而,种间竞争也可以通过捕获稀缺资源(例如铁)的能力或有针对性地杀死其他细菌(例如细菌素)来维持微生物群落的稳态,从而防止一种微生物主宰群落。
因此,急性病毒感染引起的免疫反应、病毒引起的宿主上皮表面的变化或病毒本身可能导致消除负责控制病原体的宿主共生体。例如,鼻孔中表皮葡萄球菌和痤疮丙酸杆菌的丰度与金黄色葡萄球菌携带呈负相关。
了解这些相互作用可能会为旨在减少流感流行或大流行期间致病菌定植的治疗干预措施创造新途径。
一组已被检查其在抑制金黄色葡萄球菌和肺炎链球菌鼻腔携带中的作用的共生菌是棒状杆菌属。日本的一项早期研究报告了将棒状杆菌菌株引入金黄色葡萄球菌持续携带者的健康成年医院工作人员的鼻孔中的效果,并成功根除 71% 的受试者,该机制似乎不依赖于细菌素。
相比之下,表皮葡萄球菌植入没有效果。所使用的表皮葡萄球菌菌株是否表达丝氨酸蛋白酶Esp(其抑制金黄色葡萄球菌生物膜形成和鼻定植)尚不清楚。
另一个小组的后续研究报告称,假白喉念珠菌抑制金黄色葡萄球菌生长,而Corynebacterium accolens和金黄色葡萄球菌似乎相互支持彼此的生长。相反,其他研究人员观察到棒状杆菌属。
在未鼻腔定植肺炎球菌的儿童中富集,并证明C. accolens通过表达脂肪酶来抑制体外肺炎链球菌生长,该脂肪酶从皮肤表面三酰甘油释放游离脂肪酸,从而抑制肺炎球菌生长。
因此,对共生体之间种间竞争的鉴定和机制了解,可能会带来新的见解,即病毒感染如何为致病生物赋予竞争优势,以及如何利用共生体采用的自然策略来恢复宿主微生物生态位的稳态。
有趣的是,最近的一项临床前研究使用RSV和肺炎链球菌双重感染的鼠模型,通过假白喉梭菌菌株进行鼻引发,以增强宿主对病毒感染的防御,从而增强对继发细菌攻击的清除并减少肺损伤。
感染病毒后的继发感染
感染病毒对细菌的直接影响可能促进病原体转变。宏基因组分析表明,与未感染患者中的相同细菌相比,pH1N1 相关的气道微生物群富含与细胞运动、转录调节、代谢和趋化反应相关的基因。这些数据表明,流感感染会扰乱呼吸道微生物组,导致产生包括免疫调节分子在内的次级代谢产物。病毒还会损害细菌生物膜的形成并破坏现有的生物膜。流感已被证明会通过下调与定植状态相关的基因表达和上调细菌素来影响肺炎链球菌转录组。因此,病毒对细菌转录模式的直接影响可能是定植细菌获得侵入潜力的一种机制,从而导致细菌重复感染。
在人体的生态位中,肠道微生物群落是研究最深入的,迄今为止已发表超过 20,000 篇论文。虽然病毒组和真菌组也在分析中,但大部分文献都集中在微生物组的细菌成分上,因此我们对肠道微生物组与宿主免疫和慢性疾病发病机制之间关系的大部分理解来自主要来自细菌群落的研究。
这里主要介绍肠道微生物组如何维持健康的宿主免疫环境的几个重要方面。
肠道微生物代谢产物与免疫
首先,肠道共生体产生的细菌代谢产物有助于维持完整的上皮完整性、调节性 T 细胞发育和相对抗炎的免疫状态。
特别是,短链脂肪酸(SCFA),例如乙酸盐、丙酸盐和丁酸盐,是大肠细菌发酵膳食纤维和碳水化合物的产物。短链脂肪酸可以:
同时,微生物代谢物对于促进肠道针对病原体的免疫反应至关重要,包括诱导 IL-18和防御素的分泌。因此,微生物组代谢产物对于粘膜屏障完整性和免疫稳态的适当调节至关重要。
此外,细菌群落的特定成员可以促进免疫系统的正常成熟和发育。虽然这仍然是一个正在深入研究的领域,但一个值得注意的例子是发现分段丝状细菌是肠粘膜 IgA 产生 和 Th17 细胞诱导的关键促进剂。
肠道微生物失衡与慢性疾病
菌群失调或微生物群组成的不平衡与多种慢病有关,其中许多疾病以慢性炎症或代谢异常为特征,包括炎症性肠病、心血管疾病、糖尿病等。因此,培养平衡的肠道微生物群多样性和组成对于促进健康和免疫稳态至关重要。
肠道微环境影响微生物动态变化
在健康期间,微生物群的组成受到每个解剖生态位特有的许多选择压力的控制,包括温度、营养可用性、pH、氧张力和局部免疫环境。由疾病、抗生素使用或饮食变化(例如饥饿)引起的肠道微环境的短期扰动可以改变肠道微生物群,并随后导致免疫反应的短暂改变。
因此,研究流感和其他呼吸道病毒是否改变胃肠道微生物组可能对病毒介导的抗菌免疫反应抑制具有机制意义。
肠道微生物群在免疫方面的这种有益作用不仅限于肠道区室,还延伸到全身区室和远处器官,例如肺部。
缺乏微生物群(即无菌)或口服抗生素(广谱或靶向抗生素,如新霉素、甲硝唑或万古霉素)治疗的小鼠对全身和呼吸道感染的反应受损。
在由例如甲型流感病毒(IAV)和呼吸道合胞病毒(RSV)引起的病毒性呼吸道感染的情况下,抗生素治疗对肠道微生物群生态的严重扰乱削弱了宿主的先天防御和适应性防御。
肠道微生物群对肺部产生I型干扰素的影响
众所周知,这些干扰素可控制病毒感染,包括SARS-CoV-2,COVID-19 的病原体。在这种情况下,脱氨基酪氨酸(源自类黄酮和氨基酸代谢)和短链脂肪酸等微生物代谢产物至关重要。
例如,研究表明,由专性梭状芽孢杆菌(Clostridium orbiscindens、对甲硝唑和万古霉素敏感)能够消化植物中黄酮类化合物,产生的脱氨基酪氨酸可以扩散到血液中,到达肺部并启动(先天)免疫系统,帮助身体抵抗流感病毒的感染。
脱氨基酪氨酸促进肺部 IFN 刺激基因的合成,而肺吞噬细胞在此机制中至关重要。在这种情况下,脱氨基酪氨酸通过 IFN-α/β 受体和信号转导器和转录激活剂增强 I 型 IFN 信号传导。例如,研究表明乙酸盐可以扩散到血液中,并激活基质细胞表达的GPR43。这种激活途径改善了 I 型干扰素反应并增加了干扰素刺激的基因表达。GPR43 促进 I 型 IFN 介导的流感感染保护作用的机制需要阐明。
除了微生物代谢物外,源自完整肠道微生物群的微生物膜成分(包括TLR配体)也在宿主防御病毒性呼吸道感染中发挥着关键作用。
事实上,局部和远端接种 Poly (I:C)(TLR3 激动剂)或肽聚糖(TLR2 激动剂)可以挽救抗生素治疗小鼠的免疫损伤。
值得注意的是,除了肠道微生物群发出的这些远端信号外,来自上呼吸道的局部微生物也调节下呼吸道免疫力,并在肺部防御病毒感染方面发挥作用。这些机制在很多研究中得到证实。
↑抗生素改变肠道菌群可能增加严重程度
肠道微生物群不仅影响先天免疫反应,还可以增强 CD8 + T 细胞效应器功能,这一过程也参与病毒(流感)清除。如上所述,用抗生素改变肠道微生物群会增加病毒性呼吸道感染(例如流感)的严重程度。
↓高纤维饮食刺激肠道菌群可能减轻严重程度
用高纤维饮食刺激微生物组会产生相反的效果。就流感而言,高纤维(可发酵菊粉)饮食通过两种不同的途径提供保护。膳食纤维的发酵导致骨髓造血功能改变,导致 IAV 感染小鼠肺中替代激活的巨噬细胞积聚。这些巨噬细胞产生较少的趋化因子(CXC 基序)配体 1,从而减少早期中性粒细胞浸润到气道中。
该途径极大地减少了过度的肺部炎症和损伤。该饮食还促进CD8+T细胞代谢,增强CD8+T细胞的效应功能,有效增强病毒清除。
保护作用需要 GPR41(也称为游离脂肪酸受体 3),口服 SCFA(丁酸盐)足以提供保护。在 RSV 感染期间也观察到了类似的保护作用。高纤维(可发酵果胶)饮食通过调节肺上皮细胞中的 I 型干扰素反应和增加肺部干扰素刺激基因的表达来防止 RSV 感染。保护作用需要 GPR43,口服乙酸盐足以提供保护。
肠道微生物群在病毒性呼吸道感染中的作用
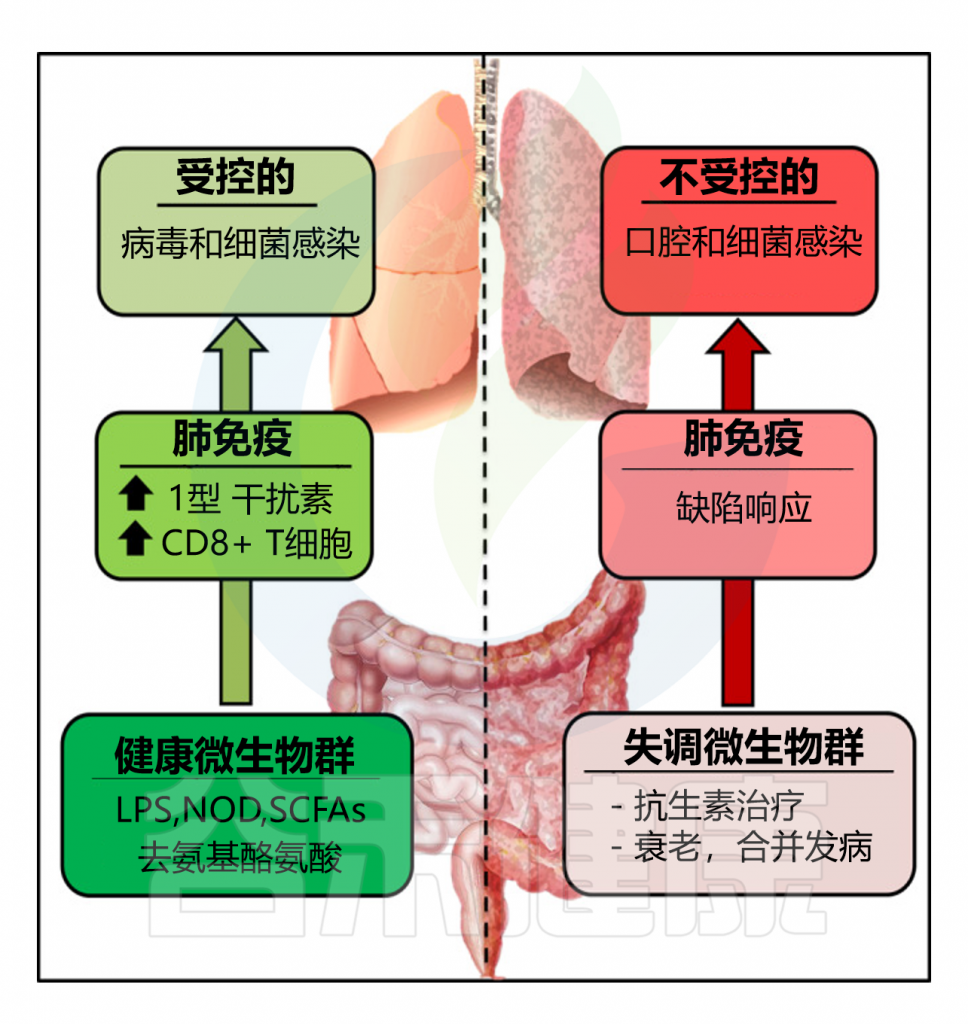
Sencio V, et al. 2021,14:296-304.
健康肠道微生物群释放的因子可以帮助肺部抵御病毒性呼吸道感染。相反,抗生素治疗会扰乱肠道内的生态平衡并改变肺部的防御能力。衰老和合并症也与肠道微生物群功能的改变和呼吸道感染的易感性增加有关。
在肺部发挥抗病毒作用的肠道共生细菌的性质仍然难以捉摸。如上所述,厌氧梭菌成员(脱氨基酪氨酸)和产短链脂肪酸菌对于肺部先天免疫系统的启动非常重要。最近,一项基于易感动物和耐药动物比较的研究表明,双歧杆菌属(假长双歧杆菌和动物双歧杆菌)的定植与流感感染小鼠的生存密切相关。很可能在不久的将来,其他共生成员将被识别出来,并将作为新的生物标志物来预测严重病毒性呼吸道感染患者的严重程度和死亡率。
因此,在稳定状态下,肠道微生物群中的常驻细菌可以自然地远程支持肺部对抗病毒性呼吸道感染。
研究表明,除了饮食模式,呼吸道病毒感染及其他压力因素也会影响肠道微生物群的组成。流感和RSV肺部感染的动物模型显示,即使肠道中没有检测到呼吸道病毒,肠道微生物组也会受到感染的显著影响。
流感病毒和肠道菌群
流感感染可导致轻度至重度肺炎、急性呼吸窘迫综合征 (ARDS)(临床上定义为急性呼吸衰竭)和多器官功能障碍。尽管肠道内明显没有病毒,但一些患者仍然出现类似胃肠炎的症状,如腹痛、恶心、呕吐、腹泻。这表明肺-肠轴在流感感染期间可能很重要。
根据可操作分类单位数量评估,细菌负荷和 α 多样性并未受到甲流病毒感染的强烈影响。在门水平上,观察到的变化很少。
H1N1 亚型 IAV 感染导致拟杆菌门/厚壁菌门比率下降。
H3N2 和 H1N1 亚型感染期间,还观察到疣微菌(主要由阿克曼氏菌属组成)增加。这些细菌会降解肠道粘液层,瘤胃球菌也是如此(这可能与感染期间粘液增加有关),其数量在 IAV 感染期间也会增加。IAV 感染后还观察到放线菌减少(主要是由于双歧杆菌科属减少)。
简而言之,大多数研究发现γ变形菌(潜在致病性大肠杆菌)增加,少数情况下发现芽孢杆菌类减少。微生物群分析还强调了厚壁菌门内部的变化。综上所述,感染过程中下列菌群数量相对较多:
总体而言,来自流感小鼠模型的数据表明,感染促进了潜在有害细菌种类的出现,例如γ变形菌门和粘液降解细菌的成员。相反,感染似乎会抑制促进健康的细菌(如乳酸杆菌、双歧杆菌和分段丝状细菌)的生长。
此外,小鼠体内 IAV 感染与分节丝状细菌(梭菌科)比例下降有关。这种细菌与肠上皮细胞密切相互作用,对于宿主抵抗啮齿类柠檬酸杆菌等肠道病原体具有重要作用。
流感感染→SFB及乳杆菌减少→肠道损伤
在流感感染的小鼠模型中,研究人员发现,虽然肠道内的细菌总数没有减少,但分段丝状菌(SFB)和乳杆菌/乳球菌的数量减少,同时肠杆菌科细菌增加。有趣的是,虽然 SFB 之前已被证明可以诱导 Th17 细胞 ,但感染流感的小鼠小肠和结肠中的 IL-17A 水平和 Th17 细胞数量增加,这似乎导致肠道损伤。
在这项研究中,流感感染前的抗生素治疗减轻了肠道损伤的程度,但没有改善肺部损伤,这表明肠道菌群失调导致局部炎症,而不是全身炎症。
活病毒感染→变形菌增加→结肠炎易感性增加
注射减毒流感活疫苗(LAIV),表明这些变化需要活病毒感染。变形菌的增加似乎是由 I 型干扰素 (IFN) 介导的,它不仅耗尽了厌氧菌,而且还增加了继发性沙门氏菌结肠炎的易感性。然而,热量限制也会导致变形菌门的相对丰度增加,以及拟杆菌门与厚壁菌门的比例增加,这增加了流感期间口服摄入量减少可能导致微生物组变化的可能性。
全身炎症信号介导肠道微生物变化
还表明,流感感染通过招募到肠道的肺源性 T 细胞产生的 II 型干扰素来改变肠道微生物群组成。因此,肠道微生物组的变化似乎不是由直接病毒效应引起的,而是由从肺部传播并触发肠道局部炎症反应的全身炎症信号引起的。
肠道微生物群可以调节肺部免疫活性
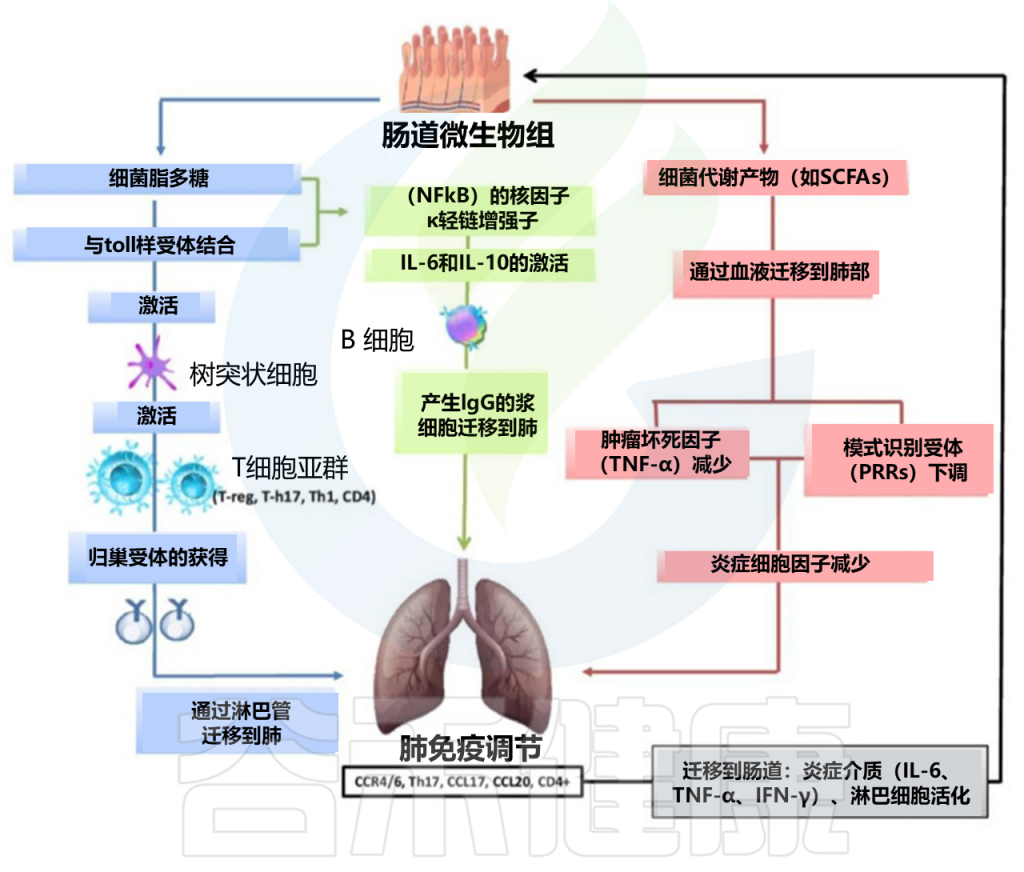
doi: 10.3390/biology9100318
肠道微生物群可以调节肺部免疫活性的双向假设:脂多糖 (LPS) 能够与肠粘膜上存在的TLR结合,从而激活树突状细胞,从而有利于各种 T 细胞的激活(T-reg、T-h17、Th-1、CD4);随后,获得角受体(CCR6、CCR9、CCR4、a4b7)并通过循环淋巴流迁移至肺部(CCR4/6Treg、CCR4/6Th17、CCR4/6Th1、CCR4/6CD4 +)。它还激活(IL-18、INF-y、TNF-a、TGF-b、IL-4、IL-1),这些细胞将进入循环(INF-y、TNF-a、IL-6)。
核因子 kappa-轻链增强子 (NFkB) 激活 IL-6、IL-10 以及 IgA 和 IgG 浆细胞 B 细胞的产生以及 IgG 向肺部的迁移。在肺部,CCL17、CCL20 增加,并且存在 CCR4/6、CD4+、CCR4/6 和 Th17。
细菌代谢物(例如,短链脂肪酸)通过血流迁移至肺部,导致模式识别受体(PRR)下调,从而减少炎症细胞因子(IL-1、IL-12、IL-18)、肿瘤坏死因子 α (TNF-α)、干扰素γ (IFNγ) 和粒细胞-巨噬细胞集落刺激因子 (GM-CSF)。
反过来,在疾病发生时,肺部会将炎症介质和淋巴细胞输送到肠道。
除了(不变)自然杀伤 T 细胞的配体之外,粘膜相关不变 T (MAIT) 细胞的配体合成也可能在病毒呼吸道感染期间发生改变。事实上,细菌共生体,包括属于拟杆菌门和变形菌门的物种,以及病原菌,可以产生维生素 B2 和维生素 B9 代谢物,已知这些代谢物可作为 MAIT 细胞的激动剂或拮抗剂。
关于 MAIT 细胞在粘膜免疫中的关键作用,在病毒呼吸道感染期间改变 MAIT 细胞配体的合成可能对疾病结果产生重要影响,这一假设需要进行研究。
呼吸道合胞病毒和肠道菌群
在小鼠模型中,RSV 导致感染后第 7 天肠道微生物群多样性(但不是丰度和 α 多样性)发生显着改变,拟杆菌门增加,厚壁菌门减少。
拟杆菌门的增加主要是由于拟杆菌科和S24-7科的增加,而厚壁菌门丰度的减少与毛螺菌科和乳杆菌科的减弱有关。RSV 感染对肠道微生物群代谢活动的影响很少被讨论。在一项研究中,RSV 感染主要增加脂质代谢,包括鞘脂、多不饱和脂肪酸和 SCFA 戊酸酯。
冠状病毒和肠道微生物群
一项研究通过 RNA 鸟枪法宏基因组学测序揭示,在患有高感染率的 COVID-19 患者的粪便样本中存在机会性细菌病原体。
相比之下,粪便样本中 SCFA 和色氨酸生产者含量丰富,具有低至无 SARS-CoV-2 感染性的特征。有趣的是,具有高 SARS-CoV-2 感染性的粪便具有更高的微生物组功能,用于核苷酸从头生物合成、氨基酸生物合成和糖酵解。
值得注意的是,在 COVID-19 患者中还描述了机会性真菌病原体(曲霉属和念珠菌属)的过度生长。有趣的是,流感 (H1N1) 和 SARS-CoV-2 患者之间的相似点和差异。核心肠道微生物特征和相关代谢物的发现可以作为潜在的诊断标志物。
急性病毒性呼吸道感染期间肠道微生物群发生变化的原因有多种:这些可能包括炎症细胞因子的释放和食物摄入量的减少。
感染病毒→食欲不振→肠道菌群紊乱
在小鼠中,感染 IAV 或 RSV 会导致体重大幅减轻,减轻幅度为初始体重的 10% 至 20%,并在感染后 5-7 天达到峰值。这种体重减轻主要是由于食欲不振。
众所周知,食物和卡路里摄入量的减少会扰乱肠道微生物群。配对喂养实验的结果清楚地表明,食物摄入量的迅速下降模拟了感染期间观察到的肠道微生物群的变化,即疣微菌门、α变形菌纲和副杆菌属的丰度增加,而毛螺菌科、瘤胃球菌属和乳杆菌属的丰度减少,尽管也观察到差异(梭菌目)。
纤维摄入量减少影响结肠上皮细胞代谢
纤维摄入量的减少一致,配对喂养的小鼠 SCFA 浓度较低。其中,丁酸盐为结肠上皮细胞提供能量,促进上皮氧消耗,从而导致肠腔缺氧。由于食物(纤维)摄入量减少而导致短链脂肪酸的下降可能会改变上皮细胞的代谢。
炎症细胞因子对肠道菌群的影响
病毒感染期间食欲不振的主要原因之一是炎症细胞因子的过量产生,包括肿瘤坏死因子α。
RSV 感染期间这种细胞因子的中和减少了体重减轻,并部分减弱了肠道微生物群的扰动。同样,在 RSV 感染期间消耗产生细胞因子的 CD8 + T 细胞可减少食欲不振并逆转肠道微生物群的变化。
I 型和 II 型干扰素是宿主抗病毒反应的重要组成部分——尤其是在流感期间。这些细胞因子强烈扰乱肠道微生物群。相对于野生型小鼠,I 型 IFN 受体缺陷的小鼠表现出变形菌(埃希氏菌属)繁殖减少,相反,分节丝状细菌的比例升高。
同样,IAV 感染期间 IFN-γ 的消耗恢复了分段丝状细菌(在 IFN-γ 活性小鼠中减少)、乳杆菌属(减少)和肠杆菌科(增强型)。因此,炎症细胞因子和食欲不振可能会导致病毒性呼吸道感染期间肠道菌群失调,至少在小鼠系统中是这样。
氧气梯度变化对肠道菌群的影响
其他机制也可能受到牵连。肠道炎症,例如由于浸润的 CD4 + T 细胞或全身性 IFN 释放而引起,已知会改变上皮细胞的代谢,从而导致一组新的营养物质的积累,而居住在肠腔中的微生物则需要这些营养物质竞争。随着氧气可用性的增加,这种现象似乎可以解释从专性厌氧菌到兼性厌氧菌(例如变形菌肠杆菌科)的转变。
缺氧是呼吸道病毒感染急性期的主要临床症状,包括 COVID-19 患者。它还与慢性肠道损伤有关。关于氧气在肠道稳态中的作用,包括微生物群的组成和功能,由于 SCFA 的可用性而造成的氧气梯度破坏,可能在呼吸道病毒感染期间肠道菌群失调和胃肠道疾病中发挥作用。呼吸道病毒感染期间肠道(上皮)免疫功能缺陷(抗菌肽产生减少)也可能参与生态失调。
病毒性呼吸道感染期间的“肠肺轴”
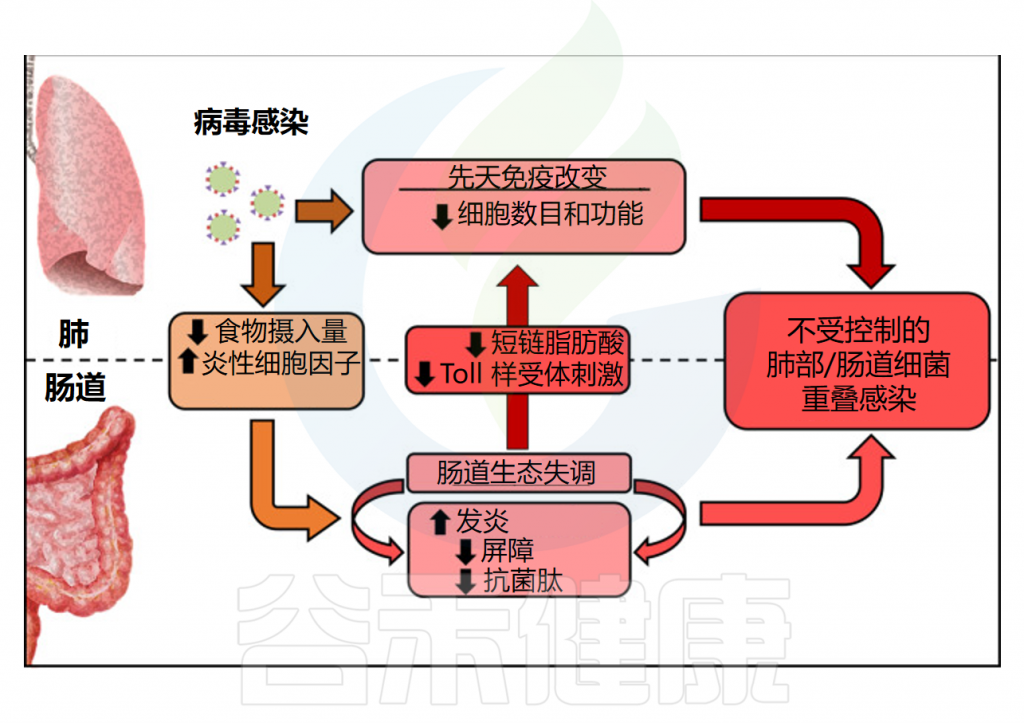
Sencio V, et al. 2021,14:296-304.
总的来说,病毒性呼吸道感染,例如流感,会改变肺部免疫和基质(上皮)细胞的功能,从而导致继发性细菌感染。与此同时,全身炎症细胞因子、食欲不振(纤维减少)、氧水平变化和上皮代谢改变会破坏肠道微生物群的组成和功能。
这些改变会导致肠道炎症、上皮屏障破坏和抗菌肽 (AMP) 产生减少。然后发生继发性肠道感染。上皮渗漏可能会增强细菌易位并引发全身炎症和器官功能障碍。病毒性呼吸道感染引起的菌群失调还会导致微生物相关分子模式的产生减少,包括 Toll 样受体 (TLR) 和核苷酸寡聚结构域 (NOD) 样激动剂以及短链脂肪酸等微生物代谢产物,从而降低抗菌肺部免疫力。因此,呼吸道病毒感染通过改变肠道稳态导致细菌重复感染。
肺和肠道的相互联系在 SARS-CoV-2 感染期间可能特别相关。
消化道可能是病毒复制和活动的场所
与 IAV 和 RSV 的情况相反,在冠状病毒感染(包括 SARS-CoV-2 感染)期间,在肠道中检测到病毒 RNA。大约一半的 COVID-19 患者的粪便中含有 SARS-CoV-2 RNA,即使呼吸道中不再发现它。
重要的是,在 COVID-19 患者的粪便样本中检测到传染性病毒,表明消化道可能是病毒复制和活动的场所。
同样,最近几项使用人类小肠类器官的研究表明 SARS-CoV-2 在肠细胞中复制。因此,局部病毒复制可能会扰乱当地生态系统,导致肠道微生物群组成和功能发生变化。
ACE2表达下调与肠道微生态变化
病毒刺突蛋白与细胞表面受体血管紧张素转换酶 II (ACE2) 的结合导致后者的表达下调。关于 ACE2 在维持肠道微生物生态中的关键作用(通过氨基酸运输故障、色氨酸缺乏和抗菌肽产量减少),SARS-CoV 期间缺乏可用的 ACE2 很可能-2感染在生态失调中起着至关重要的作用。
粪菌移植实验表明,从流感 (H7N9) 感染后幸存的小鼠中收集的肠道微生物群可以为受到 IAV 攻击的受体(未接触过的)小鼠提供保护。这种保护作用是否适用于其他 IAV 亚型和其他呼吸道病毒尚不清楚,值得未来研究。因此,呼吸道病毒感染引起的肠道微生物群组成和功能活动的改变可能会产生有益的影响。然而,如下所述,急性呼吸道病毒感染引起的菌群失调也会导致有害影响并参与感染的结果。
肠道微生物群对于维持上皮完整性和调节性 T 细胞的发育至关重要。这一关键作用主要取决于微生物群的代谢物。
在稳态期间,肠道发挥相对抗炎的免疫状态
肠道微生物群的扰动会导致多种肠道疾病,例如炎症性肠病、肠易激综合征、乳糜泻和结直肠癌。根据这些观察结果,研究人员调查了病毒性呼吸道感染期间观察到的肠道菌群失调对肠道稳态和功能可能产生的负面影响。
病毒性呼吸道感染对肠道的影响
如上所述,病毒性呼吸道感染,包括 IAV、RSV 和冠状病毒感染,可诱发胃肠炎样症状,如腹痛、恶心、呕吐、腹泻。
感染 IAV 的小鼠出现肠道损伤,包括结肠长度缩短(炎症标志物)、小肠粘膜层消失和轻度腹泻。同时,观察到炎症细胞因子和 IFN 刺激基因的 mRNA 表达增强。
肠道屏障的破坏
肠道的一个重要特征是其屏障功能;在健康条件下,这可以防止微生物成分的过度扩散。许多疾病与屏障功能破坏有关,从而导致细菌移位、全身炎症和休克。IAV 对肠道屏障特性的影响值得深入研究。就RSV而言,除了粪便中脂质运载蛋白2(肠道炎症标志物)水平升高之外,在小鼠模型中没有观察到明显结肠炎症的组织学证据。
肠道菌群改变,可能是炎症的结果,也可能是炎症的原因
人们可能会质疑,在病毒性呼吸道感染的情况下,微生物群的改变是否会引发肠道炎症,或者炎症是否会导致菌群失调。这是一个“先有鸡还是先有蛋”的情况。如上所述,炎症可能会影响肠道微生物群的组成。另一方面,失调的微生物群可能直接引发和维持肠道疾病。
事实上,IAV 感染期间的抗生素治疗(以限制肠杆菌科细菌的增加)可以减轻肠道炎症。此外,粪便移植实验表明,经历过 IAV 的微生物群触发了辅助 T17 细胞的局部积累,随后引起肠道炎症。
与肠杆菌科细菌一样,粘液降解细菌(例如瘤胃球菌和阿克曼氏菌)也可能是肠道炎症维持和/或放大的重要因素。事实上,已知这些细菌会侵蚀结肠粘膜并促进肠腔细菌与肠上皮之间的相互作用。反过来,这会导致炎症和屏障功能受损。
总体而言,病毒性呼吸道感染可能通过改变共生体平衡来影响肠道稳态。肠道疾病的性质以及感染对肠道生理学(例如,摄入的食物和液体的消化和吸收)、代谢和免疫稳态的影响仍有待详细分析。
病毒性呼吸道感染后肠道功能的改变可能会对健康产生直接和长期的影响。例如,肠道屏障对于维持体内平衡至关重要,任何扰动都可能导致细菌成分的系统性传播,从而导致有害的健康结果。
肠道微生物群通过直接微生物拮抗和刺激宿主效应反应(例如抗菌肽)来局部控制病原性感染至关重要。生态失调会增加发生肠道细菌感染的风险。动物模型实验表明,IAV 增强了肠沙门氏菌Thyphimurium 继发肠道感染的易感性。这种增强至少部分归因于 I 型干扰素以及相关的抗菌肽和炎症细胞因子释放的减少。
肠道微生物群多样性的减少也可能与继发性肠道感染有关;例如(在小鼠系统中),分段丝状细菌(已知在 T 辅助细胞 17 介导的免疫反应中很重要)的比例在 IAV 感染期间下降。
与此同时,SCFA 生产者比例的下降对于继发性肠道感染可能很重要。已知这些脂肪酸可以控制肠道微生物群的平衡,并防止肠道病原体(如粪肠球菌和鼠伤寒沙门氏菌)的发展。 IAV 感染期间口服补充 SCFA 是否可以降低继发肠道感染仍有待观察。
呼吸道病毒感染的主要并发症之一是继发细菌感染的易感性增加,主要是由肺炎链球菌、金黄色葡萄球菌和流感嗜血杆菌引起的继发细菌感染。这种并发症主要影响儿童和老年人,并导致流行病和大流行期间观察到的发病率和死亡率过高。动物模型实验表明,敏感性增强部分是由于抗菌先天免疫反应受损。
健康的肠道微生物群在肺部免疫中具有有益的生理作用,包括在控制肺炎链球菌或金黄色葡萄球菌等机会性细菌的发育方面发挥积极作用。
研究了流感感染期间肠道微生物群的扰动可能增加肺部细菌重复感染发生率的可能性。事实上,粪菌移植实验表明,IAV 条件下的微生物群损害了肺部对肺炎球菌感染的防御能力。
从机制上讲,乙酸盐(微生物群产生的主要短链脂肪酸)产量的下降影响了肺泡巨噬细胞的杀菌活性。在流感和肺炎球菌双重感染的情况下,补充乙酸盐可以降低细菌负荷,减少肺部疾病,并提高生存率。因此,流感期间肠道微生物群的变化(以及 SCFA 产生的减少)与细菌重复感染有关。该领域的进一步研究可能有助于定义预测标记(例如,系统性 SCFA)和/或开发针对细菌重复感染的治疗方法,例如通过益生元和/或益生菌利用肠道微生物群的力量。
值得注意的是,除了肠道微生物群之外,与急性病毒性呼吸道感染相关的呼吸道微生物组成和功能的变化也可能在细菌重复感染中发挥重要作用。这种局部生态失调可能会改变微生物间相互作用的动态,从而增强潜在致病细菌物种的增殖。局部微生物代谢输出的变化也可能降低肺部对继发细菌感染的防御能力。
总的来说,病毒性呼吸道感染(至少是流感)期间的肠道菌群失调会影响肺部的细菌重复感染。微生物变化是否会影响病毒性呼吸道感染期间的其他疾病结果,例如急性呼吸窘迫综合征、脓毒症和多器官功能障碍,仍然是一个悬而未决的问题。
流感和其他呼吸道病毒感染后常常导致细菌性肺炎,这种现象在历史和现在上已造成大量死亡。病毒感染能导致呼吸道结构和功能的损伤,改变呼吸道的微生物组,增加细菌的侵袭和定植机会,从而促进细菌性肺炎的发生。研究表明,病毒感染不仅影响上呼吸道(URT)的微生物群落结构,而且还可能促进下呼吸道(LRT)的细菌定植,从而增加了肺炎的风险。尽管在临床上普遍使用抗生素以防治细菌性并发症,但病毒与细菌之间的相互作用仍可能在病程中发挥作用。
呼吸道病毒感染后引发细菌性肺炎的机制是复杂的,涉及宿主免疫系统的变化、微生物组的改变以及病原体之间的相互作用等因素。以下是一些可能的机制:
1
免疫系统损伤
病毒感染,如流感病毒或冠状病毒感染,会损伤宿主的免疫系统,特别是呼吸道黏膜的屏障功能和局部免疫应答。这使得细菌更容易侵犯和定植在下呼吸道,从而导致细菌性肺炎。
2
微生物组改变
病毒感染可以改变呼吸道的微生物群落结构,这可能破坏了正常的微生物群平衡,使得某些致病细菌(如肺炎链球菌、葡萄球菌和流感嗜血杆菌)得以增殖和引起感染。
3
病原体协同作用
一些研究表明,病毒和细菌之间可能存在协同作用,病毒感染可能会促进细菌的黏附和侵袭能力,或者细菌可能利用病毒感染后宿主免疫应答的变化来促进自己的生长。
4
炎症反应
病毒感染引起的炎症反应可能会损害呼吸道的细胞,导致细胞死亡和组织损伤,这为细菌提供了更多的营养物质和一个更易于感染的环境。
5
抗生素使用
在治疗病毒感染的过程中,过度或不当使用抗生素可能会导致抗生素敏感的细菌被清除,而抗生素耐药的细菌得以生长,这可能增加了细菌性肺炎的风险。
6
肠道微生物组的改变
感染促进了潜在有害细菌种类的出现,例如γ变形菌门和粘液降解细菌的成员。相反,感染会抑制促进健康的细菌(如乳酸杆菌、双歧杆菌和分段丝状细菌)的生长。与此同时,全身炎症细胞因子、食欲不振(纤维减少)、氧水平变化和上皮代谢改变会破坏肠道微生物群的组成和功能。
7
宿主因素
宿主的年龄、基础疾病、免疫状态等因素也会影响病毒感染后细菌性肺炎的风险。
8
继发细菌感染
在病毒性疾病的紧急情况下,注意力最初集中在原发感染的临床管理上,但必须考虑患者在初次感染期间或之后发生的继发细菌感染。此外,混合感染(同时感染另一种病毒或细菌病原体)也可能发生,并最终导致相同的结果:患者患有由两种不同病原体引起的并发症。
未来诊断细胞性肺炎的项目通常需要综合临床表现、影像学检查以及微生物学检测。微生物学检测可能包括痰液培养、血液培养、支气管肺泡灌洗液(BAL)或粪便培养或分子检测。这些检测有助于确定引起肺炎的具体细菌种类,并为抗生素的选择提供指导。
在干预和管理方面,预防继发性感染策略包括:
改善免疫力
确保患者获得适当的营养和休息,以支持免疫系统的正常功能。对于易感人群,如慢性疾病患者,应考虑接种针对特定细菌的疫苗(如肺炎球菌疫苗)。
这些营养素在免疫健康中发挥作用:
β-胡萝卜素
β-胡萝卜素存在于植物性食品中,如红薯、菠菜、胡萝卜、芒果、西兰花和西红柿。
维生素 C
富含维生素 C 的食物,包括柑橘类水果、浆果、甜瓜、西红柿、青椒和西兰花。
维生素 D
维生素 D 存在于富含脂肪的鱼类和鸡蛋中。牛奶和 100% 添加维生素 D 的果汁也是很好的来源。
锌
锌更容易从牛肉和海鲜等食物中吸收,但也存在于植物性来源中,包括麦芽、豆类、坚果和豆腐。
益生菌
益生菌是促进健康的好细菌。发酵乳制品(例如酸奶)和发酵食品(例如开菲尔和泡菜)中有。
蛋白质
蛋白质来自动物和植物来源,包括牛奶、酸奶、鸡蛋、牛肉、鸡肉、海鲜、坚果、种子、豆类和扁豆。
抗病毒
对于治疗某些呼吸道病毒感染,如病毒感染,可以使用抗病毒药物来减少病毒复制,从而降低继发细菌性肺炎的风险。
维持微生物群平衡
使用益生菌和益生元可能有助于保持或恢复正常的微生物和落平衡。这可以通过补充或通过饮食调整来实现,例如增加富含纤维或益生菌的食物来促进有益菌的生长。
抗生素的审慎慎用
在病毒感染的患者中,不宜过分使用抗生素,以免造成不必要的抗生素抗性和抗生素失衡。只有在明显有细菌感染或经过微生物学验证的情况下,才应使用抗生素。
症状管理和支持治疗
针对呼吸道病毒感染的症状进行管理,如使用退热药、镇咳药和解痉药。同时,保持良好的水分和营养平衡,维生素B族,氨基酸等,必要时提供氧气支持。
监测和早期识别
对于高风险患者群体,应进行定期监测,以便于早期识别继发性细菌性肺炎的迹象,及时进行干预。
通过这些策略的综合应用,可以有效地管理呼吸道病毒感染后的气道和肠道微生物组的变化,并预防继发性肺炎的发生。然而,每个患者的具体情况可能不同,因此,治疗计划应根据个体的需求和病情进行个性化调整。
主要参考文献:
Hanada S, Pirzadeh M, Carver KY, Deng JC. Respiratory Viral Infection-Induced Microbiome Alterations and Secondary Bacterial Pneumonia. Front Immunol. 2018 Nov 16;9:2640.
Lee KH, Gordon A, Foxman B. The role of respiratory viruses in the etiology of bacterial pneumonia: An ecological perspective. Evol Med Public Health. 2016 Feb 15;2016(1):95-109.
Kaul D, Rathnasinghe R, Ferres M, Tan GS, Barrera A, Pickett BE, Methe BA, Das SR, Budnik I, Halpin RA, Wentworth D, Schmolke M, Mena I, Albrecht RA, Singh I, Nelson KE, García-Sastre A, Dupont CL, Medina RA. Microbiome disturbance and resilience dynamics of the upper respiratory tract during influenza A virus infection. Nat Commun. 2020 May 21;11(1):2537.
Sencio V, Machado MG, Trottein F. The lung-gut axis during viral respiratory infections: the impact of gut dysbiosis on secondary disease outcomes. Mucosal Immunol. 2021 Mar;14(2):296-304.
Manna S, Baindara P, Mandal SM. Molecular pathogenesis of secondary bacterial infection associated to viral infections including SARS-CoV-2. J Infect Public Health. 2020 Oct;13(10):1397-1404.
Santacroce L, Charitos IA, Ballini A, Inchingolo F, Luperto P, De Nitto E, Topi S. The Human Respiratory System and its Microbiome at a Glimpse. Biology (Basel). 2020 Oct 1;9(10):318.
谷禾健康
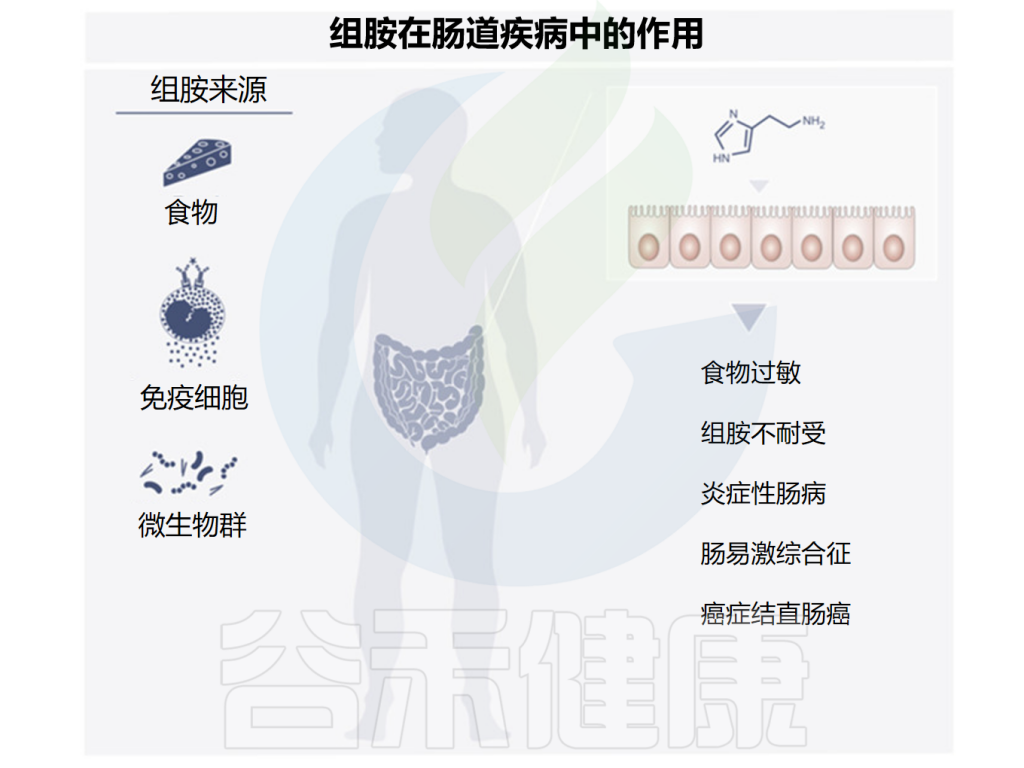
组胺是人体内的一种生物胺,最为人们所熟知的作用是作为过敏反应的介质,但也是神经系统、肠道、皮肤和免疫系统中重要的信号分子。
花粉之所以会引起人体过敏反应,并不是因为花粉本身,而是由于花粉颗粒携带了能引起过敏的抗原。接触过敏源后,体内的组织会释放组胺,作为一种神经递质,组胺会使毛细血管和微静脉的管壁通透性增加,导致局部组织水肿,造成皮肤发红、疹块;也会使气管平滑肌收缩,造成呼吸道狭窄,引起气喘、呼吸困难。
组胺主要由肥大细胞和嗜碱性粒细胞等免疫细胞产生和释放。一些细胞如树突状细胞或T细胞可以表达组氨酸脱羧酶,这是一种在刺激后合成组胺的酶。
尽管如此,一些食物中的微生物群和部分人体肠道微生物群也可以分泌组胺。表明组胺在肠道免疫调节中的潜在作用。事实上,最近的一项研究表明,组胺可以通过抑制肠道中白细胞介素18的产生来发挥抗炎作用。
组胺在免疫反应、神经传递和过敏反应中发挥着重要作用。在某些浓度范围内,组胺起着保护作用,对于维持健康状态至关重要。例如组胺可以刺激胃酸分泌,有助于消化;还会影响排便情况;组胺作为重要的神经递质,参与睡眠调节和体温调节。
然而,在较高浓度下,会导致包括组胺中毒和组胺不耐受或扩张血管,影响血压,导致免疫调节紊乱、胃肠道症状和神经系统疾病。组胺中毒是在食用被大量组胺污染的食物后发生的。如果个体由于基因突变而缺乏生物胺降解能力,或者正在服用单胺氧化酶抑制剂等抗抑郁药来减缓胺的降解,他们就会更容易出现组胺不耐受的症状。
组胺在体内与相应受体(H1、H2、H3、H4)结合而介导的各种症状。不同类型的组胺受体与该化合物之间的相互作用可能会引起多种影响,例如H1R主要介导过敏反应,而H2R具有抗炎作用,而H3R主要影响睡眠、认知障碍等精神健康。
肠道内的组胺对许多细胞过程的广泛影响会导致各种胃肠道疾病,包括食物过敏、肠易激综合征、炎症性肠病、结直肠癌等。谷禾将在本文带大家了解组胺在免疫及各种肠道疾病中的保护或致病作用。
组胺(C5H9N3)是一种重要的生物胺类神经递质,于20世纪初首次发现。从那时起,它的功能开始被逐渐发现并得到越来越详细的描述。
组胺存在于多种细胞类型中,是人体的关键信号分子,具有多种功能。例如作为神经递质或免疫反应调节剂。
拓展知识:组胺的发现
1903年,一位叫威廉·邓巴的医生证明了吸入花粉后的过敏反应并不是由花粉本身引起的,而是机体对花粉的反应引起的一种毒素的释放所造成的。
后来,亨利·戴尔于1910年在研究黑麦的毒性时,从麦角菌中提取出一种叫做组织胺的物质。他发现用组胺可以人工诱发荨麻疹。如果将组胺滴入气管,还会诱发哮喘发作。
★ 组胺的重要作用
介导过敏反应
组胺最被人熟知的是在过敏反应中的作用。例如在过敏性鼻炎、荨麻疹和血管神经性水肿等过敏性疾病中发挥重要的病理生理学作用。
当免疫系统对过敏原产生过度反应时,免疫细胞(如肥大细胞和嗜酸性粒细胞)会释放存储在细胞内的组胺。释放的组胺会结合到特定的受体上,引发一系列生理反应,例如瘙痒、肿胀、打喷嚏等。
扩张血管,影响血压
组胺对人心血管系统最突出的作用是扩张小血管。血管扩张使外周阻力降低,血压下降,并伴有潮红、头痛等症状。
组胺还增加毛细血管的通透性,使渗出增加,引起水肿,严重时甚至导致循环血量减少,可能引起休克。组胺对心脏的直接作用包括增强心肌收缩力、加快心率和减慢房室传导。
刺激胃酸分泌
首先,组胺可以刺激胃黏膜细胞中的H2受体,当组胺与H2受体结合时,会激活细胞内的信号转导通路,导致胃壁上的壁细胞释放胃酸。
其次,组胺还可以通过刺激嗜酸性细胞(胃窦细胞)来促进胃酸的分泌。嗜酸性细胞是胃黏膜中的一种细胞类型,它们包含大量的组胺。当胃黏膜受到刺激时,嗜酸性细胞会释放存储的组胺。释放的组胺通过与H2受体结合,刺激壁细胞分泌胃酸。
影响排便
一项研究中,发现细菌产生的组胺与小鼠结肠运动和粪便排出量增加有关,并且组胺受体拮抗剂治疗在很大程度上阻断了细菌组胺对结肠运动的影响。
收缩平滑肌
此外,组胺会诱导平滑肌细胞(包括支气管和肠道)收缩,哮喘患者对组胺比正常人敏感100~1000倍,组胺可引起支气管痉挛导致呼吸困难。组胺收缩胃肠平滑肌和子宫平滑肌还会引起痉挛性腹痛。
重要的神经递质
在中枢神经系统中,组胺作为一种神经递质,参与睡眠调节和体温调节等生理过程。组胺对感觉神经末梢有强烈的刺激作用,尤其对调节痛和痒的神经,该效应由H1受体所调节。
除此之外,有研究发现组胺还会影响伤口愈合、食欲和情绪等方面。
组氨酸是组胺的前体物质
组胺的主要细胞来源是肥大细胞和嗜碱性粒细胞。在细胞的高尔基体中,组胺是通过组氨酸脱羧酶将L-组氨酸氧化脱羧形成的,其辅助因子是吡哆醛(维生素B6)。
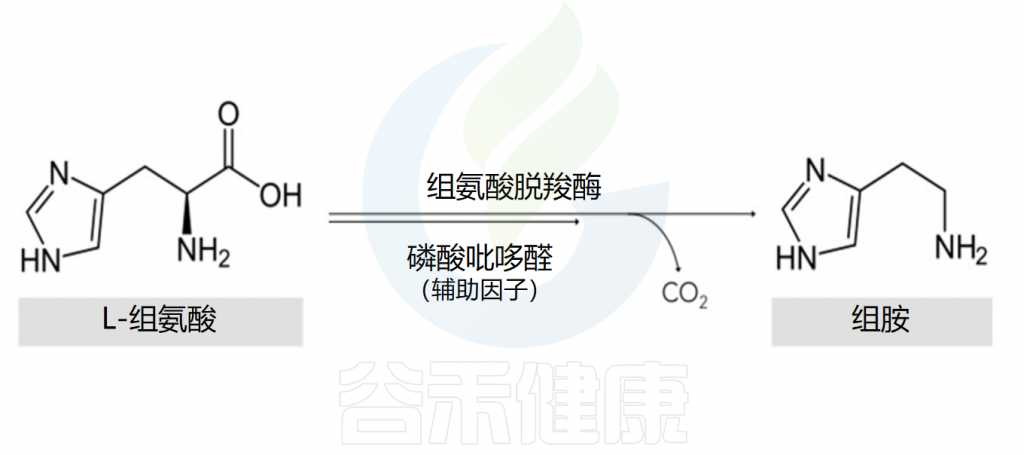
Comas-Basté O,et al.Biomolecules.2020
该反应的结果是组胺的形成,组胺随后与其他胺(如5-羟色胺)、蛋白酶、蛋白聚糖、细胞因子/趋化因子和血管生成因子一起储存在细胞质颗粒中,并在细胞致敏和脱颗粒后释放。
过敏时组胺会大量释放
肥大细胞的脱颗粒和组胺的释放主要是由于特异性抗原与FcRI受体结合以及对非免疫刺激(例如神经肽、补体系统的一部分、细胞因子、血小板活化因子)的反应。
IgE抗体是过敏性疾病期间肥大细胞脱颗粒的介质。IgE与其肥大细胞表面的高亲和力IgE受体的结合称为“致敏”。肥大细胞和嗜碱性粒细胞释放的组胺通过激活四种G蛋白偶联受体,即H1R、H2R、H3R(主要在大脑中表达)和H4R,发挥其生物活性。
细菌感染
某些细菌感染也可以引起组胺的释放。例如,胃幽门螺杆菌感染可以导致胃黏膜炎症,促使组胺释放,进而引发胃酸增多和溃疡等胃部疾病。
药物和化学物质
某些药物和化学物质也可以诱发组胺的释放。例如,非甾体抗炎药(如阿司匹林)和某些麻醉药物以及酒精可以引起组胺的释放,可能导致过敏样反应。
物理刺激
组胺释放也可由多种物理因素引起,如极端温度、创伤、摩擦等。这种情况下的组胺释放通常是局部性的,导致局部组织的充血和炎症反应。
运动和应激
剧烈运动、情绪激动和精神压力等也可以促使组胺的释放。这种情况下,组胺的释放通常是全身性的,可能导致血管扩张、心率增加和其他应激反应。
注:组胺也可以由其他细胞类型(例如胃肠嗜铬样细胞、组胺能神经元、树突状细胞、T淋巴细胞、血小板等)合成和释放。
食物和肠道微生物也是组胺的重要来源
一部分组胺通过食物进入人体或由肠道微生物产生。最受欢迎的富含组胺的食物是鱼和海鲜、发酵食品(如奶酪、葡萄酒、泡菜等)和一些蔬菜(如菠菜、茄子、番茄等)。
组氨酸主要在自溶或细菌过程中产生,因此高浓度的组胺主要存在于微生物发酵产物中。食品中生物胺形成的条件是游离氨基酸的可用性、脱羧酶阳性微生物的存在以及细菌生长和脱羧酶活性的条件。
影响细菌组氨酸脱羧酶活性的因素
细菌分泌脱羧酶受许多因素(例如,发酵碳水化合物、氧气或氯化物浓度的存在)调节。在酸性环境中,氨基酸脱羧酶活性的表达增加。
组氨酸脱羧酶活性较高的细菌种类有:
Morganella morganii,Eschericha coli, Hafnia alvei, Proteus vulgaris, Proteus milabilis, Enterobacter aerogenes, Raoultella planticola, Raoultella ornithinolytica, Citrobacter freundii, Pseudomonas fluorescens, Photobacterium damselae。其中一些细菌具有代谢组胺的能力。
我们将在下面的一个段落中展开讲述肠道中的组胺与微生物群。
在人体内,组胺降解主要有两种途径,涉及二胺氧化酶(DAO)或组胺-N-甲基转移酶(HNMT)。组胺降解酶缺失会导致组胺浓度过高甚至中毒,组胺不耐受患者就是组胺降解酶的酶活性降低或受到抑制,并且不足以灭活食物中的组胺并阻止其进入血液。
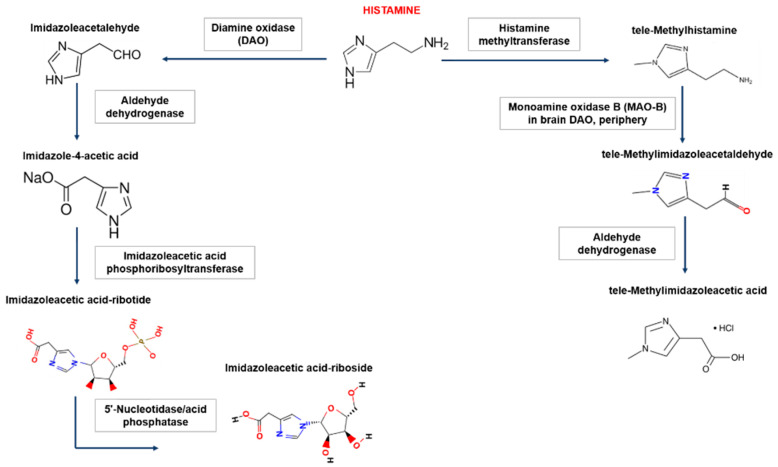
Shulpekova YO,et al.Nutrients.2021
组胺-N-甲基转移酶负责细胞内的组胺降解
组胺-N-甲基转移酶(HNMT)主要负责细胞内组胺的降解。HNMT在肾脏和肝脏以及脾脏、结肠、前列腺、卵巢、脊髓细胞、支气管和气管中的表达最高。一小部分组胺在HNMT的作用下转化为N-甲基组胺。
注:在原始形式中,约2%-3%的组胺被排泄。
二胺氧化酶负责细胞外的组胺降解
二胺氧化酶(DAO)是一种分泌蛋白,负责细胞外组胺的降解。DAO的最大活性记录在小肠、结肠、胎盘和肾脏中。DAO在细胞内囊泡中积累,然后进入细胞外空间,在那里降解组胺。绝大多数组胺通过DAO转化为咪唑乙酸。
保护身体免受组胺过高的侵害
二胺氧化酶执行“屏障功能”,从而限制组胺从肠道进入血液;组胺-N-甲基转移酶通过B族维生素和S-腺苷甲硫氨酸的参与使组胺甲基化,从而调节细胞内的组胺含量。
尽管二胺氧化酶和组胺-N-甲基转移酶这两种酶都存在于肠上皮中,但吸收组胺进入血流的主要障碍是 二胺氧化酶,组胺-N-甲基转移酶在此过程中仅发挥次要作用。在正常情况下,这种酶屏障足以防止组胺被吸收到血流中。
注:二胺氧化酶和组胺-N-甲基转移酶基因的多态性可能是组胺代谢个体差异的基础。在突触中,乙醛脱氢酶在组胺降解中发挥作用。
虽然健康人体内的组胺很快就会被二胺氧化酶降解,但即使在健康人体内,也可能会因摄入组胺含量高的食物(例如鲭鱼)导致血液中组胺浓度升高而出现严重症状——组胺中毒。
• 组胺中毒的症状
组胺中毒症状多种多样,可能包括口腔麻木、头痛、头晕、心悸、低血压、吞咽困难、脉搏微弱、荨麻疹、面部肿胀、潮红、呕吐、恶心和腹泻等。
组胺中毒的症状可能持续数小时或一天,但在极少数情况下,症状可能会持续数天。
• 组胺中毒的主要原因是食用鱼类和海鲜
不仅是鲭鱼,一些非鲭鱼(鲯鳅鱼、蓝鱼、沙丁鱼等)都含有大量组胺,此外发酵产品(奶酪、葡萄酒、罐头食品、腌制蔬菜和一些饮料)中组胺和血清素的含量也较高。
一项针对科学报告的分析显示,在98%的病例中,组胺中毒的原因是食用鱼类和海鲜,其余2%是由于食用奶酪等发酵食品。
鱼组织中的组胺是由多种细菌对游离组氨酸进行转化而产生的,这些细菌包括摩氏摩根菌(Morganellamorganii)、产气肠杆菌 (Enterobacter aerogenes), Raoultella planticola, Raoultella ornithinolytica和Photobacterium damselae。
鱼分解产生的其他物质会增强组胺的毒性并促进吸收
组胺中毒的发病机制不能仅用组胺的作用来解释。与口服等效剂量的纯组胺相比,鲭鱼毒性较高。还提出了其他机制,例如控制组胺的酶的抑制或增强、导致肥大细胞脱颗粒的物质的存在、其他组胺激动剂的存在。
鱼分解过程中产生的其他物质(尸胺和腐胺)会增强组胺的毒性,这些物质还会抑制肠道单胺氧化酶和二胺氧化酶,或从与粘膜粘蛋白的结合中释放组胺,促进其吸收。
组胺中毒后的干预及治疗
在生理值范围内。组胺中毒的病程通常是轻微的、自限性的、持续时间较短且不需要治疗。
对于严重的组胺中毒病例,需要进行治疗干预——支持性容量疗法和氧气疗法,并给予口服抗组胺药和支气管扩张剂。
组胺不耐受是由于组胺摄入量与机体清除能力不相称,导致组胺过度蓄积,并与相应的物质结合而出现症状的病理过程。
与组胺中毒不同,组胺中毒的严重程度与营养物质中组胺的含量成正比,组胺不耐受的发病机制与先天性或后天性缺乏中和组胺的酶有关。在健康患者中,肠上皮细胞具有由二胺氧化酶和组胺-N-甲基转移酶创建的酶屏障。该屏障可防止血液中外源性组胺的过度吸收。如果这些酶被抑制或减少,即使摄入少量的组胺也可能出现组胺不耐受的症状。
• 组胺不耐受的症状
组胺不耐受的神经系统症状包括头痛。除头痛外,其他重要症状包括弥漫性胃痛、绞痛、胀气和腹泻。
组胺不耐受的患者,在摄入组胺或酒精含量高的食物时或摄入后会出现流鼻涕、鼻塞等症状,极端情况下甚至会出现支气管哮喘发作、支气管收缩、咳嗽、喘息并伴有肺功能下降。
组胺不耐受患者还会出现皮肤瘙痒、发红、肿胀甚至荨麻疹。而患有组胺不耐受的女性会出现与月经周期相关的痛经和头痛。
• 影响组胺降解酶活性的因素
在胃肠道疾病(炎症性肠病、寄生虫感染、微生物菌群失调、代谢吸收不良)中,损伤的肠上皮细胞会减少二胺氧化酶的产生,或其他生物胺、酒精或药物也会抑制二胺氧化酶。
二胺氧化酶活性降低可见于慢性肾功能衰竭、病毒性肝炎、晚期肝硬化和慢性荨麻疹(一种典型的组胺相关疾病,对内源性组胺的耐受性降低)的患者。
二胺氧化酶降解能力下降还可能是由于缺乏其辅助因子、维生素B6、维生素C、铜和锌而导致的。
• 肠道微生物会影响组胺不耐受的发生
肠道微生物群也会影响组胺不耐受的发生。最近的研究表明,组胺不耐受可能是由肠道微生物群的改变引起的。肠道中大量的组胺分泌细菌可能会导致组胺不耐受。健康人体内双歧杆菌的数量较多。而在血清二胺氧化酶活性降低的人群中观察到变形菌属数量较多。
组胺不耐受人群存在肠道菌群失调
在组胺不耐受人群中观察到肠道菌群失调,与健康个体相比,与肠道健康相关的普氏菌科(Prevotellaceae)、瘤胃球菌(Ruminococcus)、粪杆菌(Faecalibacterium)和普拉梭菌(Faecablibacterium prausnitzii)的比例明显较低。组胺不耐受人群还具有显著更高丰度的组胺分泌细菌,包括葡萄球菌属和变形杆菌属、属于肠杆菌科的几个未识别属,以及产气荚膜梭菌和粪肠球菌。
需要注意的是,几项研究表明,即使在没有二胺氧化酶缺陷的个体中,大量的组胺产生细菌也会导致肠道内高水平组胺的积累、随后在血浆中的吸收以及不良反应的出现。
低组胺饮食
组胺不耐受疗法的基础是基于低组胺含量食物的消除饮食。一般来说,公认的饮食指南尚未制定;但是,建议避免食用富含组胺的食物如奶酪、油性鱼类和贝类,以及生发酵肉制品、腌菜、发酵豆制品、葡萄酒和啤酒、鸡蛋、巧克力和蘑菇。
避免食用易刺激组胺释放的蔬果
此外,应避免食用会刺激内源性组胺释放的蔬菜和水果(如菠菜、西红柿、柑橘类水果、草莓、茄子、鳄梨、木瓜、香蕉、猕猴桃、菠萝和李子)。


Kovacova-Hanuskova E,et al.Allergol Immunopathol.2015
补充二胺氧化酶
研究还发现补充二胺氧化酶可以改善组胺不耐受患者的生活质量。已开发出基于肠溶猪肾提取物的含二胺氧化酶食品添加剂。豆芽也被用作二胺氧化酶的来源,这种酶的活性是未发芽种子的250倍。
施用抗组胺药物
纠正组胺不耐受的药物方案基于使用组胺受体阻滞剂、肥大细胞膜稳定剂和其他具有抗组胺活性的药物。
在这方面,H1R拮抗剂是最有效的,特别是依巴斯汀。每天服用20毫克该药物,持续12周,46%的患者出现临床缓解。
一些生物活性物质有助于增强组胺降解
维生素C被用作纠正组胺不耐受的佐剂。每日剂量300-500毫克可增强组胺降解并抑制肥大细胞脱颗粒。
天然类黄酮(非瑟酮、山奈酚、槲皮素、芦丁和木犀草素)和活性生物碱小檗碱在体外抑制肥大细胞脱颗粒。这些都有助于减轻组胺不耐受。
虽然组胺含量过低的情况相对较少见,但也可能会对身体产生一些影响,包括:
• 伤口愈合减慢
组胺在人体中起着重要的调节作用,其能够帮助调节纤维细胞生长因子的生成,该成分可以加速局部肉芽的生长,帮助促进伤口的愈合。因此组胺含量较低时,伤口愈合能力也会减弱,速度会随之减慢。
• 消化不良
组胺可以通过刺激胃部,进而起到促进胃酸分泌的效果。若身体中没有组胺,则会导致胃酸减少,无法有效消化分解食物,进而引起消化不良的问题。
• 情绪不良
组胺还可以作为神经递质帮助调节中枢神经系统,而一旦没有组胺,则会影响中枢神经系统稳定性,引起睡眠异常、激素水平紊乱等情况,进而导致情绪不良。
• 血压升高
人体缺少组胺还会导致血压升高。因为组胺可以促进血管扩张,增加血管的通透性,所以具有降低血压的作用。若没有该成分,则血管会由于舒张能力减弱,因此出现血压升高的问题。
• 免疫力下降
组胺对免疫细胞,如吞噬细胞、T细胞、B细胞等,起到增强活性的功效,因此组胺还具有增强免疫功能的作用。若人体缺少该物质,则人体免疫力会下降,无法有效抵抗细菌、真菌等有害物质的侵入。
组胺对人体许多生理功能都可以起到积极作用,但是人体中的组胺也不能过量,否则容易导致过敏反应,出现头晕、皮肤瘙痒、胸闷等不适症状。
此种情况需及时就医,并且在医生的指导下服用抗组胺药物,例如盐酸西替利嗪片、氯雷他定片等药物,进行抗过敏治疗。
★ 组胺的免疫调节因受体的不同而存在区别
先天性和适应性免疫系统的细胞都可以被组胺调节。组胺的调节取决于它与组胺受体的四种亚型的结合,这些亚型按发现时间顺序命名——H1R-H4R,在多种细胞类型中差异表达,进而产生不同的作用。
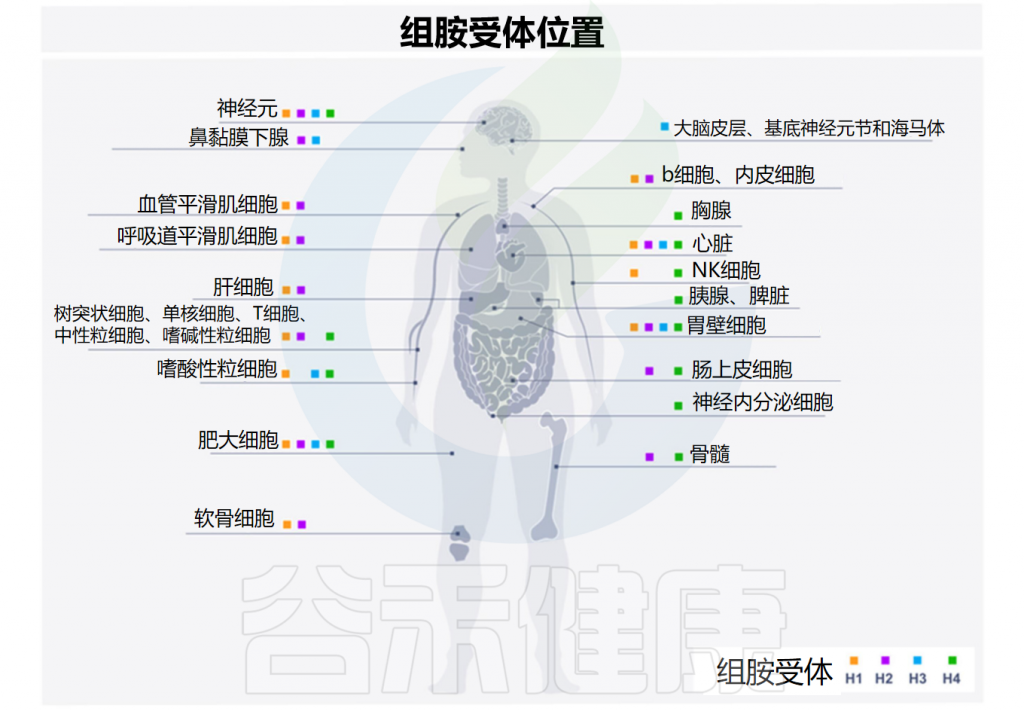
Smolinska S,et al.Metabolites.2022
1
H1R
H1R受体存在于几乎所有组织的内皮细胞和血管平滑肌细胞中(它们的密度在胃肠道中特别高),以及下丘脑、肾上腺髓质和免疫活性细胞(单核细胞、中性粒细胞、T细胞和B细胞)中。
• H1R受体介导的主要作用
H1R刺激的主要作用是炎症、全身血管舒张、血管通透性增加、支气管收缩、回肠收缩和昼夜节律周期调节。
外周H1R介导的影响包括鼻漏、支气管收缩、过敏反应、结膜炎和荨麻疹,而中枢相关的H1R影响包括食物和水摄入的调节、抽搐、注意力和睡眠调节。
• H1R是过敏反应的主要受体
IL-3、IL-4和组胺可上调H1R基因表达,而H1R激活导致许多与过敏性即时型超敏反应相关的特征,例如发红、瘙痒和肿胀。
例如小鼠模型中H1R的激活会诱导IFN(干扰素)产生增加,这与1型辅助性T细胞的增殖有关,并诱导促炎作用。
研究表明,瘙痒因子(例如神经生长因子、信号素 3A)的表达受组胺H1R的调节。在小鼠模型和特应性皮炎患者中,使用H1R拮抗剂可降低IL-31(白细胞介素-31)水平,这与瘙痒的发作有关。
2
H2R
与H1R类似,H2R的表达存在于多种组织和细胞中,包括脑、胃壁细胞、平滑肌细胞、T和B细胞、树突状细胞和心脏组织。H2R受体位于突触后,主要通过环腺苷单正膦(cAMP)传递信号并与Gαs偶联。
不同组胺受体下游的信号通路
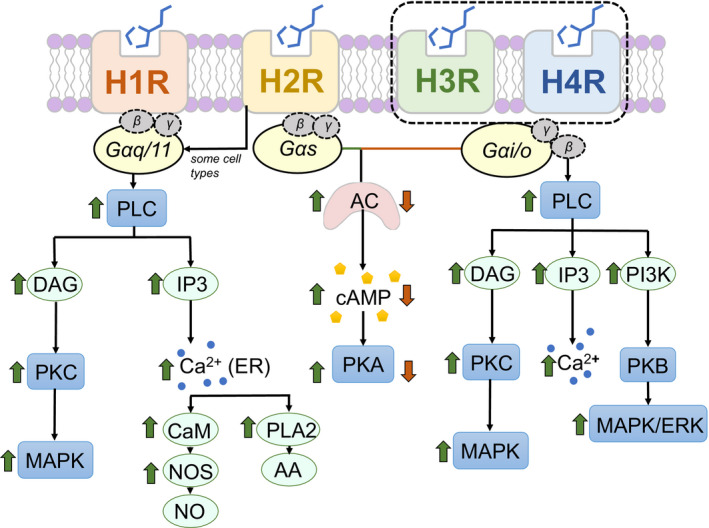
Sudarikova AV,et al.Physiol Rep.2021
H2R刺激的主要作用是外分泌(主要是盐酸)、心动过速、平滑肌细胞松弛、抗炎作用、抗体合成等免疫活动。
• H2R具有抗炎作用
在组胺与H2R结合期间,H2R刺激通过抑制单核细胞或巨噬细胞和肥大细胞产生IL-12、IFN-γ、TNF-α细胞因子,而IL-10分泌增加,从而引起抗炎作用。
• H2R影响对微生物的免疫反应
组胺(内源性和外源性)通过H2R显著改变对微生物的先天免疫反应。特别地,树突状细胞对微生物配体的反应被组胺以H2R依赖的方式显著改变。
H2R敲除的小鼠免疫系统紊乱以及胃缺陷(胃酸分泌减少),此外还观察到认知能力下降和伤害感受异常。
• 注意:不同组胺受体可能具有相反的作用!
H2R以与H1R不同的方式传输信号,H1R和H2R的激活对各种生物过程具有相反的影响。在T细胞介导的免疫反应中,H1R激活促进Th1极化,而H2R激活则抑制Th1极化。
在平滑肌收缩中也观察到H1R和H2R激活的相反作用。H1R和H2R拮抗剂分别抑制和加剧组胺引起的轻度哮喘患者的支气管痉挛。这些数据表明,组胺可能会产生相反的作用,具体取决于被激活的特定组胺受体。
3
H3R
• H3R主要存在于神经系统中
H3R受体在神经系统细胞中表达,特别是在大脑皮层,基底神经节神经元和海马体中。H3R位于含组胺神经元的突触前区域。它们的功能是调节组胺以及其他神经递质(如多巴胺、去甲肾上腺素、γ-氨基丁酸、乙酰胆碱和血清素)的合成和释放。
• H3R受体影响睡眠、认知障碍等精神健康
H3R受体表达和激活的变化在睡眠-觉醒周期障碍、注意力缺陷多动障碍、癫痫和认知障碍以及炎症的发展中发挥重要作用。
H3R缺陷小鼠表现出行为反应和运动的变化,以及伴有肥胖、食欲过盛以及瘦素和胰岛素水平升高的代谢综合征。
• H3R可能会促进炎症反应
H3R刺激可增加促炎活性以及免疫细胞呈递抗原的能力。H3R敲除也可导致神经炎症性疾病的严重程度和T细胞中IFN-诱导蛋白10的表达增加。
因此,组胺H3R拮抗剂的使用有可能用于预防或抑制炎症性疾病(例如呼吸系统疾病)的发展。
4
H4R
H4R受体是最近才发现的,它们的作用尚未完全了解。H4R主要存在于免疫细胞(嗜酸性粒细胞、嗜碱性粒细胞、肥大细胞、自然杀伤细胞、树突状细胞、单核细胞和T细胞)中,也存在于脾脏、胸腺、骨髓、胆管、胰腺、肠上皮细胞和神经内分泌细胞中。
• H4R会增强过敏和炎症反应
与其他类型的组胺受体相比,H4受体在中枢和周围神经系统中表达并不显著。H4R通过与蛋白质Gα/io相互作用介导,参与炎症和超敏反应的发展。
H4R介导的肥大细胞活化,促炎细胞因子和趋化因子IL-6、TNF-α、TGF-β1、RANTES、IL-8、MIP-1α和MCP-1均被表达。研究发现H4R激活显著增强结肠炎、放射性结肠炎、肠道缺血/再灌注损伤和过敏反应的炎症反应。
因此,选择性H4R阻滞剂的开发可能代表了治疗炎症性肠病的一种有前途的方法。
• H4R的激活还会导致瘙痒
H4R的激活已被证明会导致瘙痒。而H1R和H4R拮抗剂的联合治疗可能通过协同抑制瘙痒和皮肤炎症对慢性皮炎产生显著的治疗效果。
此外,H4R通过激活Th2细胞并产生 IL-31,可能引发过敏性皮炎的发生。H4R和H3R的激活增加了乙酰胆碱对肠道蠕动的影响。H4R受体还参与消化性溃疡的形成和癌变。
//小结
如前所述,组胺特异性受体的组织定位、功能和对组胺的亲和力不同。组胺调节广泛的代谢过程,具有促炎和抗炎作用,具体取决于受体亚型和受刺激细胞的类型。
Barcik W,et al.Curr Opin Immunol.2017
组胺对免疫调节的各种作用似乎与其受体及其各自的细胞内信号的差异表达和调节有关。此外,这些受体对组胺的亲和力的差异在组胺和组胺受体药物配体的生物效应中发挥着关键作用。组胺受体在多种病理生理状况中发挥重要作用,是治疗过敏的有效靶点。
组胺因其在速发型超敏反应中的作用而广为人知。在胃肠道内,组胺以相对较高的浓度存在,特别是在炎症过程中。组胺水平的增加改变了宿主与微生物群的免疫相互作用,并导致体内平衡的破坏,导致许多难以应对的肠道疾病的发展。
★ 组胺的水平会影响肠道稳态
肠道中的组胺水平受到宿主过敏和炎症反应的影响,除了宿主微生物群的产生外,还会以某种方式改变降解或合成组胺的酶的活性及其饮食摄入量。
此外,在刺激产生组胺的免疫细胞时,可以增加内源性组胺的水平。所有这些都会影响肠道稳态,导致组胺积累,并影响特定疾病。组胺还可能对肠道寄生虫和细菌感染产生一定影响。
注:除H3R外,所有组胺受体都在人体肠道中表达。从量的角度来看,H4R表达的丰度明显低于H1R和H2R。
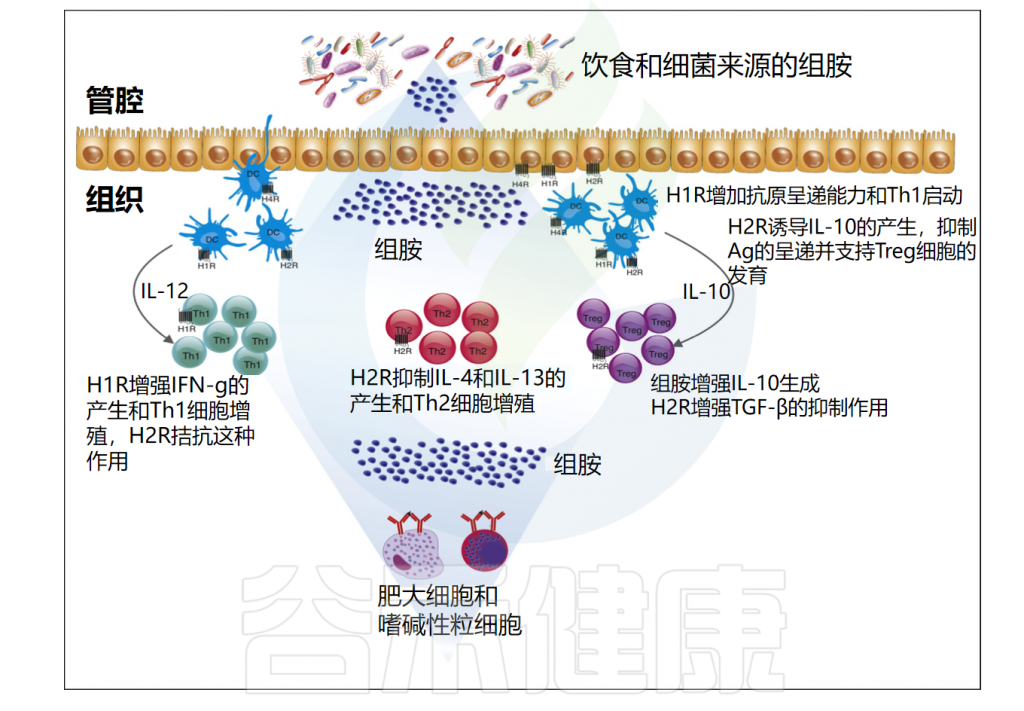
粘膜内的组胺
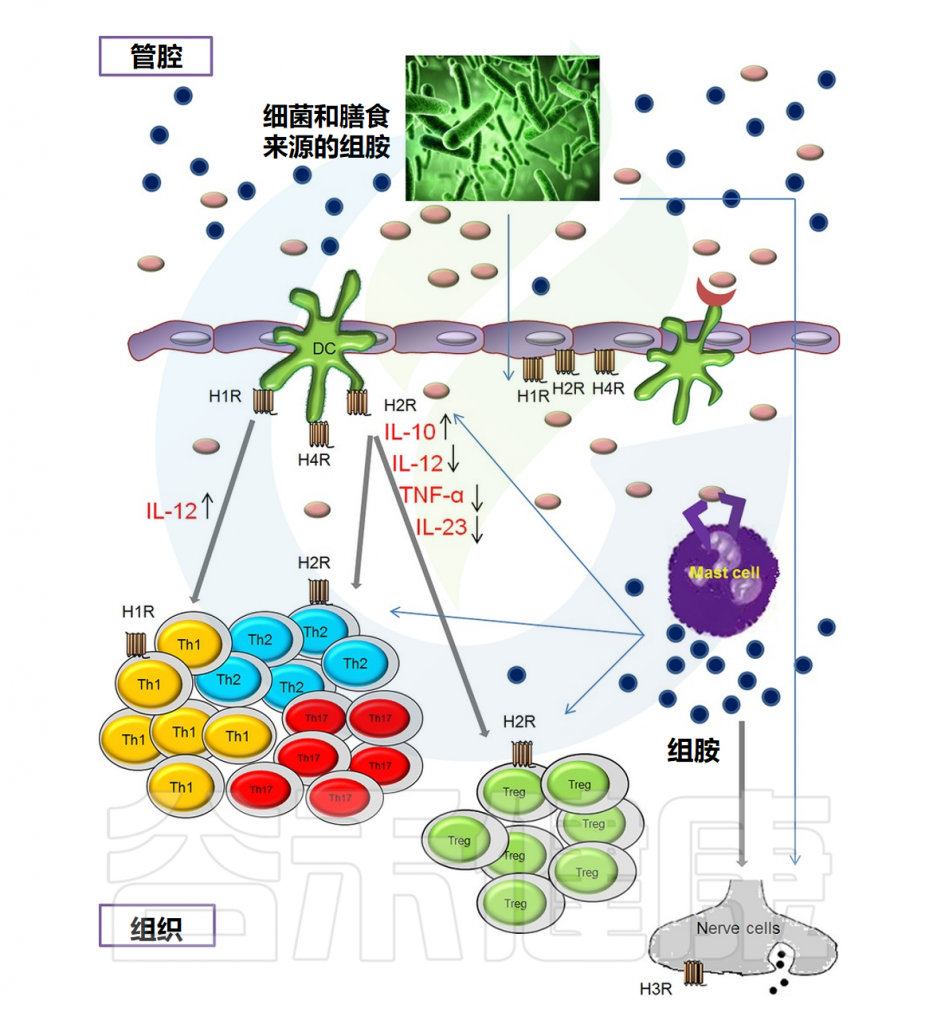
Smolinska S,et al.Allergy.2014
• 食物中的一些菌株会分泌组胺
除哺乳动物细胞外,还发现许多不同的细菌菌株能够在组氨酸通过组氨酸脱羧酶作用后分泌组胺。
最有力的证据就是那些能够在食品中分泌组胺的细菌。它们在奶酪、肉罐头、乳制品以及啤酒和葡萄酒发酵过程中产生组胺。鲭鱼中毒就是在储存或加工过程中处理不当,细菌代谢了鱼类中的组氨酸,导致组胺含量过高。
• 部分肠道微生物也会分泌组胺
迄今为止,对产生组胺细菌的研究主要集中在从食物样品中分离的菌株。肠道微生物群的组胺生成能力直到最近才被研究,数据仍然有限。
2021年发表的一项荟萃分析展示了人类肠道微生物组中具有假定组胺分泌能力的物种 ,其中许多属于广泛报道的组胺产生属,例如摩根菌属(Morganella),乳杆菌(Lactobacillus),葡萄球菌属(Staphylococcus), Photobacterium和巴氏梭菌(Clostridium)。
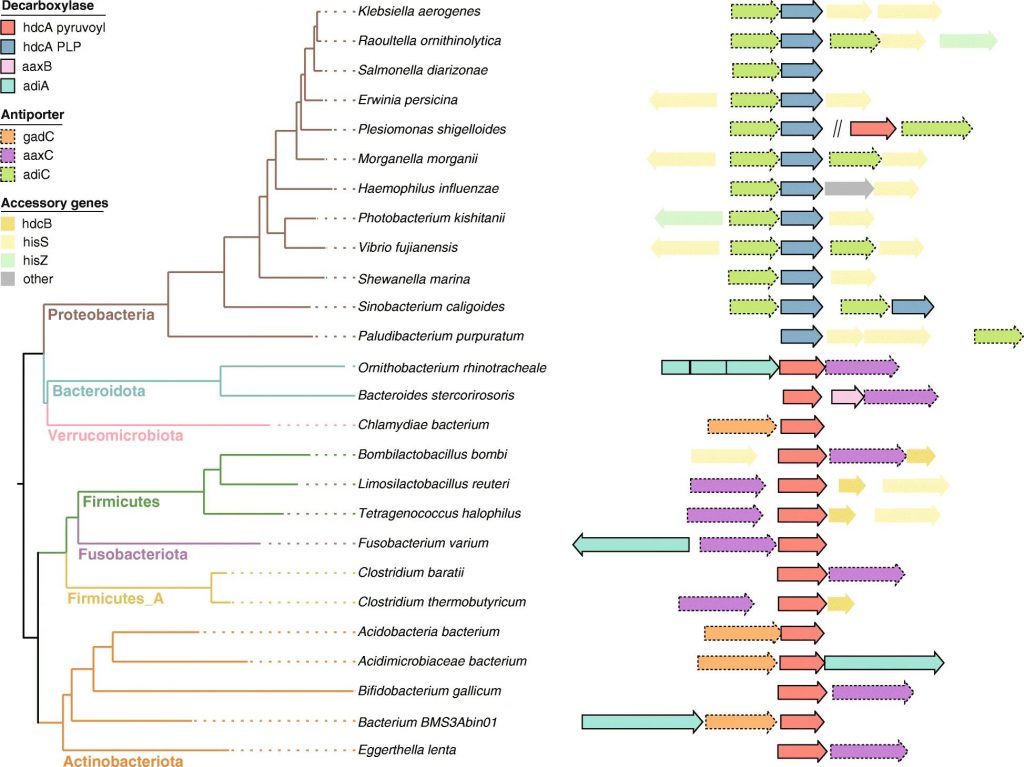
Mou Z,et al.BMC Genomics.2021
• 一些细菌具有组氨酸脱羧酶活性,也可能产生组胺
需要注意的是,一些具有组氨酸脱羧酶活性并与组胺中毒有关的细菌,可能也是潜在的组胺产生细菌。包括:Morganella morganii,
Eschericha coli,
Hafnia alvei,
Proteus vulgaris,
Proteus milabilis,
Enterobacter aerogenes,
Raoultella planticola,
Raoultella ornithinolytica,
Citrobacter freundii,
Pseudomonas fluorescens,
Photobacterium damselae。
证据表明细菌衍生的组胺具有多种影响,例如对宿主健康和食品安全。
• 抑制了促炎细胞因子的分泌
初步体外研究表明,组胺抑制人单核细胞衍生的树突状细胞中趋化因子和促炎细胞因子的分泌。
小鼠研究表明,给予分泌组胺的鼠李糖乳杆菌具有抗炎作用,各种白细胞介素和肿瘤坏死因子α的分泌减少。这种作用在缺乏组胺2受体的动物中消失,表明微生物群衍生的组胺可能具有免疫调节作用。
• 组胺的含量对于免疫效果至关重要
与鼠李糖乳杆菌相比,另一种乳杆菌能够分泌大约100倍多的组胺,服用这种乳杆菌会导致动物体重减轻和健康状况恶化。谷禾认为,微生物分泌的组胺量可能是决定其作用性质的关键。
最近的研究还表明,肠道中细菌释放的组胺不仅会引起宿主免疫系统的局部调节,而且还会在远处的粘膜部位(例如肺部)产生免疫学后果。例如成年哮喘患者肠道中分泌组胺的细菌丰度有所增加。
由于许多食物中含有组胺,一部分肠道微生物群也可以产生组胺,研究发现组胺在肠道疾病中可能发挥多种作用,下面讲述了一些与组胺相关的肠道疾病,组胺在其中发挥着有益或有害的作用。
Smolinska S,et al.Metabolites.2022
消化道是接触大量不同分子的地方,这些分子中的一部分是潜在的过敏原,可能从而引起食物过敏。食物过敏可表现为轻度和重度症状,最严重的可能危及生命。
拓展:常见的食物过敏症状
食物过敏的症状表现在呼吸系统、消化系统、心血管系统和皮肤系统等。
皮肤反应:皮肤反应是最常见的食物过敏症状之一。它可以表现为皮疹、荨麻疹、瘙痒、红肿、水肿等。这些反应通常发生在口腔、面部、颈部和四肢等部位。
呼吸道症状:食物过敏还可以引起呼吸道症状,包括鼻塞、流涕、打喷嚏、喉咙痒、咳嗽、喉咙紧闭感和哮喘。严重的过敏反应可能导致呼吸困难和哮喘发作。
消化系统症状:食物过敏可以引起消化系统症状,包括腹痛、腹胀、恶心、呕吐和腹泻。这些症状还可能与肠道炎症反应、肠道收缩有关。
循环系统症状:在严重的食物过敏反应中,可能出现循环系统症状,如低血压、心悸、头晕和昏厥。这些症状是由于过敏反应导致血管扩张和血压下降。
全身症状:某些人可能出现全身性症状,如疲劳、焦虑、情绪波动和体重下降。这些症状可能与食物过敏引起的炎症反应和免疫系统的激活有关。
• 食物过敏时组胺分泌增加
IgE依赖性食物过敏是免疫系统紊乱的结果,使耐受性丧失。这导致将温和的食物抗原识别为病原体。组胺受体参与食物过敏的发展。研究表明,在人类中使用H2R拮抗剂会增加针对食物抗原的IgE的产生。在Th2细胞的刺激下,IgE与效应细胞(肥大细胞和嗜碱性粒细胞)上的Fcε受体结合。由于效应细胞的激活,组胺以及其他介质被释放。
在食物过敏受试者中,组胺分泌增加和肥大细胞数量增加已得到充分证明。嗜碱性粒细胞释放的组胺与皮肤点刺试验和食物激发呈正相关。与非过敏患者相比,食物过敏患者抗IgE介导的肥大细胞组胺释放增加。
此外,将食物过敏患者的活检与抗IgE(人类)抗体或过敏原一起孵育,会导致组胺释放增加9倍。
• 食物过敏的治疗
由于食物过敏的发生率不断增加,已经进行了大量研究以开发新的治疗和预防策略。严格避免食物过敏原是管理IgE介导的食物过敏的长期策略。
关于食物过敏发病机制的各个阶段也有许多研究,例如对Th2通路的影响、阻断IgE、抑制效应细胞和微生物治疗。长期免疫耐受应该是治疗食物过敏最理想的效果。
口服免疫疗法是食物过敏的发展治疗方法之一。它包括以每2-4周增加一次的剂量向患者施用过敏原,直到达到最大维持剂量。这个过程的结果是发展对食物的耐受性。该方法已用于对牛奶、鸡蛋、小麦、花生、坚果和贝类的食物过敏。
目前正在研究表皮和舌下免疫疗法。对牛奶和鸡蛋过敏的表皮免疫疗法也进行了临床试验。
炎症性肠病是特发性、慢性复发性肠道疾病。溃疡性结肠炎和克罗恩病是两种主要临床表现。
在克罗恩病中,炎症出现在弥漫性病变中,这些病变遍布消化道并深入穿透肠壁,可能影响所有层。相反,溃疡性结肠炎的炎性病变从直肠开始,向上但不超过结肠,并在黏膜浅表处保持。克罗恩病导致整个胃肠道的透壁性炎症,但其特征是模式不连续。与炎症为浅表的溃疡性结肠炎相比,溃疡主要局限于结肠粘膜。
• 炎症性肠病患者黏膜组胺水平升高
这两种疾病的发展都是遗传易感患者宿主免疫系统、肠道微生物群和环境因素之间复杂相互作用的结果。
炎症性肠病患者的黏膜组胺水平(而非血浆水平)升高。在患者尿液中发现与疾病活动度相关的N-甲基组胺水平升高。当受到抗原刺激时,源自活动性克罗恩病或溃疡性结肠炎切除结肠的肥大细胞能够释放比正常结肠更多的组胺。
• 炎症性肠病患者的组胺通路被破坏
克罗恩病和溃疡性结肠炎患者的组胺信号通路均被破坏,研究分析显示。炎症性肠病患者的组胺受体表达和功能活性发生改变。
在结肠炎模型中,H2R的阻断导致更严重的炎症性疾病。组胺主要抑制IFN-γ和TNF-α的分泌,这些细胞因子的基因表达与溃疡性结肠炎患者的H4R和H2R表达呈正相关。
发炎黏膜中组胺-N-甲基转移酶(HNMT)表达降低,二氨基氧化酶(DAO)多态性与炎症性肠病风险增加有关。使用H2R拮抗剂会增加克罗恩病患者的风险。
注:在小鼠结肠炎模型中,缺乏H2R或使用非莫替丁(H2R拮抗剂)抑制H2R的T细胞转移可加速体重减轻并增加疾病严重程度。
• 炎症性肠病的治疗
在许多情况下,获得缓解的唯一选择是根治性手术,切除发炎区域。潜在地,使用H1R和H4R拮抗剂同时使用H2R激动剂可能对炎症性肠病患者有益。
炎症性肠病患者还可接受抗炎药、类固醇、抗生素、氨基水杨酸盐治疗,或使用英夫利昔单抗(抗 TNF-α)进行生物疗法。
肠易激综合征是一种与腹部不适或疼痛有关的慢性疾病,其中吃的食物是更严重症状的触发因素。一些证据表明,肠易激综合征中肠道菌群发生了改变,这种改变会破坏黏膜的免疫应答。
• 内源性组胺与肠易激综合征的严重程度相关
研究显示,超过一半的患者因食用释放组胺的食物或富含生物胺的食物而出现胃肠道症状。内源性组胺的水平与肠易激综合征患者症状的严重程度绝对相关。活化的肥大细胞产生更多的组胺,这与肠易激综合征患者的腹痛相关。肠易激综合征患者的黏膜上清液的组胺含量高于健康受试者的上清液。
组胺水平更高,组胺受体表达也更高
肠易激综合征患者中组胺受体H1R和H2R的表达水平上调。我们推测:伴有组胺分泌或含组氨酸脱羧酶细菌的菌群失调可能与肠易激综合征的发生和加重有关。
给予特定微生物具有治疗作用,这也可能是微生物群变化导致疾病的原因。肠易激综合征没有特效治疗方法,减少炎症的药物正在使用中。
结直肠癌是第三常见癌症,也是癌症相关死亡的重要原因。与一般人群相比,炎症性肠病患者发生结直肠癌的终生风险会增加。
• 组胺可能具有潜在的抗肿瘤作用
肠道微生物群在结直肠癌中的作用已在谷禾之前的文章中详细描述过。而特定的肠道微生物代谢物可能与结直肠癌的病因有关。组氨酸脱羧酶缺乏已被证明通过CD11b+Gr-1+未成熟髓系细胞的积累促进炎症相关的结直肠癌,表明组胺具有潜在的抗肿瘤作用。
几种益生菌菌株,包括长双歧杆菌(Bifidobacterium longum)、嗜酸乳杆菌(Lactobacillus acidophilus NCFM)和鼠李糖乳杆菌GG,在结肠癌的小鼠模型中显示出有益作用。这些产生组胺的益生菌减少了结肠肿瘤的数量和大小。
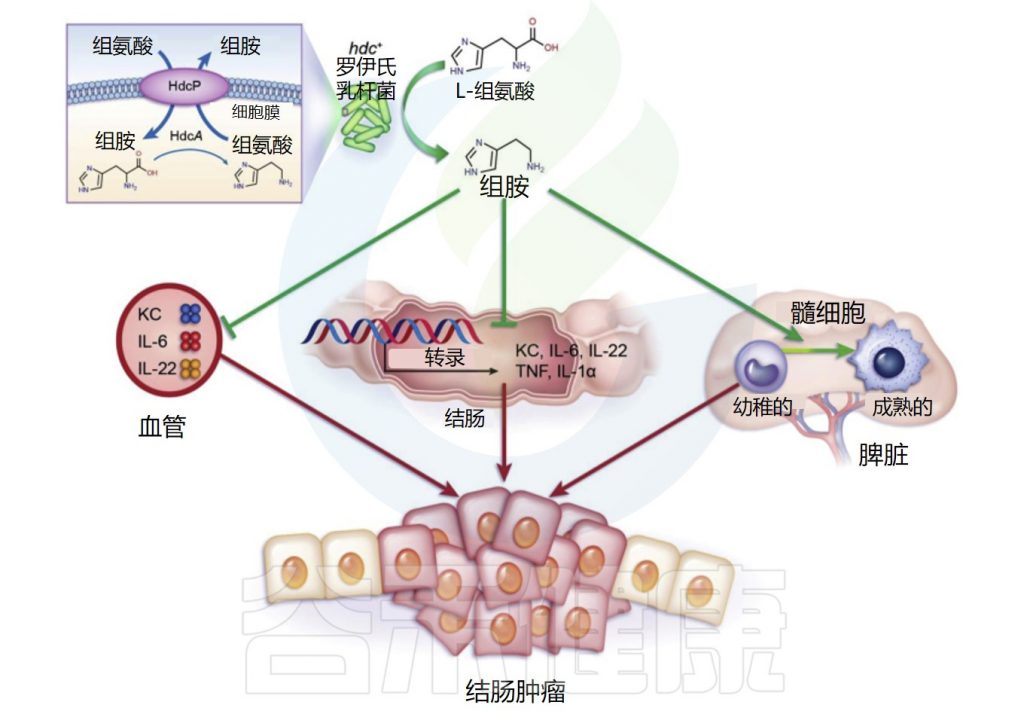
Gao C,et al.Am J Pathol.2017
罗伊氏乳杆菌(L. reuteri)抑制结肠粘膜中IL-22、IL-6、tnf-α和IL-1α 基因的表达,并减少血浆中促炎、癌症相关细胞因子、角质形成细胞趋化剂、IL-22和IL-6的数量。
此外,不能产生组胺的等基因组氨酸脱羧酶(HDC)缺陷罗伊氏乳杆菌突变体不能抑制致癌作用,这表明共代谢物组胺在抑制慢性肠道炎症和结直肠肿瘤发生中具有重要作用。
• 结直肠癌患者组胺含量和活性增加
在结直肠癌患者的结肠黏膜中,组氨酸脱羧酶活性和组胺含量均高于正常样本。在实验诱导的结直肠癌小鼠中,与野生型小鼠相比,组氨酸脱羧酶的缺失导致肿瘤发生增强,这也表明组胺具有抑制结直肠肿瘤发生的作用。
• 注意:过高的组胺含量可能对癌症不利!
需要注意的是,已发现肥大细胞在结肠癌中含量丰富,并促进小鼠化学诱导的结直肠癌的癌变,并且与人类结直肠癌患者的不良预后有关。与组胺通过H4R的促炎作用类似,H4R表达的缺失也导致小鼠化学诱导的致癌作用减少。
组胺作为一种重要信号分子,主要因其在诱导过敏症状中的作用而被识别,但它也参与非过敏性炎症反应。
组胺是肠道稳态的重要调节剂。通过增强分泌和蠕动,组胺促进潜在有毒物质的快速清除。这种生物胺对于肠道细菌的生存也至关重要。与健康个体相比,组胺不耐受患者的肠道菌群失衡或失调。
在某些浓度范围内,组胺起着保护作用,对于维持健康状态至关重要。然而,由于先天性或后天性缺乏组胺降解酶,以及肠道肥大细胞过度增生和过度活跃,使组胺浓度较高,会导致粘膜炎症性疾病,产生腹泻和腹痛。增强H2R表达或其细胞内信号,同时降低H1R和 H4R活性是提高粘膜免疫力的合理方法
组胺不耐受的现代治疗方法包括避免高组胺食物的摄入、影响组胺水平的药物(H1R阻滞剂、肥大细胞膜稳定剂和米氮平)和补充天然成分(维生素C、类黄酮、二氨基氧化酶强化补充剂和益生菌) 。
与此同时,使用具有释放组胺潜力的肠道微生物群提供了一种新的治疗视角。
主要参考文献
Smolinska S, Winiarska E, Globinska A, Jutel M. Histamine: A Mediator of Intestinal Disorders-A Review. Metabolites. 2022 Sep 23;12(10):895.
Sánchez-Pérez S, Comas-Basté O, Duelo A, Veciana-Nogués MT, Berlanga M, Latorre-Moratalla ML, Vidal-Carou MC. Intestinal Dysbiosis in Patients with Histamine Intolerance. Nutrients. 2022 Apr 23;14(9):1774.
Shulpekova YO, Nechaev VM, Popova IR, Deeva TA, Kopylov AT, Malsagova KA, Kaysheva AL, Ivashkin VT. Food Intolerance: The Role of Histamine. Nutrients. 2021 Sep 15;13(9):3207.
Krell T, Gavira JA, Velando F, Fernández M, Roca A, Monteagudo-Cascales E, Matilla MA. Histamine: A Bacterial Signal Molecule. Int J Mol Sci. 2021 Jun 12;22(12):6312.
Mou Z, Yang Y, Hall AB, Jiang X. The taxonomic distribution of histamine-secreting bacteria in the human gut microbiome. BMC Genomics. 2021 Sep 26;22(1):695.
Gao C, Ganesh BP, Shi Z, Shah RR, Fultz R, Major A, Venable S, Lugo M, Hoch K, Chen X, Haag A, Wang TC, Versalovic J. Gut Microbe-Mediated Suppression of Inflammation-Associated Colon Carcinogenesis by Luminal Histamine Production. Am J Pathol. 2017 Oct;187(10):2323-2336.
Kovacova-Hanuskova E, Buday T, Gavliakova S, Plevkova J. Histamine, histamine intoxication and intolerance. Allergol Immunopathol (Madr). 2015 Sep-Oct;43(5):498-506.
Barcik W, Wawrzyniak M, Akdis CA, O’Mahony L. Immune regulation by histamine and histamine-secreting bacteria. Curr Opin Immunol. 2017 Oct;48:108-113.
Smolinska S, Jutel M, Crameri R, O’Mahony L. Histamine and gut mucosal immune regulation. Allergy. 2014 Mar;69(3):273-81.

谷禾健康

营养对于身体健康和疾病预防的重要性已得到充分证实,全球膳食指南建议也得到了明确定义,以指导有效的政策。然而,吃什么、何时吃以及如何保持健康地优化饮食模式实际上非常复杂。
许多因素会影响饮食模式,包括个人的生理需求、疾病或健康状况、社会环境和文化因素,以及对饮食干预的生物、生理和社会心理反应,都需要考虑。
各种饮食可以带来积极的健康结果,但到底哪种饮食最适合个人以及这种饮食在整个生命周期中如何变化,是目前许多研究人员及临床医生迫切想要了解的。
如今“精准营养”快速发展,它强调使用个人化信息来制定针对某一特定群体的营养建议和饮食计划。其主要目标之一是提供量身定制的饮食建议以及预期的个性化反应。虽然越来越多的证据支持精准医学对于最佳个人健康和慢性病管理的需求,但“个性化”或“精准”营养干预要进行推广还需要一些路要走。
美国国立卫生研究院 (NIH) 国家心肺血液研究所(NHLBI)于2021年1月11日至12日召开了为期两天与会者讨论了四大慢性疾病领域的精准营养:1)心血管疾病,2)认知能力下降和阿尔茨海默氏病,3)2型糖尿病和血糖调节受损,以及4)与饮食相关的癌症。会议也着重阐述了对旨在影响健康和疾病的饮食调节的显著的个体差异。这突显了国际研究对于个性化营养在慢性疾病中的作用的重视。
饮食是慢性病风险的主要驱动因素,基于人群的指导应考虑个人反应。通过饮食减少慢性病需要更精确地:
(a)确定整个生命周期中健康和疾病的基本营养需求;
(b) 考虑营养素和其他食品物质对支持健康衰老的代谢、免疫、炎症和其他生理反应的影响;
(c) 考虑健康的饮食行为。
既然是精准营养,那么应该是人口亚群,而不是整个人口,可能以相似的方式对某种饮食或食物成分摄入或接触做出相似的反应,通过这种反应规律我们能给出比大人群层面更具体,更有针对性或有效解决某个反应指标的建议,这种建议是基于小且特定慢病人口亚群的遗传特征、代谢状态、生活方式、疾病状态或指标、括肠道微生态等。
本文主要讨论和阐述通过精准营养降低慢性病风险的可能性和挑战,一些可以作为慢性病精准营养的生物标志物以及减少慢性病的营养考虑因素。希望有助于精准营养这一领域更好地发展。
精准营养(个性化营养)是根据遗传特征、生活方式、健康状况、代谢特点、微生物组成等因素来定制针对特定群体的饮食建议和营养计划的一种方法。
★ 每个人的营养需求是独特的
研究认为,由于每个人的生物学特性和环境暴露都是独一无二的,因此营养需求也是个性化的。
个性化营养的目标是为每个人提供最适合其个体化需求的营养建议,从而促进健康、预防疾病、改善生活质量。
在实现长期生活方式改变方面,个性化营养建议比传统的方法更有效。这可能是由于精准营养更进一步,它假设我们可以提供已知对个人有益的个人饮食建议,基于对个体、表型和食物消费之间关系的定量理解。
★ 个性化营养需要考虑的点
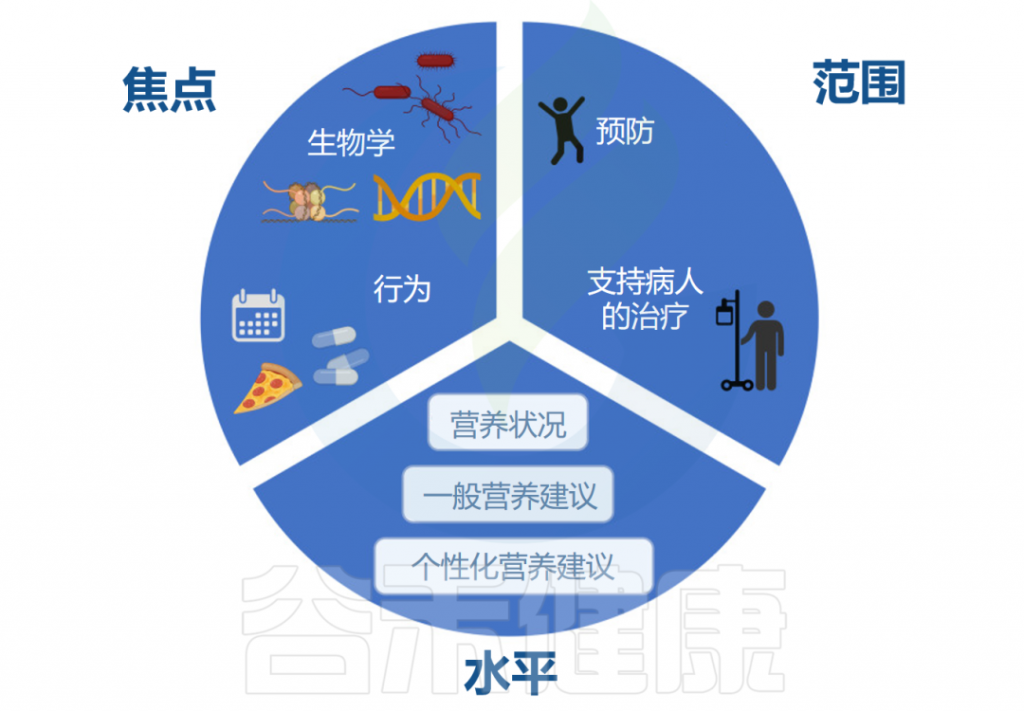
个性化饮食方案或个性化营养需要考虑三个方面:1)个性化的水平;2)个性化的重点;3)个性化的范围。
个性化营养的水平
个性化的水平有三层。在第一层或底层,我们找到了基于按年龄和性别划分的人群的一般准则的传统营养建议。这与营养科学传统上关注人群对某种饮食或营养素的平均反应相一致,可以被视为一级预防干预措施。
在第二个层面上,我们通过添加有关个人营养状况的表型信息(例如生化和人体测量数据)来添加个性化层。
在第三个层面,我们的目标是达到个性化(或精准)营养的水平,该水平考虑了多个方面,例如基因型、肠道微生物群或代谢组。与药物类似,营养素能够与生物体生理功能相互作用并调节分子机制,因此有可能帮助制定个性化的饮食建议。
个性化营养的重点
个性化营养的重点:生物学或行为。在精准医学中,我们的目标是根据遗传、表观遗传和肠道微生物等特征等了解对饮食和营养素的差异反应。这种程度的生物学理解也可以指导营养建议。
例如,更好地了解特定营养素和非营养成分在遇到特定肠道细菌菌株时如何以及哪些特定营养素和非营养成分可能诱发肠道炎症反应,可能是炎症性肠病个人建议的关键。然而,改变饮食模式应包括对当前行为、偏好、障碍和目标进行彻底评估。
个性化营养的范围
个性化营养的最后一个方面是它的范围。除了不同程度的个性化之外,其范围可以是解决公共卫生保健和疾病预防方面的新问题,也可以旨在支持患者的治疗。
Wellens J,et al.Pharmgenomics Pers Med.2023
纵观人类历史,食物塑造了人体基因组,以便在多种环境背景下生存和人口扩张。在许多人的食物生态系统不断变化且越来越不适应的背景下,导致了现代慢性病发病风险的变化。
✦ 食物供应和成分会影响人类遗传和表型
食物供应和食物成分是导致现代人类遗传和表型变异的主要环境选择压力之一。基因组通过遗传选择和随机漂移等过程进化,这些过程可以改变饮食与疾病的关系。
并非基因组中的所有基因都以相同的速度进化。在人类和其他物种中高度保守的基因通常编码具有维持生命的基本功能且基本上不受外部环境影响的蛋白质。
相比之下,快速进化的基因在不同人群中表现出DNA一级序列的变异,从而改变生理功能并导致人类遗传和表型变异。这种适应性基因历来允许在特定的环境中生存。
✦ 对食物环境变化的适应不良产生疾病基因
因此,涉及食物、营养、新陈代谢以及免疫功能的基因表现出一些最高的基因进化率并因此出现遗传和表型变异也就不足为奇了,因为随着时间的推移生存和扩张的人类群体必须适应当地独特的食物和致病环境。这些适应使人们能够在所处区域环境下生存,但当环境发生变化时,包括从狩猎采集社会向农业社会转变导致的食物环境变化,可能会出现适应不良,从而产生疾病等位基因。
饥荒是人类历史上的常见现象和选择压力,它以维持物种所需最低剂量的必需营养素优化了生物功能。通过细胞与营养环境的相互作用,在人类和其他哺乳动物中观察到这种选择压力的影响,其中必需营养素与酶和转运蛋白(Km、Kt)的结合亲和力在人类中(通常在哺乳动物中)高度相似并以维持生理功能的要求变化最小的方式保存下来。
✦ 食物过剩导致了慢性病发病率上升
因此,在维持基本营养充足性的基础上推导膳食参考摄入量(DRI)时,对准确性的需求更为迫切,因为建立群体亚群的需求仅限于整个生命周期生理需求的差异,而不是健康人群中独立于生命周期影响(如遗传学)的生理变化。
也有一些例外,例如常见的亚甲基四氢叶酸还原酶突变体(MTHFR C677T)对辅因子结合的影响,导致维持充足性所需的叶酸更高。
然而,我们生活的环境已经从饥荒转变为食物过剩,从解决营养缺乏症转变为解决与饮食相关的慢性病发病率上升的问题,所有这些都是在食品供应全球化的背景下进行的。
我们越来越认识到饮食与疾病关系中存在有意义的异质性,因此有必要建立饮食建议的新方法,包括识别和分类亚组的新方法(即提高精度)。
预防营养缺乏和随后的营养缺乏相关疾病以及维持生理功能是营养素和其他食物物质(即能量、纤维、宏量和微量营养素)摄入量建议的目标。
✦ 膳食参考摄入量框架的提出
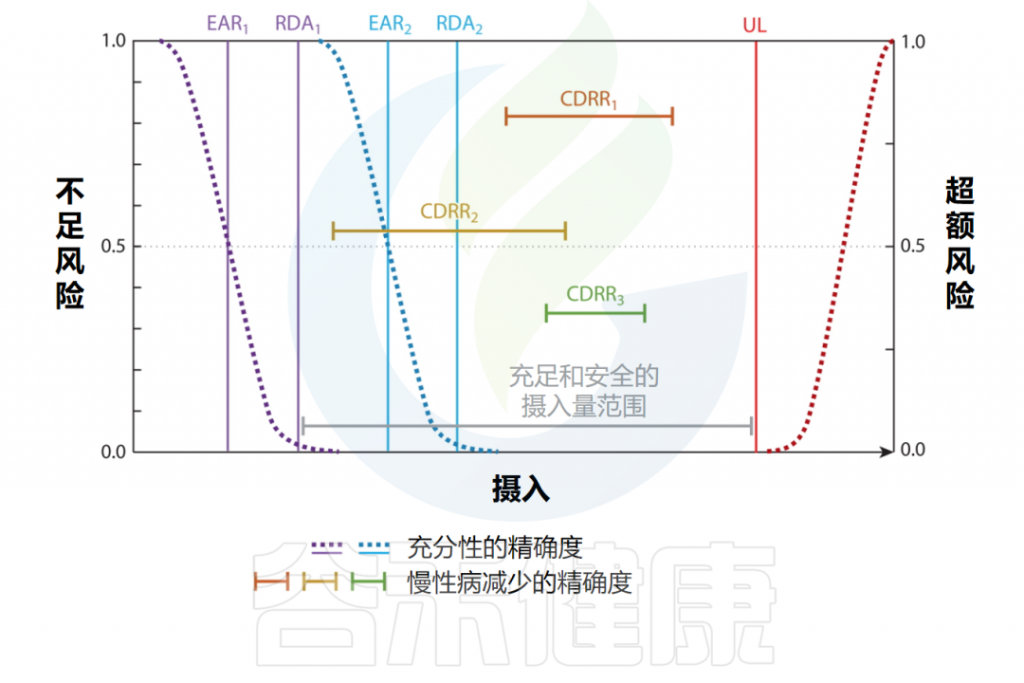
20世纪90年代末,制定了一套涵盖营养不足和营养过剩风险的建议,广泛称为膳食参考摄入量(DRI)表,如下所示:

Bailey RL,et al.Annu Rev Nutr.2023
✦ 人群中必需营养素的需求存在差异
DRI框架首先引入了精确指导的概念,承认有限数量的人群亚组中必需营养素的剂量反应关系存在差异。
鉴于与减少慢性疾病相比,预防营养缺乏和毒性状态所需的精确度较低,因此在建立基于保持特定亚组充足性的DRI时,只需要考虑有限数量的人口亚组,这符合人类自然历史。这是因为健康人群中的大多数个体往往对必需营养素暴露有类似的反应,并且因为营养素暴露是缺乏性疾病的单一根本原因,并且在健康人群中的相似时间过程中在整个人群中表现出特定症状。
例如,仅缺乏维生素C的饮食会导致坏血病的早期非特异性症状,例如大约4周内的疲劳,并在8至12周之间开始出现更具体的严重症状,包括包括瘀点和螺旋状毛发。
过高剂量可能导致毒性反应
同样,高剂量的毒性反应通常会导致人群出现相似的特征;例如超生理性摄入锌会导致胃肠道症状和疲劳,进而可能引发铜缺乏症。
采用总体、精确和个性化方法的膳食参考摄入量
Bailey RL,et al.Annu Rev Nutr.2023
✦ 整体饮食模式的分析
虽然DRI框架特别关注饮食中的其他食品物质,但美国人饮食指南(DGA)自1980年以来就提供了基于食品和饮料的建议,早期强调特定食物组,最近已转向整体饮食模式。
DGA旨在提供一套基于证据的饮食建议,以“帮助促进健康和预防慢性病”。基于食物的方法,特别是饮食模式研究,扩大了DGA的范围,并在此过程中代表了“饮食中不同食物、饮料和营养素的数量、比例、种类或组合,以及其频率”。它们被习惯性地消费”。
饮食模式可以通过多种方式得出;所有方法都可以分为独立的或依赖于特定健康结果的方法。结果依赖方法将感兴趣的结果或中间生物标志物纳入用于推导模式的模型中;示例包括降序回归和分类以及回归树分析。
尽管这些方法可用于检查饮食与特定结果之间的关系,但大多数营养研究人员使用与感兴趣的结果无关的方法来描述一般饮食质量。
开发与结果无关的饮食模式的方法有两大类:数据驱动的技术,例如因子分析或聚类分析,强调数据缩减技术或根据报告的饮食摄入量对个体进行聚类;以及基于索引的方法,它们是基于饮食指南或建议的先验模式。通过数据驱动的方法对饮食模式进行分类可能会导致研究人员在推导饮食模式时做出多种主观决定,从而使不同群体或人群中模式的比较复杂化,并降低其在定义基于食物的模式的研究中的效用。
然而,因子分析和聚类分析都是有用的数据缩减技术,可以确定复杂数据集中的基础结构,就像饮食暴露的情况一样。指数和分数的使用本质上创建了一张报告卡,说明饮食在多大程度上符合预定义的准则。虽然评分标准的制定方式存在主观性,但该方法提供了一个标准化框架来比较不同研究。
整体饮食模式和疾病风险之间存在关联
为此,2015年美国人膳食指南咨询委员会在系统审查的基础上,得出的结论是,指数和分数是捕捉饮食模式和整个饮食复杂性的首选方法。
存在各种指标和分数例如健康饮食指数、地中海饮食评分,以及控制高血压的饮食方法(DASH)得分。
使用该框架,2020年得出结论,存在强有力的证据表明较高质量的饮食模式与较低的全因死亡率风险相关和心血管疾病并且存在与2型糖尿病相关的饮食模式的适度证据、骨骼健康、超重和肥胖,以及成人结直肠癌和乳腺癌。
✦ 特殊的生命阶段具有独特的营养需求
最近采用生命阶段方法来评估现有的科学证据,包括所有生命阶段,并特别强调怀孕和哺乳期间以及婴儿和幼儿(出生至24个月)期间的独特营养需求。
除了建议遵循的食物模式外,还首次确定了与公共健康相关的特定生命阶段的其他食品物质以及所有生命阶段的公共健康问题,包括饮食暴露、生物终点或疾病患病率或经过验证的疾病替代标志物。
因此,虽然一些膳食指南是通用的,但人们认识到需要根据生命阶段向人群扩大具体建议——这代表了第一个超越DRI的、基于生命阶段针对特定群体的公共卫生营养方法。
DGA还认识到,存在多种潜在的饮食模式可实现类似的健康结果,例如地中海饮食或素食模式可预防心血管疾病。DGA审查并纳入的饮食模式并不代表膳食补充剂的使用,大大低估了使用膳食补充剂的一半成人和三分之一儿童的营养暴露量。
因此,开发了总营养指数,除了食品和饮料中的营养暴露之外,还包括补充剂中的营养暴露,与基于食品和饮料的指数结合使用,以提高暴露分类的全面性。
心血管疾病、糖尿病、癌症和关节炎等慢性疾病是全球范围内发病和死亡的重要原因,其患病率在所有年龄组、性别和种族中都在稳步上升。
★ 精准营养在预防和管理慢性病中起重要作用
越来越多的证据表明,精准营养在慢性病的预防和管理中发挥着至关重要的作用,被认为是未来十年健康研究的重点领域。
尽管如此,精准营养的主要挑战之一在于准确可靠地评估食物和营养素,特别是复杂食物和大分子。此外,需要确定这些食物和营养素如何影响个人的健康和疾病状况。
令人鼓舞的是,强有力的证据支持使用生物标志物作为中介工具,有效地建立精准营养和慢性疾病之间的联系。这种联系有助于客观评估食物消耗,并精确确定复杂食物和成分的生物效应。
尽管取得了这些进步,但我们目前对精准营养如何调节生物标志物以预防具有个体差异的慢性疾病的理解仍处于起步阶段。关键生物标志物参与慢性疾病的分子机制仍未得到充分阐明,需要进行全面和广泛的研究工作来弥补这一知识差距。
✦ 多种慢性疾病具有潜在的生物标志物
研究已确定了多种慢性疾病的新型潜在生物标志物。其中,单一物质作为生物标志物,如用于冠心病的25-羟基维生素D、用于非酒精性脂肪肝的视黄醇、2型糖尿病的荧光晚期糖基化终产物和用于烟雾病的支链氨基酸。
不同物质的比例也可作为生物标志物
此外,不同物质的比例已被提议作为生物标志物。发现肌酸与半胱氨酸蛋白酶抑制剂C(Cr/CysC)比值和白蛋白与球蛋白比值可以分别作为慢性肾病和泌尿系癌症预后的非侵入性生物标志物。
值得注意的是,研究发现Cr/CysC比率也可能是骨质疏松症的潜在生物标志物。此外,确定老年营养风险指数是老年高血压患者中风的潜在标志物。
研究还发现磷与白蛋白结合的水平可以作为全因死亡率和心血管死亡率的潜在标志。而与脱铁性贫血相关的差异表达基因可以作为新的生物标志物用于识别缺血性中风和指导治疗干预。
这些发现的生物标志物为相关慢性病的预防和管理提供了重要的理论基础,也有助于了解各种慢性病的发病机制。
✦ 一些膳食因子可以改善慢性疾病
研究表明,膳食中补充花青素、谷氨酰胺、维生素K和脂肪酸等功能因子,可以分别缓解心力衰竭、高盐高血压、血管钙化和骨矿物质损失。
控制功能性膳食因子的摄入量非常重要
然而,重要的是要谨慎控制饮食功能因子的剂量,例如过量摄入脂肪酸会导致代谢性疾病。
一些食物对各种慢性疾病存在积极影响。例如,膳食水果摄入可改善功能性便秘;以植物为基础的饮食可以用来控制代谢综合征;特定的口服营养补充剂(ONS)来解决炎症/氧化问题;藏红花缓解心血管疾病。
值得注意的是,还观察到益生菌补充剂与特定口服营养补充剂(ONS)对炎症/氧化的协同抵抗作用。
此外,联合训练,包括阻力训练和高强度间歇训练或中等强度连续训练,对非酒精性脂肪肝的治疗是有益的。除了这些预防或治疗慢性病的饮食策略外,饮食模式也会导致一些慢性疾病的发生,例如高盐饮食会加剧肠道衰老过程。
这些膳食功能因素和复合食品为慢性病的治疗提供了有效的预防措施。然而,负责调节这些生物标志物的具体机制尚未得到彻底探索,突显了该领域进一步研究的必要性。
微生物群多样性与改善脂质谱、抗炎细胞因子、肝酶以及最终的遗传途径有关,所有这些都是改善健康的代谢指标。
✦ 肠道微生物群影响宿主的新陈代谢
异常的肠道菌群和每日进食/禁食周期都会影响宿主的新陈代谢,并可能导致肥胖等代谢疾病的出现。研究发现,饮食和禁食周期会导致肠道微生物组发生周期性变化,从而充当控制宿主代谢的机制。
这些差异增加了肠道微生物群的多样性。因此,在确定微生物组对宿主生理和代谢的贡献时,进食模式、时间以及膳食的成分是需要考虑的重要特征。
肠脑之间的联系对于确定膳食量非常重要
肠道和大脑之间的沟通对于确定膳食的适当份量并向大脑发送信号以控制饥饿感和饱腹感至关重要。机械敏感性胃迷走神经传入(GVAs)在营养成分和化学途径方面表现出对食物相关刺激的昼夜节律性。这使得饱腹感信号可以通过肠脑交流在一天中的特定时间发生。
因此,GVA轴缺乏昼夜节律可能会导致食欲亢进和肥胖的增加。最近对动物和人类的研究表明,肥胖的出现与微生物群多样性降低、肠道微生物群活性改变和微生物群丰度分散相关,特别是拟杆菌门和厚壁菌门这两个门。当拟杆菌被允许留在肠道中时,它们会继续与宿主保持动态且在很大程度上是有益的关系。
✦ 肠道微生物群失调与慢性疾病相关
肠道微生物群失调是多种人类疾病出现的介质。研究表明,肥胖、动脉粥样硬化、神经系统疾病和糖尿病等代谢性疾病和炎症性疾病的患病率与拟杆菌的数量呈反比关系。
同样,拟杆菌被发现与低密度脂蛋白(LDL)和甘油三酯(TG)水平呈负相关,并且这两种细菌都表现出抗肥胖反应。在类似的模式中,小鼠体重的下降与细菌属成员的增加密切相关。
✦ 肠道微生物对人体产生的有益影响
肠道微生物群是影响时间生物学、代谢传感器、炎症细胞因子、神经功能和免疫系统的”物质工厂”。
肠道微生物消耗膳食中的营养物质来产生能量和代谢物。许多这些代谢物随后进入循环,在那里它们可能会经历额外的代谢并改变宿主的代谢和营养状况。
肠道微生物群对人体具有有益的作用。主要好处是它在维持粘膜屏障完整性方面的作用;由难以消化的纤维产生的短链脂肪酸对宿主有利的作用,包括对抗肥胖和糖尿病的活动;此外肠道微生物可以合成必需维生素,包括维生素K、烟酸、核黄素、吡哆醇、泛酸和硫胺素,以防止感染病原体;增强免疫系统;调节昼夜节律;以及身体的神经功能。
✦ 肠道菌群产生的代谢物影响宿主免疫、代谢
肠道微生物产生的许多代谢物进入血液,可以立即发挥作用,也可以通过宿主进行额外的代谢,产生可能影响宿主代谢和组织功能的生物活性化学物质。
除了促进脂肪吸收外,次级胆汁酸还会被重新吸收到血液中,作为宿主细胞法尼醇X受体(FXR)和TGR5胆汁酸受体的配体,对免疫功能和能量代谢等产生影响。
同样,由细菌产生的短链脂肪酸,如乙酸盐、丁酸盐和丙酸盐,不仅是肝脏和肠上皮的重要能量来源,而且还能够改变胰岛素分泌、免疫系统活动、食欲、大脑功能和脂肪脂肪。
这些短链脂肪酸影响身体的免疫、激素和神经系统,因为它们参与细胞因子、神经递质、内分泌信号和细胞凋亡过程的产生。
✦ 一些细菌代谢物对宿主是有害的
同时需要注意的是,一些细菌代谢物对宿主代谢的影响有时可能是有害的。
细菌从膳食蛋白质中产生的N-亚硝基化合物、氨和硫化氢会产生活性氧(ROS)并在DNA损伤中沉淀。
这些化合物还可以激活促炎途径。膳食胆碱的最终产物三甲胺-N-氧化物(TMAO)会促进动脉粥样硬化的发展,并与心血管疾病(CVD)、中风和死亡率相关。
因此,肠道微生物群落的这些改变有可能在2型糖尿病、体重增加、心血管疾病和代谢综合征等慢性疾病的出现中发挥作用。
✦ 许多人患有慢性病
如今,大多数人患有一种或多种慢性疾病以及药物使用等相关因素可能会改变营养需求,导致营养缺乏和继发性合并症。
超过一半的人存在超重或肥胖,并且过去二十年来严重肥胖的患病率有所增加。超重和肥胖率高是一个重要的公共卫生问题,其还会增加患心脏代谢疾病和某些类型癌症的风险。
✦ 疾病会影响营养吸收、分配造成需求差异
众所周知,疾病过程会影响组织之间的营养吸收、分解代谢和营养分配,可能导致维持某些关键营养充足的需求差异。
术语“特殊营养需求”是指在疾病状态下维持充足性所需的营养需求。慢性病、遗传性疾病,包括先天性代谢错误、炎症、饮食不耐受、药物、过敏、创伤和感染等病理状态,可以改变基本营养需求的缺乏和毒性。
目前,在医学营养治疗中考虑了其他食品物质的这些差异性要求,这是一种更个性化的指导,目前超出了DRI的范围。
开发旨在减少慢性病的DRI的生物学基础并不支持将基于营养和食物的建议限制于表面健康的个体,因为对于疾病何时开始没有明确的诊断标准。慢性疾病在整个生命周期中发生和显现,与衰老以及许多静态和动态因素以及环境暴露(包括食物)密切相关。
生物系统的逐渐衰退是衰老和慢性疾病进展的标志,这些疾病从生命的最初阶段就开始了。生物网络和系统衰退导致功能侵蚀或随机行为增加,导致网络输出和系统行为的可变性/稳定性增加,从而与健康不相容。
例如,在分子水平上,这可以通过人类基因组表观遗传景观的侵蚀导致基因表达模式和网络功能的改变来量化。和血浆代谢物与年龄相关的变化,其中一些是营养状况的生物标志物,以及氧化还原电位的变化。
✦ 生活方式影响慢性疾病的发生和进展
许多生活方式、环境和内在生理危险因素都会影响生物衰老的速度以及慢性疾病的发生和进展,包括某些癌症、2型糖尿病、心脏代谢和神经退行性疾病等。
家族史(即遗传学)是寿命的主要不可改变的预测因素。慢性疾病是在人类发育的最早阶段通过基因组突变和干细胞表观遗传编程等机制引发的。在这方面,营养需求可以被视为一种动态对策,以减缓或维持与年龄相关的促进健康所需的生物系统和网络功能能力的下降,而这正是精准营养努力可能最有益的地方。
基于人群的减少生物衰老的方法需要将营养暴露、状态和功能的生物标志物与疾病和衰老的生物标志物更好地联系起来。
2017年,开发了一个框架,正式将慢性病风险降低价值观纳入DRI。这一变化强调了饮食指导的转变,除了避免不足之外,还转向促进健康和降低慢性病风险。
这种通过饮食降低慢性病风险的范式转变及其基本原理已在其他地方发表。与缺乏症不同,大多数慢性疾病会随着时间的推移而显现出来,并且是由衰老过程以及行为和生活方式因素的累积影响造成的。
✦ 减少慢性病需要考虑多个因素的相互作用
如上所述,建立减少慢性病的食物和营养摄入建议需要考虑多个独立和相互作用的因素。这些额外的生物因素增加了饮食与疾病关系中的人群异质性,进一步推动了对饮食建议更加精确的需求。
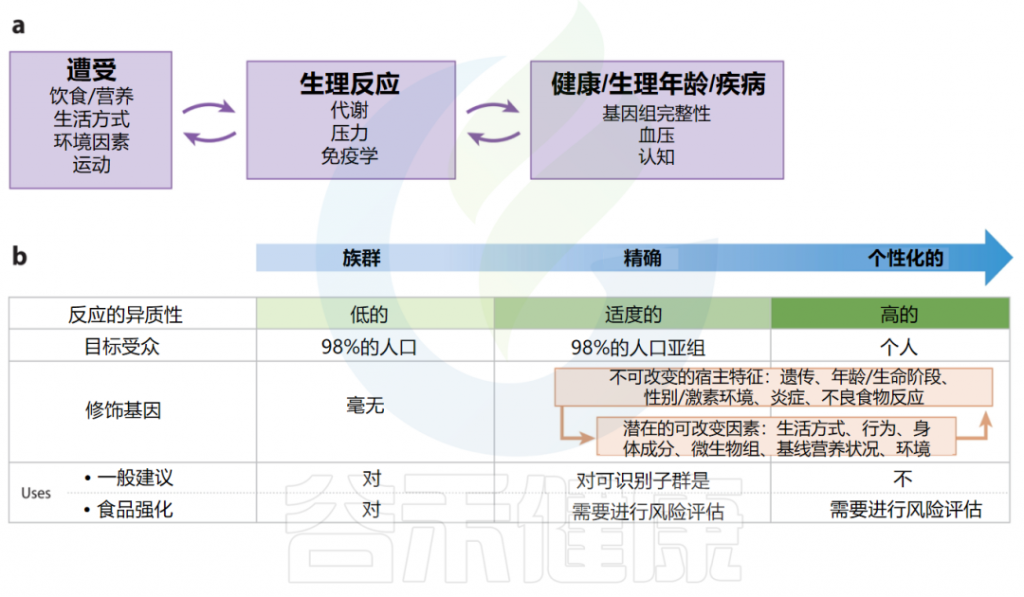
实施精确营养需要知识和工具(如生物标志物),这些知识和工具可以量化暴露(如饮食/营养、生活方式、环境因素、锻炼)与对健康和疾病(如基因组完整性、血压、认知)的生理反应(如代谢、压力、免疫)并将其联系起来。
值得注意的是,暴露和生理反应之间的联系是通过反馈循环相互影响的(例如,饮食可以影响炎症,进而影响饮食需求),并且生理反应与健康、疾病和衰老是相互影响的。
饮食与慢性病关系的原因和调节因素包括但远远超出必需营养素在维持代谢和其他功能中所发挥的作用。其他食品物质可以通过:
(a)必需营养素缺乏和过量的继发致病作用来影响慢性疾病的发作和进展;
(b) 必需营养素摄入不平衡的致病作用;
(c) 氧化应激、免疫反应和接触特定食物成分的其他反应的致病作用;
(d) 在没有必要性或毒性的情况下摄入影响慢性疾病的非必需生物活性食物成分;
(e) 饮食行为,包括时间饮食模式,也称为时间营养。
由必需营养素缺乏和毒性引起的疾病已得到临床认可和充分表征并且历史上在建立DRI(膳食参考摄入量)的过程中一直被考虑,重点是维持营养充足和生理功能。
当DRI(膳食参考摄入量)扩展到包括降低慢性病风险时,必须考虑营养物质在其已知功能作用之外的其他生理反应。饮食与慢性病的关系超越了生理功能,延伸到对饮食成分的免疫和应激反应,这些反应可以降低或增加疾病风险。
✦ 必需营养素的缺乏会引起炎症
必需营养素的缺乏和过量都会引起炎症,某些营养素的升高会改变生理过程,从而增加或降低慢性疾病的风险但不引起毒性。
例如,在动物模型中,镁缺乏会刺激氧化应激和吞噬细胞分泌促炎介质,导致慢性炎症。在人群中,膳食镁摄入量与心脏代谢疾病、代谢综合征、结直肠癌以及血清或血浆C反应蛋白(CRP)呈负相关。注:CRP是炎症的生物标志物,是许多慢性疾病的危险因素。趋化因子和CRP等炎症介质可作为生物标志物,报告衰老、运动、营养和慢性疾病,包括动脉粥样硬化、糖尿病、肥胖、肌肉减少症和阿尔茨海默病。
同样,亚临床维生素C不足与炎症、血浆C反应蛋白水平升高和免疫功能下降有关。
✦ 适当补充营养素水平可以降低炎症水平
另一方面,在不缺乏营养的情况下,大量摄入必需营养素可能会降低患有慢性疾病的人的炎症。补充维生素D可能会降低超重和肥胖儿童的血液CRP水平并改善小儿肠道综合征的炎症标志物。
叶酸和维生素B12缺乏已被证明会加剧与疾病和感染相关的炎症,而叶酸补充剂已被证明可以降低CRP血液水平。
总的来说,目前对必需营养素缺乏或过量在氧化应激和炎症中的作用的理解还是不够。需要进一步发展营养与慢性疾病关系的基础知识以及开展更多人体临床试验来验证特定营养素与慢病干预之间的关系。近年来,许多医院开设了临床营养科推进转化营养干预在疾病治疗和康复进程。
慢性疾病发生和进展的多因素病因学的特点是内在生物系统和外在环境因素(包括必需营养素)之间的相互作用,这些因素影响对维持健康至关重要的生理功能。
✦ 营养状况的不平衡会加速或加剧慢性疾病
事实上,所有代谢、信号传导和其他生理网络都涉及多种必需营养素之间的相互作用,同一系统中营养素之间营养状况的不平衡与加速或加剧慢性疾病有关。
钠/钾的平衡与血压以及心血管等疾病的风险相关
钠、钾和氯作为电解质发挥着重要作用,可调节细胞内的液体平衡,并在维持血压方面发挥关键作用。尿液中钾/钠比例的失衡反映了饮食暴露,并与成人高血压和心血管疾病的风险增加以及早产儿的发病率有关。
虽然一些人的饮食钠摄入量与血压之间没有关联,但一些人群亚组被认为对盐敏感,并且根据年龄、性别和血统,以及肾功能受损、肥胖和现有高血压的人群,更有可能对较高的钠对血压产生负向反应。
叶酸与维生素B12不平衡会致病
钠与钾的膳食比例对于高血压的影响至关重要。其他矿物质以及与饮食模式的相互作用也与健康相关。
B族维生素叶酸和维生素B12的状态不平衡及其相互作用可能会致病。叶酸介导的一碳代谢是合成核苷酸前体和将同型半胱氨酸重新甲基化为蛋氨酸所必需的代谢网络,支持100多种细胞甲基化反应。
该网络需要许多必需的微量营养素,包括维生素B12、维生素B6、叶酸、烟酸和核黄素。维生素B12缺乏情况下叶酸状态升高与单独维生素B12缺乏的神经、代谢和临床表现的恶化有关。这些潜在的有害相互作用引起了人们对叶酸摄入过量的担忧。
这些例子强调,需要考虑并推荐人群中的营养状况范围,通过优化给定生物网络中的营养素相互作用来促进健康,从而预防慢性病,为我们提供更精确指导的能力。
食物不耐受和食物过敏是对食物的免疫不良反应。它们是常见的慢性炎症性疾病,其患病率正在增加,影响生活质量,并且与其他慢性病的较高风险相关。
据估计,高达20%的人表现出胃肠道食物不耐受。
✦ 食物不耐受会增加胃肠道疾病风险
食物不耐受的原因有很多,包括:(a)饮食成分的药理学作用,例如短链可发酵碳水化合物,也称为可发酵寡糖、二糖、单糖和多元醇 (FODMAP);(b) 非免疫性麸质敏感性;(c)酶和运输缺陷。它们通常通过排除饮食来管理。
食物不良反应最常见的临床表现是肠易激综合症,它会增加胃肠道癌症的风险,但食物不良反应也会对皮肤、呼吸、神经和心血管系统产生负面影响。并增加患乳腺癌的风险。
✦ 不同的食物抗原会引起独特的免疫反应
食物过敏与其他形式的食物不耐受不同,有时相同的饮食成分可能引发多种不耐受机制。当免疫球蛋白E(IgE)介导的针对特定食物成分中存在的抗原表位的免疫反应时,就会发生食物过敏。
其他食物抗原也可以介导免疫和炎症反应。数据揭示了IgG抗体对患者食物中存在的表位的反应性增加,最常见的反应性食物是酪蛋白、牛奶、小麦、麦醇溶蛋白、蛋清和大米;不太常见的是对坚果、蔬菜、鱼类、海鲜和肉制品的反应。
对麸质中的麦醇溶蛋白敏感导致乳糜泻
例如,乳糜泻是一种与遗传相关的自身免疫性肠病,它使个体对某些谷物中存在的麸质中的麦醇溶蛋白和麦谷蛋白敏感,从而导致炎症反应。它出现在大约1%的人口中。
另一方面,麸质不耐受更为常见,影响高达6%的人口,总体而言,非乳糜泻小麦敏感可能影响 10% 的人。麸质不耐受与遗传无关,也不会引发过敏反应,但由于麸质成分激活先天免疫系统和多种炎症途径,它可能会出现与乳糜泻类似的症状。
✦ 食物成分引起的不良反应还会对肠道微生物产生负面影响
对食物和食物成分还有多种其他不良反应,它们独立于免疫参与,并通过许多不同的已知和未知机制表现出来,它们被分为依赖宿主或独立于宿主。
最常见的临床表现包括荨麻疹或血管性水肿,但也包括哮喘、胃肠道症状、低血压、头痛和湿疹。非免疫性、不依赖宿主的食物不耐受涉及食物中具有药理活性的化学物质,这些化学物质会影响致敏个体,包括水杨酸盐、血管活性胺(如组胺)、谷氨酸盐和咖啡因,但其病因和管理仍然难以捉摸。
非免疫性、宿主依赖性食物不耐受通常包括缺乏宿主代谢能力,如乳糖和果糖不耐受以及对某些食物(包括FODMAP)的非特异性反应。这些化合物在胃肠道中引起渗透作用,促进结肠细菌的不良发酵,并可对微生物群的组成产生负面影响,引发炎症,并诱发肠易激综合征症状。
✦ 摄入生物活性膳食成分可以降低慢性病风险
摄入非必需生物活性膳食成分(也称外源性成分)有可能降低慢性病风险,因此可以在建立DRI的过程中进行评估。
类胡萝卜素与眼睛健康有关
其中包括非维生素原A类胡萝卜素、叶黄素和玉米黄质素等生物活性物质,它们与眼睛健康和眼睛发育有关。
类黄酮与多酚具有抗氧化活性
类黄酮和其他多酚具有抗氧化和转录激活活性,并与保护多种慢性病有关,以及潜在的半必需营养素,例如omega-3脂肪酸。
ω-3的摄入与多种疾病存在关联
omega-3脂肪酸的摄入量与心血管疾病风险、认知功能、抑郁症风险和早产等结果有关。然而,支持这些关联的数据在文献中并不一致,这可能说明当食用不同的摄入量范围时,可能会改变健康结果。
✦ 生物活性膳食成分对于不同人群的健康益处存在差异
由于这些化合物可以改善健康,但从技术上讲并不是生命所必需的,因此与必需营养素相比,其历史区域丰度的变化和可能起作用的不同选择压力,预计其生物和健康影响会出现更大的群体异质性。
此外,生物活性膳食成分以及许多合成药物的细胞浓度受其分解代谢的调节。这些底物被细胞色素P450酶降解或生物激活,细胞色素P450酶在人群内部和人群之间表现出底物特异性和催化活性的广泛差异,导致对各种底物的功能反应存在异质性。
人们越来越认识到一天内进食的时间和频率会影响健康结果。
进食时间和某些其他食品物质(包括咖啡因和多酚)的直接生物效应可以改变生物钟。其他食品物质与生理昼夜节律、禁食(包括每日进食频率)和其他饮食行为的相互作用都会影响代谢过程并促成饮食与疾病的关系。
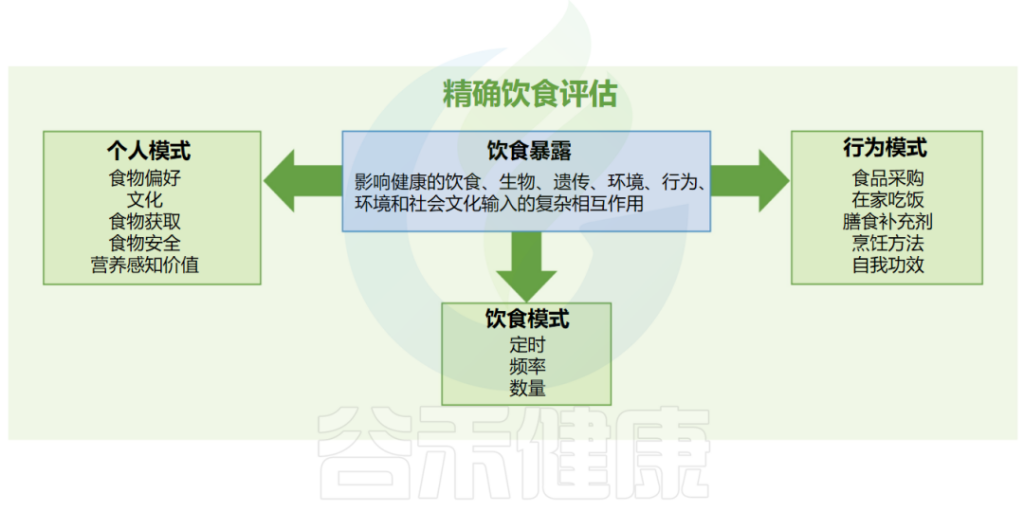
✦ 了解个人饮食及行为模式利于精确饮食评估
此外,饮食行为在生理反应中表现出个体间的差异,目前不被认为在DRI或DGA过程的范围内,但它们是饮食暴露组中的一个重要维度。
不仅要了解人们吃什么,还要了解饮食行为的这些背景因素,对于了解如何制定精准的营养建议至关重要。需要对精确饮食评估进行研究以捕获这些和其他背景因素,以提高我们提出更精确饮食建议的能力。
Bailey RL,et al.Annu Rev Nutr.2023
Bailey RL,et al.Annu Rev Nutr.2023
个体对特定饮食的反应是代谢、环境、社会和遗传等因素相互作用的结果,这表明不同的个体对相同的干预措施会有不同的反应。
例如,在最近一项针对600多人的随机对照试验中,12个月的低脂饮食导致一些人体重减轻超过30公斤,但另一些人体重增加超过10公斤,这表明单一饮食没有对所有人都有效,而精准营养可能会更有效。
饮食的良好保留和差异化,这些因素可能可以用微生物组等未测量的元素来解释。
在研究中,来自7个欧洲国家的1607名成年人被招募参加随机对照试验,该试验提供常规饮食建议(对照)或基于个人基线饮食的个性化建议;个体基线饮食加上表型(人体测量和血液生物标志物)或个体基线饮食加上表型和基因型(五种饮食反应性遗传变异)。这项研究结果表明个性化饮食优于常规饮食。
在这种情况下,为患者设计精准饮食计划可能会:
1)在治疗疾病方面更有效;
2)提高依从性,因为个性化饮食更容易被患者接受;
3)限制较少。
这些循证医学的研究证据也正是我们以及相关公司开展业务的基础——搜集、整理文献,构建数据库获得解决方案。然后,从客户的行为数据反馈中学习、进一步反哺研究。
除此之外,很多慢病和饮食模式之间的因果关系都逐渐清晰,或者反过来说,现代科学家和临床医生、营养学家(通常是几个角色兼具者)已经开发出了很多应对慢病的饮食模式(“疗效”多数远大于药物),其中最大一块就是“胃肠道慢性疾病”,FODMAP饮食、排除食物不耐受的饮食法等,就可以干预很多胃肠道疾病,胃肠道疾病也是最刚需的市场。
目前国内外已经有很多商业公司推出多种慢性病提供个性化营养计划,包括糖尿病、过敏、自身免疫问题和胃肠道问题。
不管通过怎样的数据驱动,为“任何地方的任何人提供一种方便且负担得起的方式,利用食物作为药物并实现可持续营养实践来推进个人护理“”应该是未来精准或个性化营落地的基本要求。
为“任何地方的任何人”、“方便且可负担、可持续个性化健康调理和营养方案”,也是我们谷禾健康长期追求的理念。
主要参考文献
Bailey RL, Stover PJ. Precision Nutrition: The Hype Is Exceeding the Science and Evidentiary Standards Needed to Inform Public Health Recommendations for Prevention of Chronic Disease. Annu Rev Nutr. 2023 Aug 21;43:385-407.
Rodgers GP, Collins FS. Precision Nutrition-the Answer to “What to Eat to Stay Healthy”. JAMA. 2020 Aug 25;324(8):735-736.
Demetrowitsch TJ, Schlicht K, Knappe C, Zimmermann J, Jensen-Kroll J, Pisarevskaja A, Brix F, Brandes J, Geisler C, Marinos G, Sommer F, Schulte DM, Kaleta C, Andersen V, Laudes M, Schwarz K, Waschina S. Precision Nutrition in Chronic Inflammation. Front Immunol. 2020 Nov 23;11:587895.
Zeb F, Osaili T, Obaid RS, Naja F, Radwan H, Cheikh Ismail L, Hasan H, Hashim M, Alam I, Sehar B, Faris ME. Gut Microbiota and Time-Restricted Feeding/Eating: A Targeted Biomarker and Approach in Precision Nutrition. Nutrients. 2023 Jan 4;15(2):259.
Lee BY, Bartsch SM, Mui Y, Haidari LA, Spiker ML, Gittelsohn J. A systems approach to obesity. Nutr Rev. 2017 Jan;75(suppl 1):94-106.
Wellens J, Vissers E, Matthys C, Vermeire S, Sabino J. Personalized Dietary Regimens for Inflammatory Bowel Disease: Current Knowledge and Future Perspectives. Pharmgenomics Pers Med. 2023 Jan 12;16:15-27.
Zhu Z, Li YL, Song S. Editorial: Biomarkers: precision nutrition in chronic diseases. Front Nutr. 2023 Jul 24;10:1257125.
Dashti HS, Scheer FAJL, Saxena R, Garaulet M. Timing of Food Intake: Identifying Contributing Factors to Design Effective Interventions. Adv Nutr. 2019 Jul 1;10(4):606-620.

谷禾健康

当地时间12月19日,微软联合创始人、亿万富翁比尔·盖茨发布了对来年的年度预测,称 2024 年将是一个“转折点”。
在这封长达 10 页的信中他展示了对人工智能领域的更多创新、婴儿营养不良问题的突破、气候变化谈判的进展等多方面的期待。
人工智能可以让世界变得更加公平。盖茨对人工智能的预测超越了去年的超声波预测,他断言人工智能的进步将广泛改善全球健康,同时促进创新。
人工智能可以帮助世界各地对抗抗生素耐药性疾病,识别高风险妊娠并估计孕龄等,他预计人工智能在开发新药、诊断疾病和增强教育资源方面将发挥至关重要的作用。
盖茨表示,世界在利用肠道微生物补充剂来解决儿童营养不良方面即将取得突破。
本文我们来看看盖茨眼中的儿童营养不良问题及解法,盖茨预计,随着对微生物组的深入研究,人们将能够开发出以微生物为基础的新型营养治疗方案,这将给解决全球儿童营养危机带来重要进展。
期待已久的营养不良问题的突破即将到来
在盖茨基金会,我们愿意下大赌注。我们知道每一次冒险都可能没有回报,但这没关系。我们的目标不仅仅是逐步取得进展。我们的目标是将我们的努力和资源投入到重大项目中,这些项目一旦成功,就能拯救和改善生命。
当你下一个大赌注时,你往往要等待很长时间才能看到它是否有回报。当你终于意识到它会成功时,那种感觉是难以置信的。我最兴奋的一次豪赌就接近了这一时刻:利用我们对肠道微生物群的了解来预防和治疗营养不良。
经常有人问我,如果只能解决一个问题,我会选择什么。我的答案不变:营养不良。这是世界上最严重的健康不平等现象,大约每四个儿童中就有一个受到影响。如果在生命的头两年得不到足够的营养,身体和智力都无法正常发育。通过解决营养不良问题,我们可以减少导致儿童死亡的最大因素之一。
营养不良的原因远比没有得到足够的食物要复杂得多。大约15年前,研究人员注意到脊髓灰质炎等口服儿童疫苗在营养不良率高的地区效果不佳,他们开始怀疑生活在肠道中的微生物群可能在其中发挥了作用。很明显,有什么东西妨碍了它们被正常吸收。
这种怀疑在2013年得到了证实,当时生物学家杰夫·戈登(Jeff Gordon)发表了一项有里程碑意义的研究,关于马拉维一对双胞胎婴儿的微生物群。微生物群不仅是健康的副产物,还是健康的决定因素。这是第一条重要线索,表明我们或许可以通过改变肠道微生物群来减少营养不良问题。
经常有人问我,如果只能解决一个问题,我会选择什么。我的答案不变:营养不良
在过去的十年里,我们对肠道微生物群的了解比之前的1000年还要多。我们发现,生活在肠道中的细菌可能处于功能失调状态,从而导致炎症,使人无法吸收营养。我们发现,如果及早干预,就能最大程度地改善肠道微生物群。
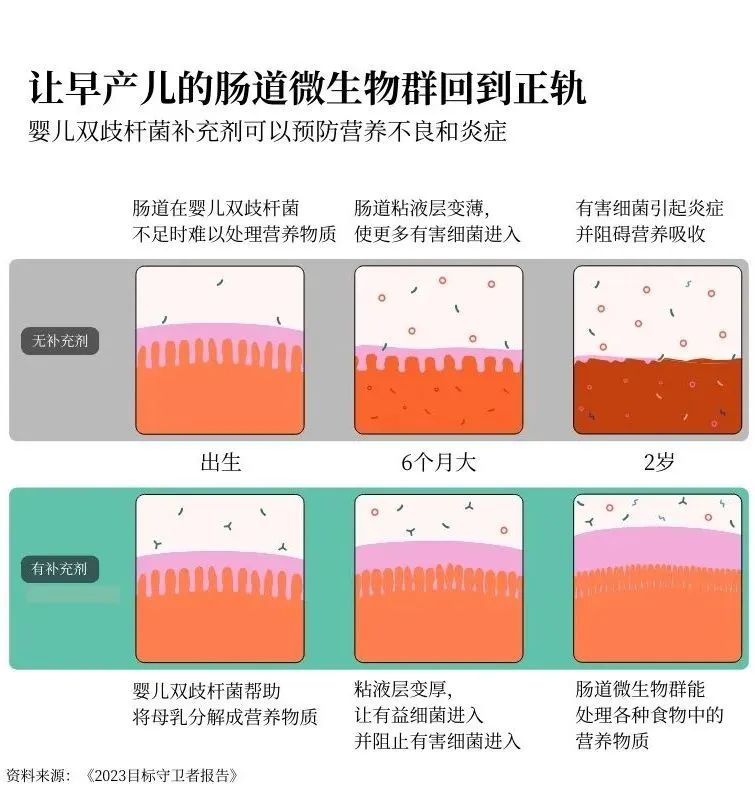
在人类发育过程中最先出现的肠道细菌之一叫做婴儿双歧杆菌。它能帮助将母乳中的糖分分解成人体生长所需的营养物质。反过来,母乳又为婴儿双歧杆菌和整个肠道微生物群提供食物。这是一个良性循环。但是,如果婴儿一开始没有足够的婴儿双歧杆菌,他们就可能无法从母乳中吸收足够的营养来支持其他必要的肠道细菌的生长。
要克服这种缺陷几乎是不可能的。你可以获得世界上所有富含营养的食物,但这并不管用。如果你的肠道生长路径过早被打乱,你可能永远无法吸收到所需的全部营养。
但是,如果我们能给高危婴儿服用婴儿双歧杆菌作为益生菌补充剂呢?我们能否及早干预,让他们正常发育?
这正是基金会合作伙伴多年来一直在研究的问题——我们终于找到了答案。我们正在对一种可添加到母乳中的婴儿双歧杆菌粉末补充剂进行三期临床。来自5个国家的16000名婴儿参与了试验,研究人员正在对每个婴儿进行跟踪,以确保益生菌既安全又有效。
目前的研究结果令人惊叹:通过给婴儿喂食这种益生菌,可以帮助他们的微生物群进入积极状态,从而使他们能够长大并充分发挥潜力。这对预防营养不良大有裨益。
在婴儿出生前就能改善肠道微生物群,这也让我感到兴奋。新的研究发现,婴儿的微生物群与母亲的微生物群息息相关。解决子宫内的炎症问题会给产妇、胎盘和发育中的胎儿带来更多益处。
如果我们能给准妈妈提供一种益生菌补充剂,让她的孩子从出生第一天起肠道就正常发育,那会怎么样呢?目前还不清楚这些活体生物药到底是什么样的,也不清楚如何给药,这还只是非常早期的研究。但研究表明,健康的微生物群可帮助婴儿在妊娠晚期每天增加5克体重。
在过去十年里,儿童健康领域发展速度超出了我一生中的预期。看到微生物群从一个完全看不见的东西变成解决世界上最大健康不平等之一的关键策略,真是令人惊叹。我迫不及待地期待在接下来一年里我们将学到多少新知识,以及我们将如何运用这些知识来拯救生命…
——比尔·盖茨
儿童营养不良问题的现状如何?带来什么问题?
儿童营养不良问题给全球健康带来巨大挑战,据世界卫生组织和联合国儿童基金会的数据统计,到 2020 年,估计有1.49亿 5岁以下儿童生长发育迟缓(年龄身高低),而 4500万儿童出现消瘦(WLZ 低)。
营养不良及其长期后遗症是该年龄段人群发病和死亡的主要原因。后遗症包括线性生长、免疫和代谢功能以及神经发育的持续损害——所有这些都对当前的干预措施有很大的抵抗力。
从比尔·盖茨的年度总结我们也可以看到,他十分重视肠道微生物对健康的影响,总的来说,微生物方面的研究给儿童健康带来了很大希望。
从肠道微生物组的角度入手,对营养不良儿童进行基于微生物群的相关干预措施,比尔及梅琳达·盖茨基金会已支持多项相关研究。我们来看近期得到该基金会支持的两篇研究文献。
在健康儿童中,肠道微生物组在分类和功能多样性方面稳步增长,直到3岁,其中最明显的变化发生在断奶期间。相比之下,营养不良儿童的肠道微生物组是年龄倒退的,即肠道微生物组与年幼儿童相似。
抗生素是严重急性营养不良的标准治疗方法,因为即使儿童没有出现明显的疾病,他们也可能因急性感染而突然恶化。
在马拉维和尼日尔,与安慰剂相比,短期(即7天)阿莫西林给药已被证明在降低全因死亡率、住院率、腹泻病和改善人体测量方面具有益处。
然而,抗生素治疗对严重急性营养不良儿童的潜在后果(如抗生素耐药性的发展和微生物组破坏)仍存在不确定性。
研究设计
研究人员想要确定 7 天的阿莫西林治疗,对接受严重急性营养不良治疗的儿童肠道微生物组和抗生素耐药组的急性和长期变化的影响。该成果发表在《Lancet Microbe》。

研究人员对尼日尔门诊治疗的严重急性营养不良儿童(6-59 个月)的阿莫西林随机、双盲、安慰剂对照试验 (NCT01613547) 进行了二次分析。从2013年9月23日至2014年2月3日从整个队列中随机选择了161名儿童(n = 2399)进行最初12周的随访。
根据人体测量结果从这161名儿童中选择了一个方便样本,2年后(2015年9月28日至10月27日)进行随访。儿童在基线、第 1 周、第 4 周、第 8 周、第 12 周以及 2 年随访队列中的第 104 周提供了粪便样本。研究人员进行了宏基因组测序,然后对粪便样本进行了微生物组和耐药组分析。38 名无严重急性营养不良的儿童和 6 名与原始队列基线年龄相匹配的严重急性营养不良儿童被用作参考对照。
研究结果
结果表明,营养不良的尼日尔儿童在接受抗生素治疗后,虽然发现克雷伯氏菌属、埃希氏菌属等增加,这些菌可能与更严重的感染相关,但是,微生物组和耐药组扰动的负面影响似乎是短暂的,在三周内完全消失。
进一步观察到阿莫西林治疗的一些意想不到的长期益处,包括改善长期微生物组丰富度,多样性和成熟度。
阿莫西林对长期微生物组成熟的影响
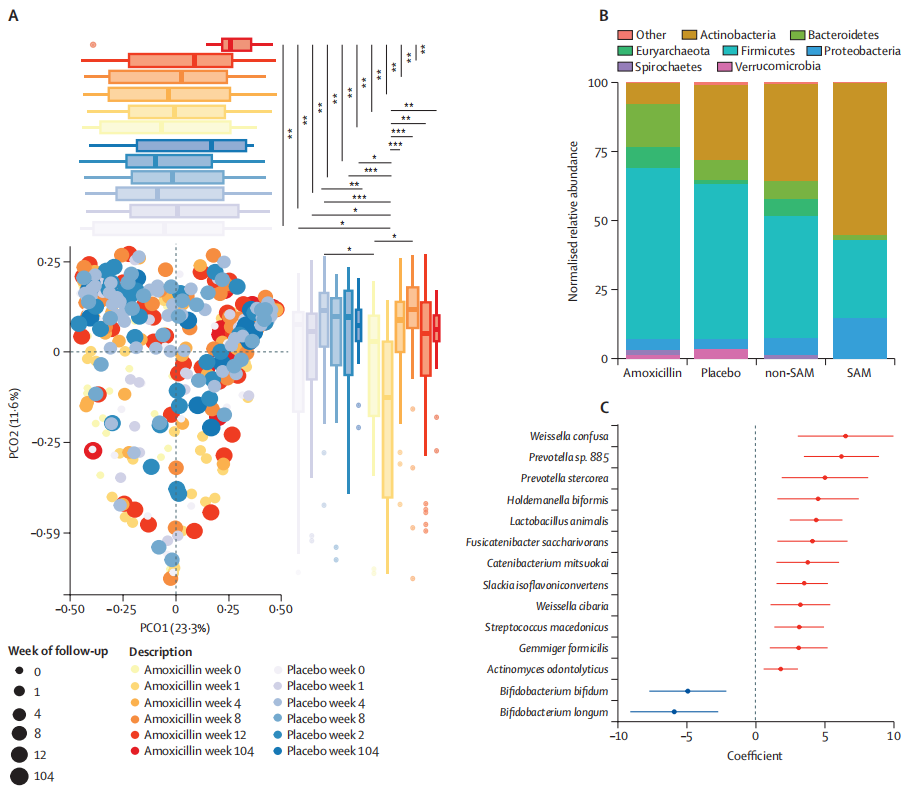
doi.org/10.1016/S2666-5247(23)00213-6
生命头三年肠道微生物组急剧成熟的一些关键驱动因素包括婴儿在出生期间和出生后接触母体微生物、加入牛奶、奶粉以及向固体食物的过渡。在营养不良的儿童中,肠道微生物组的成熟过程和生长都受到阻碍。但在治疗计划中加入抗生素改善了他们的身体测量和微生物组发育。
抗生素可能起到重置的作用,使有助于固体食物消化的微生物群在肠道生态系统中繁衍生息。
“
接受抗生素治疗的效果,对严重营养不良儿童来说算是个好消息,对于这一特定人群,抗生素治疗的益处似乎大于风险,但风险确实存在。研究人员警告说,儿童中已知的耐药细菌和耐药基因的增加不应被忽视。
营养干预措施,如即食治疗性食品,已被证明可以暂时改善肠道微生物组的成熟度和人体测量得分;然而,这种改善并不总是持续的。
在过去的5年里,合理设计的以微生物群为导向的治疗性食品在中度和重度急性营养不良儿童的微生物组恢复和人体测量得分方面显示出了希望。
接下来我们来看对于微生物群为导向的治疗性食品研究的最新进展。
生命头两年,肠道微生物群的发育需要与身体其他部分、其他器官系统的发育同步。当这个微生物群不完全形成时,仅仅增加热量是无法修复的。研究人员试图寻找特定的食物成分,以滋养健康的肠道微生物群,希望修复营养不良儿童肠道微生物群的功能失调。
使用传统食疗食品治疗可减少死亡,但并不能实质性改善营养不良的其他长期影响,包括新陈代谢、骨骼生长、免疫功能和大脑发育问题。
近日,来自华盛顿大学医学院的一项研究,已经确定了一种新型治疗食品中天然存在的关键生化成分,以及处理这些成分的重要细菌菌株。
该研究表明,识别这些成分以及作为其治疗靶标的关键促生长肠道细菌菌株,研究强调了了解细菌菌株如何加工特定食物成分的重要性,可以帮助指导当前食品配方的治疗,并可以在未来创造新的、更有效的配方。该研究成果于2023年12月13日发表在《Nature》杂志。
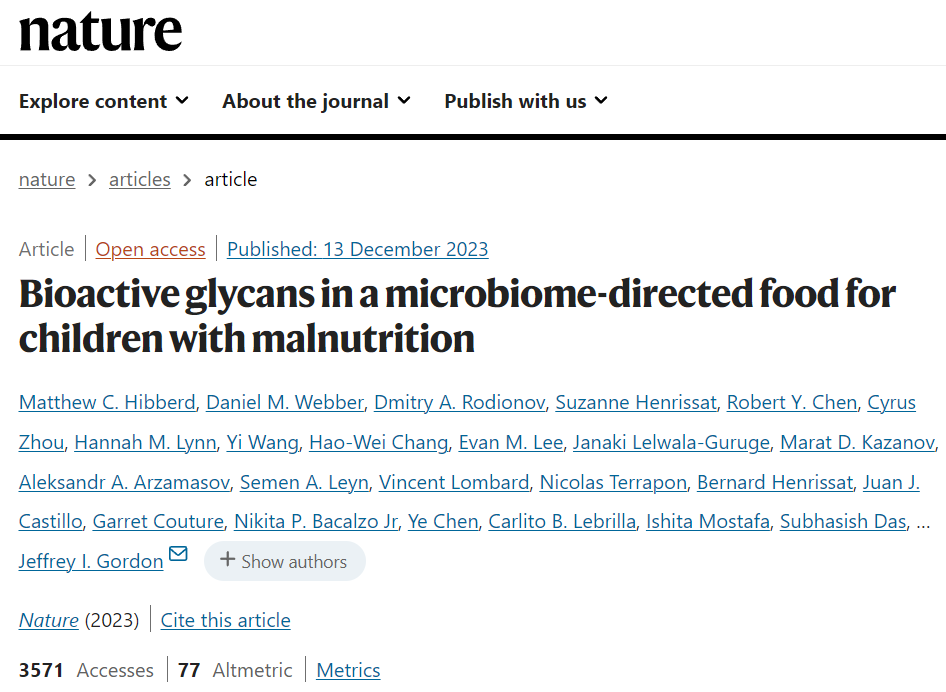
研究人员从试验参与者的粪便微生物群中重建了1000个细菌基因组(宏基因组组装基因组,简称MAG),确定了75个丰度与体重生长(体重长度Z评分变化,简称WLZ)正相关的MAGs。将MAG基因表达的变化表征为治疗类型和WLZ反应的函数,并定量MDCF-2和粪便中的碳水化合物结构。
结果表明,两个与WLZ呈正相关的普雷沃氏菌MAG是MDCF-2诱导的代谢途径表达的主要贡献者,这些代谢途径涉及利用MDCF-2的组分聚糖。
结果强调了微生物组反应的显著菌株特异性,并指出两种普氏菌菌株(MAG Bg0018 和 MAG Bg0019)是 MDCF-2 聚糖代谢和宿主体重生长反应的关键介质。
在生长相关细菌分类群代谢的MDCF中鉴定生物活性聚糖结构,有助于指导关于其在急性营养不良儿童中使用的建议,并有助于开发其他制剂。
在这项工作的基础上,世界卫生组织与比尔及梅琳达·盖茨基金会正在支持一项大型多站点临床试验,研究这种新的治疗性食品——MDCF-2,或微生物组导向的补充食品。
MDCF-2 治疗性食品包括哪些食物成分?
在 2021 年的临床试验中,MDCF-2 治疗性食品作为膳食补充剂提供,以提供儿童每日能量需求的约 20%。
Prevotella copri 两个菌株对MDCF-2干预改善营养不良的关键作用
研究人员发现:普雷沃氏菌Prevotella copri与儿童的生长发育呈正相关。
P. copri是拟杆菌门的成员,该门的成员含有一组叫多糖利用基因座(PUL)的基因,这些基因介导特定聚糖或一组聚糖的检测、导入和代谢。
它们在利用MDCF-2治疗食品中有益的生物活性碳水化合物结构的代谢途径中表现出活性的增加。
与接受传统治疗性食物的儿童相比,接受MDCF-2的儿童血液中支持肌肉骨骼生长和神经发育的某些蛋白质水平较高,与炎症有关的蛋白质水平较低。也表明,微生物群修复的作用远不止于肠道。
研究人员对这些儿童的粪便样本进行了广泛的基因组分析,了解对这些食物成分有反应的细菌,以及这些细菌对治疗的反应所具有和表达的代谢能力。事实证明,许多与儿童生长密切相关的细菌富含碳水化合物代谢的途径。
分析不同食物中的关键有效成分
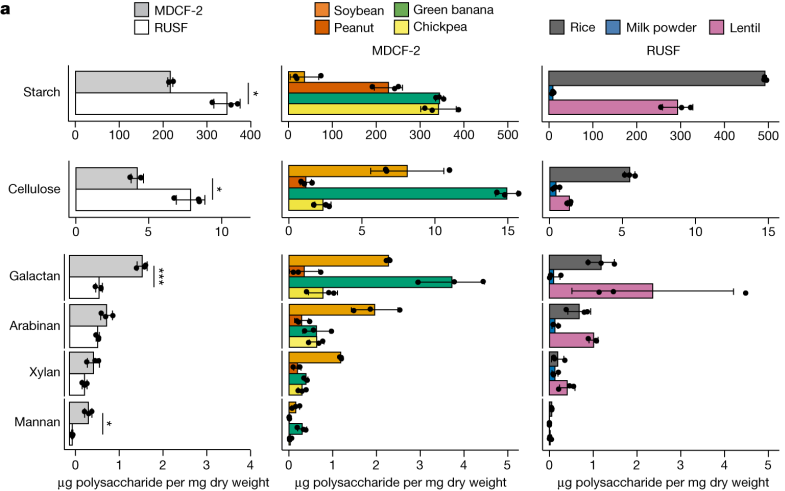
在该研究中,通过质谱分析MDCF-2和RUSF两类食品,确定其组成成分。
MDCF-2、RUSF 及其食品成分中的多糖
DOI: 10.1038/s41586-023-06838-3
对这些食物详细分析表明,MDCF-2食物中L-阿拉伯糖、D-木糖、L-岩藻糖、D-甘露糖和D-半乳糖醛酸 (GalA) 显著更丰富(P < 0.05)。
也就是说,MDCF-2含有更多的多糖,即半乳聚糖和甘露聚糖。而传统的治疗性食物RUSF含有更多的淀粉和纤维素。
MDCF-2 有效提高营养不良儿童生长
尽管MDCF-2的热量密度比高热量食物低15%,但与接受传统补充食物的儿童相比,接受MDCF-2治疗的儿童表现出更高的生长速度。
与MDCF-2治疗相关的生长增加与儿童微生物群中某些普氏菌菌株中存在的代谢途径的表达增加有关,这些途径与生物体利用MDCF-2中存在的多糖有关。
“来自食物频率问卷的信息表明,对MDCF-2反应最大的儿童也食用了更多的坚果和豆类作为他们日常饮食的一部分” ,研究人员表示,“这些坚果和豆类具有一些与MDCF-2相同的多糖。这表明可能有机会调整MDCF-2的成分和剂量,以进一步增强其治疗效果”。
营养不良儿童“微生物组修复”的一个定义是,重新平衡有益菌的表现和表达功能,使其呈现出更有利于健康微生物组-宿主共同发育的构型。
以这种方式将膳食聚糖和微生物代谢联系起来,为基于培养的计划提供了一个起点,这些计划旨在检索这些“效应”类群的分离物,用作潜在的益生菌制剂,或者如果与他们渴望的关键营养素相结合,修复对单独基于食物的干预反应不足紊乱的微生物群,提供合生元配方。
当然,P.copri菌株和MDCF-2聚糖之间的关系并不排除其他宏量或微量营养素对MDCF-2在增重方面优于RUSF的贡献。
总之,为改善微生物群的营养干预提供了一种新的见解。
这些和其他研究的结果有助于加深我们对微生物如何与人类细胞和器官合作,从而影响发育生物学的理解,帮助相关产业科研人员开发微生物组导向疗法的方法,帮助开发构建儿童营养健康的新策略,以确保在出生后的头几年形成健康的微生物组,尽可能规避儿童营养不良风险。
同时,随着人工智能技术及高通量测序等先进技术的不断发展,整合食物-微生物-人体交互作用研究,通过对婴幼儿的肠道菌群进行定期检测,可以帮助家长更好地监测儿童肠道菌群的变化,及时发现异常,采取相应的纠正措施,有利于促进儿童的身心健康发育。

谷禾健康

“肚子里有蝴蝶”这句话的词源最早出现在弗洛伦斯·匡威(Florence Converse) 于 1908 年撰写的《祈祷之家》一书中。一百多年来,这句话一直被广泛用作成语,它描述了一个人面对压力或惊心动魄的事件时的不安感。
扑腾的蝴蝶不仅隐喻我们体内扑动的感觉,它也是一个术语,描述了一个巨大事件的一系列连锁反应中的最初动作。
特别令人迷惑和应该注意的是,人们将这种感觉描述为起源于肠道而不是其他地方的东西。二十多年来,科学家们一直在追寻这些“蝴蝶”及其起源,他们意识到这种成语可能与受胃肠道(GI)共生微生物影响的“感觉”有关。这些年科学家们在了解共生肠道微生物在人类生理学和病理生理学中的重要性方面取得了惊人的发现。

肠道中的共生微生物通过“肠-脑轴”对宿主行为产生各种影响。 “肠脑轴”是胃肠道系统和中枢神经系统之间的远端连接;它由跨越两个身体系统的复杂信号转导途径组成(这方面详细的文章可以翻看以往谷禾发的相关文章)。肠道细菌及其代谢物发挥“蝴蝶效应”,将信号传播到大脑,最终改变宿主的行为。
下丘脑-垂体-肾上腺 (HPA) 轴是压力调节的经典途径,是将共生肠道微生物、胃肠道、大脑和行为相互连接的最有前途的途径之一;这也反映了肠道内的颤动感。此外,压力信号可以通过迷走神经和传入/传出神经元连接传输到大脑。
面临威胁时通常会感到压力、紧张、担心和焦虑。最近的研究结果表明,应激反应和肠道微生物会相互影响宿主的许多行为结果。为了了解共生肠道微生物在应激调节和反应中的作用,许多研究使用限微生物动物、16S rRNA 测序、宏基因组测序、粪便微生物群移植、抗生素治疗和益生菌来揭示宿主-微生物之间相互交织的相互作用。
本文重点关注啮齿类动物作为模式生物,探索肠道微生物群、压力和行为之间的因果关系。还纳入了一些临床观察结果来深入说明问题。
✓
心理压力是一个全球性问题,影响着全世界至少三分之一的人口,并增加了患多种精神疾病的风险。这几年越来越多的证据表明,肠道及其栖息的微生物可以调节压力和与压力相关的行为异常。
当面临厌恶压力刺激时,压力感知、整合和应对是大脑的重要功能。压力相关信息被整合到感觉皮层,然后将信号发送到边缘系统、下丘脑和脑干,以激活 HPA 轴以及交感神经和副交感神经。
交感神经和副交感神经传播应激反应以引起体内各个系统的快速适应。检测来自外部环境的压力信号的大脑区域与参与情绪的大脑区域重叠,从而协调一致地协调动物或人类的压力反应。
下丘脑室旁核的核心作用:整合压力信号和调节应激反应
在参与调节应激反应的大脑区域中,下丘脑室旁核 (PVN) 在整合来自环境刺激的信号并进一步触发下游神经传递方面发挥着核心作用。 PVN 接受来自边缘系统和脑干的神经支配,调节 HPA 轴并整合应激后的反应。 PVN 中分布着各种类型的神经元。主要是,PVN 和其他相关大脑区域中的促肾上腺皮质激素释放激素 (CRH) 神经元对不同形式的压力做出反应。
注:促肾上腺皮质激素释放激素 (CRH) 是一种由下丘脑室旁核 (PVN) 产生的神经肽。它是下丘脑-垂体-肾上腺轴 (HPA轴) 中的关键调节因子之一。CRH的主要功能是刺激垂体前叶分泌促肾上腺皮质激素 (ACTH)。ACTH进而刺激肾上腺皮质分泌皮质醇和其他类固醇激素。CRH的释放通常受到多种刺激因素的调控,如应激、体内环境的变化和生物节律。CRH的异常释放与许多疾病和症状有关,如抑郁症、焦虑症和肾上腺皮质功能不全等。
在下图中,总结了PVN中的CRH神经元与终纹床核(BNST)和杏仁核细胞在应激反应中相互作用的发现。这些大脑区域对于确定循环皮质酮水平和动物行为输出至关重要。
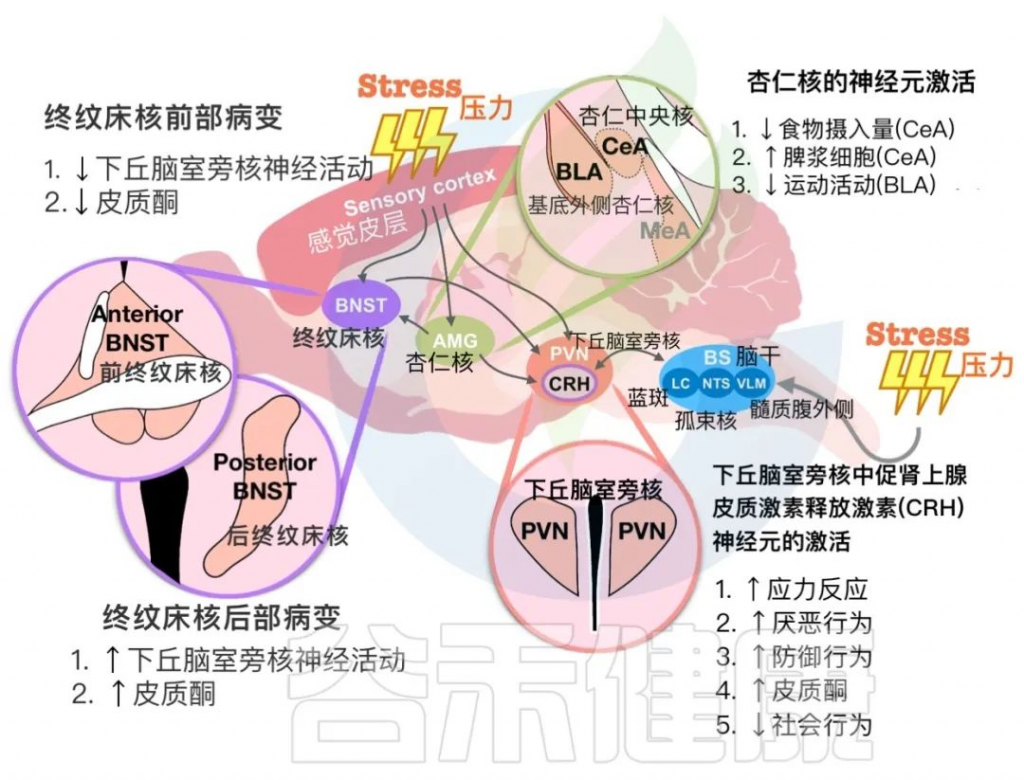
下丘脑室旁核(PVN)与其他大脑区域对压力暴露的反应
doi: 10.1186/s12929-023-00984-6
注:循环皮质酮是指在血液中循环的一种皮质酮激素,也被称为血浆皮质酮。皮质酮是由肾上腺皮质分泌的一种类固醇激素,它在机体的应激反应中起着重要的作用。循环皮质酮的水平在应激反应中通常会升高,它可以促进葡萄糖的产生、调节蛋白质和脂肪代谢,并参与免疫和炎症反应。
作为应激反应的中枢,PVN CRH 神经元可以被应激刺激触发,随后引发多种内在行为反应。
研究表明,在迫在眉睫的严峻任务中可以检测到 PVN 中 CRH 神经元活动的增加,这是一种模仿来自天空的捕食者威胁的方法,其中大多数小鼠表现出逃避行为,对迫在眉睫的阴影几乎没有冻结反应。
注:冻结反应是指在面对威胁或危险时,个体表现出明显的静止和冻结的反应。这种反应是动物和人类在面对潜在的威胁时的一种自我保护机制。当个体感知到威胁时,自主神经系统会释放肾上腺素等应激激素,导致身体进入一种高度警觉和紧张的状态。在这种状态下,个体可能会减少运动、停止呼吸、固定目光等,以最大程度地减少被威胁的可能性。这种反应在野生动物中常见,并且在人类中也可以观察到,尤其是在面对极端的恐惧、惊吓或创伤性事件时。增加冻结反应有助于个体在危险环境中保持相对安全,但在某些情况下也可能导致无助和无法应对的感觉。
PVN中的CRH神经元:与逃避行为和冻结反应的调节有关
沉默 PVN CRH 神经元会减少逃避行为,但会增加冻结反应。此外,PVN CRH 神经元预测迫在眉睫的威胁并编码压力可控性。同样,另一项研究表明,PVN 中的 CRH 神经元会对厌恶刺激做出反应,例如强迫游泳、尾巴约束、头顶物体、迫近,甚至腹腔注射。
PVN中的CRH神经元:与易感内脏疼痛的关联性
在一项研究中,与弹性小鼠相比,在小鼠经历母体分离后,易感内脏疼痛的小鼠在下丘脑室旁核(PVN)中显示出c-Fos阳性的促肾上腺皮质激素释放激素(CRH)神经元数量增加。
c-Fos是一种早期基因转录因子,它在神经元受到刺激后会被激活,并表达出来。因此,c-Fos的阳性表达可以作为神经元活动的标志。
在这种情况下,研究发现,在经历母体分离后易感内脏疼痛的小鼠中,PVN中的CRH神经元数量增加。这表明这些神经元在应激反应中被激活,并可能参与调节内脏疼痛的感受和处理。
PVN中的CRH神经元:在光遗传学刺激下对小鼠行为和压力信号传递的调控作用
研究发现,通过光遗传学刺激PVN 中的 CRH 神经元,可以增加小鼠的自我梳理行为,并改变其他家庭笼行为,类似于经历了足部电击的小鼠。通过增加环境的威胁水平,可以减弱刺激下丘脑下垂体后叶神经元的光遗传刺激对自我梳理行为的影响。研究还发现,从受压小鼠到未受压小鼠的压力信号传递需要激活两者的下丘脑下垂体后叶神经元。在社交互动测试中,刺激下丘脑下垂体后叶神经元会抑制社交行为并增加挖掘行为,同时导致皮质酮水平升高。
与此相反,并非所有的应激反应都与下丘脑下垂体后叶神经元有关。通过光遗传学激活下丘脑向腹外侧延髓的兴奋性投射,而不是下丘脑下垂体后叶神经元,可以在未经应激暴露的情况下在小鼠中重现应激诱导的高血糖现象。尽管如此,这些研究表明,当面临迫在眉睫的威胁时,下丘脑下垂体后叶神经元对于控制应激反应和行为至关重要。
BNST中CRH神经元:在应激反应调节中的作用
BNST(终纹床核)是连接与情绪有关的各个脑区的中继站。杏仁核-BNST和BNST-PVN回路参与应激反应调节。先前的研究表明,BNST由几个亚区组成,并向下丘脑下垂体发送不同的投射。在BNST回路中光遗传学激活基底侧杏仁核可以预防接受社交挫败应激的小鼠的焦虑行为。BNST的前部病变抑制了应激刺激后下丘脑和下丘脑-垂体-肾上腺轴的激活。
注:终纹床核是是应激反应神经回路中的关键节点,与身体的压力反应、焦虑和成瘾有关。BNST 损伤会改变与压力相关的皮质醇释放。
BNST后部损伤会增加急性束缚应激后下丘脑下垂体中的皮质酮水平和c-Fos阳性细胞数。应激暴露也会影响BNST的神经活动。捕食者应激、十字迷宫和束缚应激可增强BNST中CRH神经元的神经活动。然而,抑制BNST中的CRH神经元无法恢复应激引起的社交障碍,表明BNST可能受应激影响但不直接调节应激反应。
杏仁核:在压力和情绪调节中的作用
杏仁核是一个关键结构,与情绪处理和对压力的生理反应相关。杏仁核的各个子区域参与不同的机制来调节不同类型的压力源暴露。急性心理压力会增加内侧杏仁核 (MeA) 中 c-Fos 阳性细胞的数量,并增强中央核中的抑制性神经元活动杏仁核 (CeA)。
然而,杏仁核和 PVN 之间有限的直接连接可以调节应激反应。应激引起的免疫失调与 CeA 中不同的神经元群有关。确定了 CeA/PVN 和脾神经之间在应激相关免疫调节中的回路。人工激活 CeA 和 PVN 中的 CRH 神经元可增加脾浆细胞的形成。
将小鼠置于高架平台上不仅增加了CRH神经元活性,还促进了脾浆细胞形成,表明CeA和PVN参与了应激诱导的免疫反应。当小鼠暴露于捕食者气味时,抑制 mSTN 中的 CRH 神经元会增加冻结和隐现行为的潜伏期,并缩短适应捕食者威胁的快速眼动-睡眠反应的持续时间。
其他大脑区域的CRH神经元:在应激反应中发挥作用
捕食者刺激促进小鼠从快速眼动睡眠中快速唤醒。 最近的一项研究。研究表明,在快速动眼睡眠期间,内侧底丘脑核 (mSTN) 中的 CRH 神经元因外部刺激而被捕食者气味暴露所激活焦虑和抑郁样行为 和 BLA 中的神经活动,从而增加食物摄入量。
其他研究还表明,外周注射脂多糖 (LPS) 会增加 CeA 中的神经活动,从而减少食物摄入。CeA 病变在全身注射白细胞介素 (IL)-1 后阻止了 CRH 和促肾上腺皮质激素 (ACTH) 的释放。
总体而言,压力暴露会触发 PVN CRH 神经元与其他大脑区域对各种刺激做出反应。 PVN CRH 神经元似乎是连接其他大脑区域以启动压力反应和应对机制的中枢。了解应激反应的中心通路对于发现肠道微生物调节的信号通路非常重要。
✓
压力应对机制和适应对于生存至关重要。动物以多种方式应对压力,包括生理和行为的变化。有趣的是,科学家通过啮齿动物模型发现压力暴露会影响肠道微生物群(下表)。
急性和慢性应激条件下共生微生物组和行为的适应



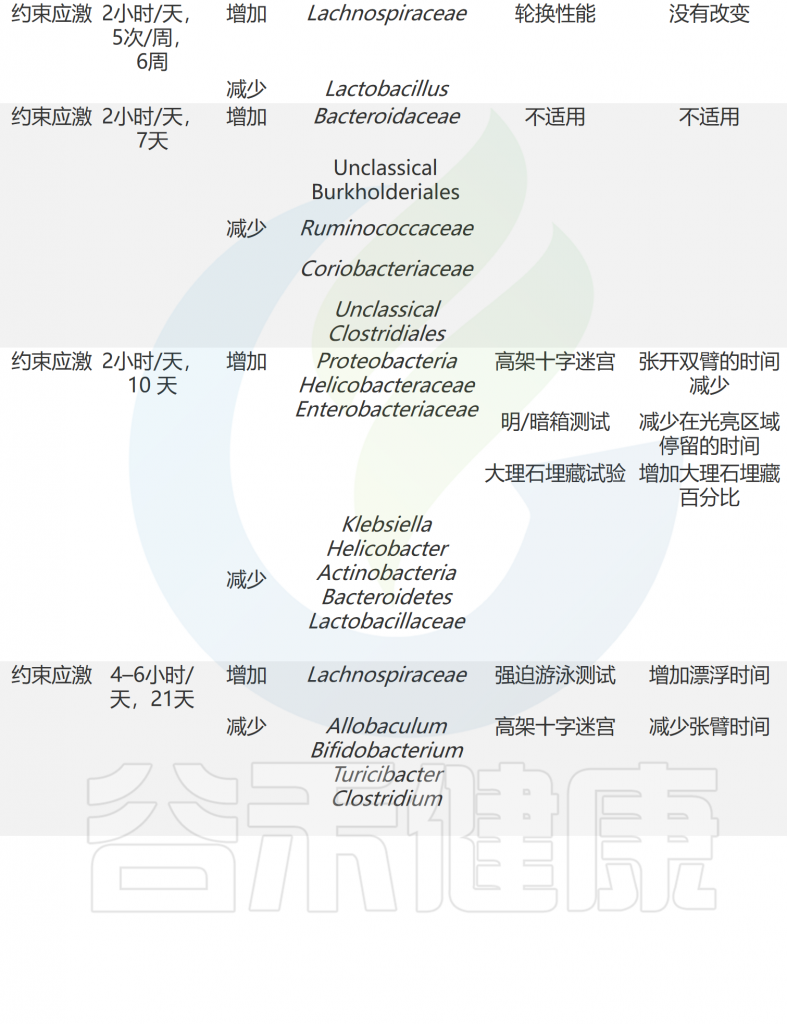
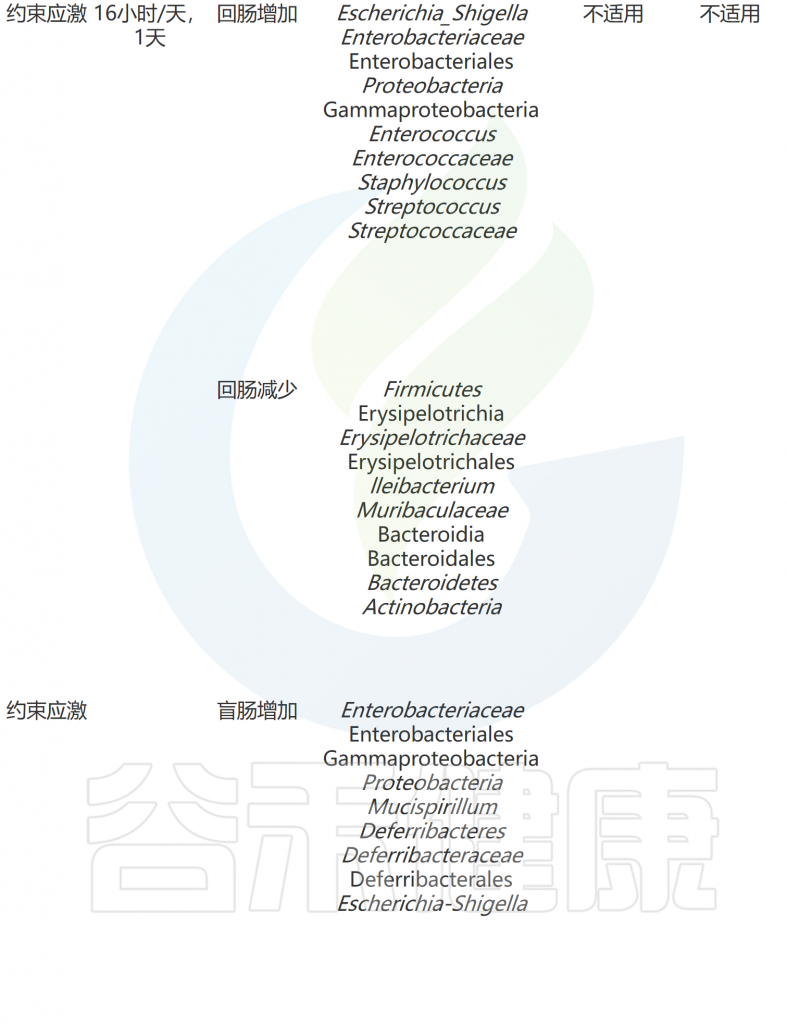


doi: 10.1186/s12929-023-00984-6
不可预测的慢性轻度应激
不可预测的慢性轻度应激 (UCMS) 是一种实验条件,会引起类似于慢性且未解决的应激暴露的生理和神经变化。该实验条件下的小鼠通常表现出抑郁样行为,与抑郁症患者相似,但焦虑样行为没有明显变化。
有趣的是,改变后的厚壁菌门和软壁菌门(Phylum Tenericutes)在 UCMS 动物中得到一致观察。
注:软壁菌门是细菌界下的一个门,典型物种如支原体。
值得注意的是,UCMS中乳杆菌科细菌减少了 。 而且肠道核心菌属-粪球菌属(Coprococcus)在 UCMS 小鼠和人类抑郁症队列中减少。
慢性社会挫败压力
慢性社会挫败压力(CSDS)是一种社会心理压力,具有非凡的面部、结构和预测有效性。CSDS后的行为结局很复杂,包括抑郁样行为、焦虑样行为增加,以及社交行为减少。同样,CSDS转移的微生物组分析比UCMS转移的微生物组分析更复杂。
慢性社交失败压力是一种实验动物模型,用于研究社交压力对动物行为和生理的影响。这个模型通常用于研究抑郁症和焦虑症等精神疾病的机制和治疗方法。
在CSDS模型中,实验动物(通常是小鼠)被暴露于反复的社交失败和攻击中,例如与更强壮的同种异性动物进行斗争。这种持续的社交失败经历会导致动物出现一系列的行为和生理改变,包括社交退缩、抑郁样行为、焦虑、食欲和睡眠紊乱等。
研究人员一般使用CSDS模型来研究社交压力对大脑神经回路、神经递质系统和免疫系统的影响,以及潜在的治疗策略,例如抗抑郁药物和行为疗法。这个模型有助于理解社交压力对心理健康的影响,并为精神疾病的研究提供了一个可靠的实验基础。
研究发现CSDS后的行为结果是复杂的,包括抑郁样行为、焦虑样行为的增加以及社交行为的减少。同样,CSDS引起的微生物组轮廓变化比UCMS引起的更为复杂。CSDS后,拟杆菌门和Helicobacteracea 的数量增加。相反,厚壁菌门中的一些细菌,如瘤胃菌科(Ruminococcaceae),在CSDS后发生了改变,唯独乳杆菌没有变化。
社交过度交叉
社交过度交叉(SOC)是一种模拟增加住房密度的方法。SOC对行为的影响很小。小鼠只在高架十字迷宫中显示出速度增加和进入光/暗盒中的黑暗室次数增加。然而,SOC后微生物组的变化更为显著。SOC增加了Akkermansia muciniphila和Anaerostipes的相对丰度,并减少了Erysipelotrichaceae家族、Lactobacillus和Bacteroides acidifaciens(产酸拟杆菌)物种的相对丰度。
约束压力
约束压力是限制啮齿动物运动的经典方法。啮齿类动物在受到约束压力后会出现类似焦虑和抑郁的行为。虽然肠道中的许多细菌类群发生了改变,但厚壁菌门似乎是最容易被慢性束缚应激改变的细菌,尤其是乳杆菌科、毛螺菌科(Lachnospiraceae)。
WAS小鼠是一种实验动物模型,用于研究自闭症和相关神经发育障碍。WAS代表”Wiskott-Aldrich syndrome”,是一种罕见的遗传性免疫缺陷病,患者通常表现出免疫功能异常、血小板减少和湿疹等症状。
WAS小鼠是通过基因突变技术,使小鼠的WAS基因发生突变,从而模拟人类WAS综合征。这些小鼠在行为和神经发育方面表现出与自闭症相关的特征,例如社交互动减少、沟通障碍、刻板行为和认知功能障碍等。
WAS是一种强烈的心理压力因素,会破坏肠道上皮紧密连接的完整性。与其他压力模型相比,单独的WAS对行为产生的影响不大。然而,WAS会影响粪便微生物组,导致拟杆菌门减少、厚壁菌门增加和变形菌门增加。当分析小肠和结肠的内容物时,WAS小鼠的乳杆菌科和未分类的拟杆菌门数量较低。
根据我们调查和总结,微生物组对压力的适应可能受到不同类型的压力、压力暴露时间、动物来源、饮食等因素的影响(下表)。在接受不同类型压力暴露后,已报道有几个细菌分类在不同研究中存在差异。
压力暴露会下会减少的细菌:
相反,压力暴露会增加的细菌:
注:研究人员观察到Anaerofustis菌与睡醒后30分钟内唾液皮质醇水平升高呈正相关
注:代表菌种幽门螺杆菌(Helicobacter pylori)
在这些研究中,Lactobacillus物种是在压力暴露后最一致减少的细菌分类。其余菌谷禾会逐步为大家科普。
✓
对小鼠的研究表明,压力暴露会改变肠道微生物组的组成并改变细菌分类群,这引出了另一个问题:肠道细菌是否在压力反应调节中发挥积极作用?
无菌和抗生素处理模型:揭示肠道微生物对应激反应的影响
为了解决这个问题,无菌和抗生素处理的啮齿动物是“敲除”共生菌群的理想模型,无论是持续性还是有条件的。令人惊讶的是,大多数研究表明,啮齿动物中的微生物群耗竭会增强应激反应并增加应激激素皮质酮。皮质酮是啮齿动物中的一种糖皮质激素(人类中的皮质醇),它是应激反应时分泌的重要类固醇激素。
然而,并非所有的研究都显示无菌啮齿动物在暴露于应激条件后会表现出过度的应激反应和更高水平的皮质酮。在测量无菌啮齿动物基线皮质酮水平时,尚未得出一致的结果。这些研究发现,下丘脑-垂体-肾上腺皮质轴(HPA)是肠道微生物改变宿主生理的重要调节因子;这引发了肠道微生物在应激抑制中可能发挥关键作用的可能性。
无菌啮齿动物明确表明肠道微生物的耗竭会导致异常的应激反应,包括皮质酮增加、与应激信号传导相关的基因表达改变以及异常的行为后果。虽然无菌模型是研究微生物对应激应对机制影响的宝贵工具,但需要注意下无菌模型的局限性。
无菌模型和抗生素给药模型:在研究应激反应时的局限性
无菌动物从出生开始就缺乏与微生物的接触,与常规饲养动物相比,它们可能表现出几个发育差异。这些差异包括肠道形态的改变、未成熟的黏膜免疫系统、口服耐受发育的延迟、上皮细胞周转减缓以及神经内分泌功能的改变,尤其是在早期生命阶段。对于这些差异的警告意识到了无菌模型在人体生理背景下的人为性质。
与无菌小鼠模型并行,广泛采用抗生素给药,以阐明肠道微生物在应激反应中扮演的角色。抗生素给药是一种控制共生微生物清除时间的有力工具。但是,年龄、治疗时间窗口、抗生素种类和剂量都是获得与无菌啮齿动物一致结果的关键因素。
此外,由于胃肠道不同区域对不同种类肠道细菌有地理选择性,完全清除肠道微生物还是一个挑战。只有少数研究能通过给予抗生素在无菌小鼠体内成功再现应激反应增强情况。
两项研究采用了类似的广谱抗生素药物组合(ABX),显示ABX的慢性治疗会导致小鼠基础皮质酮水平升高以及社交暴露后也会升高。
另外两项研究显示,口服单一抗生素的急性或慢性给药会导致小鼠在受到急性应激后,皮质酮水平升高。
皮质酮
皮质酮是一种 21-羟基类固醇,由在11和21位被羟基基团和氧代基团取代的孕 4-烯组成在位置3和20。皮质酮是在肾上腺皮质中产生的皮质类固醇类型的21碳类固醇激素。它具有人类代谢物和小鼠代谢物的作用。
糖皮质激素(人类为皮质醇,啮齿类动物为皮质酮)是类固醇激素,由肾上腺根据生理信号和压力以昼夜节律方式合成和释放。
肾上腺释放糖皮质激素的昼夜节律受下丘脑-垂体-肾上腺 (HPA) 轴调节。来自视交叉上核 (SCN) 的输入刺激下丘脑的室旁核 (PVN),释放促肾上腺皮质激素释放激素 (CRH) 和精氨酸加压素 (AVP)。
这些激素作用于垂体前叶,激活促肾上腺皮质激素细胞,将促肾上腺皮质激素 (ACTH) 分泌到体循环中。
随后,ACTH作用于肾上腺皮质,刺激糖皮质激素的合成和释放。一旦从肾上腺释放到血液循环中,糖皮质激素就会进入靶组织,调节多种生理过程,包括新陈代谢、免疫功能、骨骼生长、心血管功能、生殖和认知。
由于其亲脂性,糖皮质激素不能预先合成并储存在肾上腺中,但必须在 ACTH 刺激下快速合成(使用许多酶促反应)。 HPA 系统内的这种前馈机制通过作用于垂体前叶和下丘脑内的糖皮质激素的负反馈来平衡,以分别抑制 ACTH 和 CRH 的进一步释放。
因此,人类血清皮质醇浓度在早晨达到峰值,在夜间最低。 HPA 轴是中央应激反应系统,负责应激反应的适应部分,试图恢复体内平衡。应激反应的不当调节与多种疾病有关,包括自身免疫性疾病、高血压、情感障碍和重度抑郁症。全身血清糖皮质激素水平由肾上腺糖皮质激素合成维持,但糖皮质激素的可用性在组织或细胞水平上进一步调节。
有趣的是,用抗生素对小鼠进行发育期治疗在各种条件下降低了皮质酮水平。对大鼠进行抗生素治疗也会降低皮质酮水平,表明存在模型依赖效应。其他研究表明,抗生素对皮质酮水平没有影响。虽然抗生素给药是研究肠道菌群对大脑和行为影响的有效方法,但在实验设计中需要仔细考虑。
✓
小鼠大脑应激反应失调的现象在无共生微生物的小鼠中被广泛观察到。一些研究调查了肠道微生物耗竭小鼠中糖皮质激素受体(GR)、CRH及下游信号通路的基因表达水平。发现在应激暴露后,无菌小鼠海马CA1区和颗粒回(DG)中观察到皮质酮水平升高和GR mRNA水平降低。
无菌小鼠下丘脑中CRF表达水平较高。皮质中GR基因表达水平较低,但下丘脑和海马中未观察到这种情况。
在基线条件下,GF小鼠海马中的GR下游信号通路,包括Slc22a5、Aqp1、Stat5a、Ampd3、Plekhf1和Cyb561等基因表达上调。
在WAS后,与SPF小鼠相比,GF小鼠海马CA1区的神经活动减少。
最近的研究发现,肠道共生微生物对于抑制宿主的应激反应和增加社交行为至关重要。在与陌生老鼠进行短暂社交互动后,无菌小鼠的应激激素皮质酮水平升高。
与此同时,多个与应对应激有关的大脑区域的神经活动也得到了上调,包括下丘脑旁室核(PVN)、海马齿状回(DG)和肾上腺背侧床核(adBNST)。
在成年阶段接受抗生素治疗的小鼠中,这种应激激素和神经活动的上调现象也得到了重复。此外,这项研究还表明,GF小鼠的海马(Arc、Fos、cJun、JunB、Egr1、Egr2、Gadd45b、Gadd45g、Bdnf)和下丘脑(Arc、Fos、Egr1)中的即时早期基因表达上调,而脑干(cJun、JunB、Egr1、Gadd45b、Gadd45g、Bdnf)中的即时早期基因表达下调。
然而,在基线和应激条件下,接受抗生素治疗的小鼠中应激相关基因表达并没有发生改变。只有在社交互动后,ABX小鼠的Crh基因表达上调,而在暴露于新环境后,ABX小鼠的Ucn基因表达上调。
以上小编概括起来就是:
最新的研究发现,肠道微生物对于调节宿主的应激反应和社交行为非常重要。在实验中,当无菌的小鼠与陌生的老鼠进行短暂的社交互动后,它们的应激激素皮质酮水平会增加。
在接受抗生素治疗的成年小鼠中,这种应激激素和神经活动的增加现象也得到了重复。此外,研究还发现,没有微生物的小鼠的海马和下丘脑中的即时早期基因表达上调,而脑干中的即时早期基因表达下调。
然而,在接受抗生素治疗的小鼠中,无论是在正常状态还是应激条件下,与应激相关的基因表达并没有发生改变。只有在社交互动后,接受抗生素治疗的小鼠的某些基因表达会增加。这些研究结果表明,肠道微生物对于调节应激反应和社交行为起着重要作用。
应激反应神经元的干扰可以改变小鼠的社交行为和皮质酮水平
为了进一步研究神经元在脑回路中的干扰是否能够改变小鼠的应激激素和社交行为,采用了基因消除策略和化学遗传学方法来干扰接受抗生素治疗的小鼠中的应激反应神经元。
该研究通过对接受抗生素治疗的小鼠进行基因消除和化学遗传学方法的干预,发现破坏应激反应神经元可以恢复社交缺陷并抑制皮质酮水平。特别是在下丘脑中基因消除GR会降低社交行为,并在社交互动后增加皮质酮水平。沉默PVN CRH神经元可以抑制皮质酮水平的增加并阻止社交缺陷的发展(下图)。
肠道共生微生物是抑制宿主应激反应神经元增加社会行为所必需的
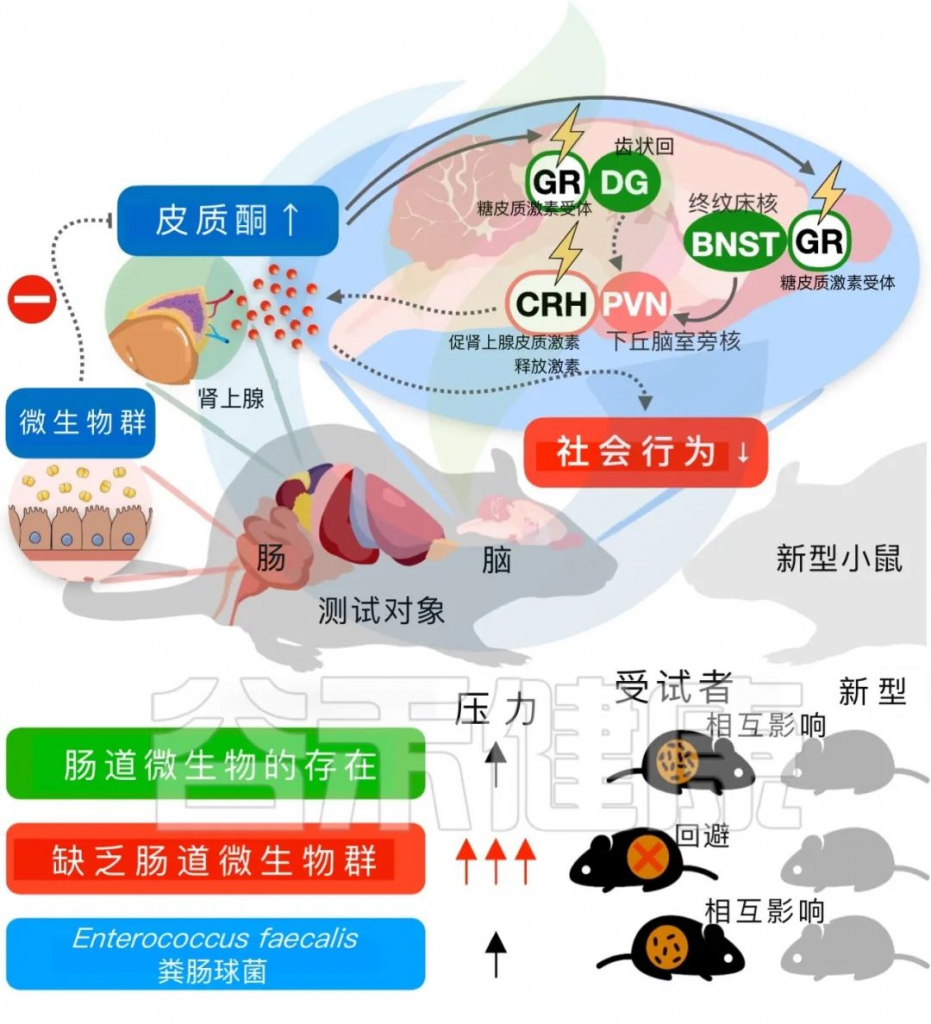
编辑
doi: 10.1186/s12929-023-00984-6
PVN中CRH神经元活动的改变,可能是社交行为和应激反应失调的原因
与此相反,adBNST CRH神经元的这些效应在ABX小鼠中未观察到。此外,去肾上腺和药物阻断GR以及皮质酮合成可以恢复微生物缺失小鼠的社交互动行为。因此,该研究认为,在没有微生物组的小鼠中,社交行为和应激反应的失调更可能是由于PVN CRH神经元的神经活动改变,而不是应激相关基因表达的改变或PVN相关神经回路的结构改变。这项研究为共生菌通过特定途径驱动宿主行为的应激应对提供了证据。
神经科学技术的进步使科学家能够精确地研究受微生物组调节的神经回路,并进一步发现微生物组介导的与应激相关的神经回路的机制。
✓
压力条件下大脑中的神经元已经得到了广泛的研究,但肠道源的压力信号尚未完全阐明。糖皮质激素是一类肾上腺皮质激素,主要由肾上腺分泌,部分由肾上腺外系统分泌。
尽管肾上腺糖皮质激素在应对压力中起着作用,但肠道中肾上腺外糖皮质激素的生理作用仍未被理解。
肾上腺中的糖皮质激素合成途径涉及线粒体中的一系列步骤:
基于合成皮质酮的生化过程的基本区别,肾上腺外糖皮质激素的生理作用被认为与应对压力无关。
肠道上皮细胞对肾上腺外糖皮质激素的合成贡献
肠道上皮细胞是肾上腺外糖皮质激素合成的主要产生者。小肠、大肠和阑尾中表达了参与类固醇合成的关键酶,包括Cyp11a1和Cyp11b1。肠道糖皮质激素被认为对肠道微环境有贡献。研究表明,系统免疫挑战会上调糖皮质激素合成,并与肠道免疫细胞相互作用。炎症因子肿瘤坏死因子α(TNFα)和脂多糖(LPS)诱导的免疫系统激活促进肠道糖皮质激素合成。然而,白细胞介素1β(IL-1β)在体外模型中抑制肠道上皮细胞的糖皮质激素信号传导。
糖皮质激素在肠道屏障功能和免疫调节中的作用
糖皮质激素激动剂地塞米松可增加上皮细胞的屏障功能,但与细胞因子联合处理可能影响屏障功能。长期使用地塞米松可能影响上皮细胞通透性和基因表达,从而改变细胞的结构和功能。细菌内毒素LPS注射后,肾上腺外组织产生的皮质酮水平显著增加。因此,肾上腺外糖皮质激素主要具有免疫调节功能,与典型的应激信号有所不同。
肠道糖皮质激素合成在IBD中的降低及其与疾病发展的关系
肠道糖皮质激素合成在炎症性肠病(IBD)患者中表达水平显著降低,暗示肠道糖皮质激素合成障碍可能参与了IBD的发展。肠道糖皮质激素在炎症期间起到抑制肿瘤发展和生长的关键作用。然而,在肿瘤期间,Cyp11b1介导的糖皮质激素合成抑制抗肿瘤免疫反应,促进免疫逃逸。这为肿瘤治疗提供了有希望的治疗靶点。这些发现突显了肠道糖皮质激素合成在调节胃肠道疾病中的重要作用。
通过对无菌小鼠的研究,了解到失去菌群会如何改变肠道的应激反应。在基线、免疫挑战和应激暴露条件下,无菌小鼠的肠道应激相关基因表达发生改变。
在社会挫败和急性束缚应激条件下,糖皮质激素合成基因表达差异
在社会挫败和急性束缚应激条件下,比较了SPF(无特定病原体动物)小鼠和无菌小鼠的垂体、肾上腺和肠道的糖皮质激素合成基因表达。简而言之,社会挫败应激后,SPF小鼠的结肠中的Crh和Ucn2基因表达上调,而GF小鼠中则保持不变,部分原因是GF小鼠基线水平的增加。
有趣的是,无论GF小鼠基线水平是否增加,在社会挫败应激后,Hsd11b1的下调在SPF小鼠和GF小鼠中都观察到。另一项研究调查了GF小鼠肠道中糖皮质激素合成的肠段特异性。急性束缚应激和菌群的存在改变了GF小鼠回肠和结肠中Nr5a2(编码LRH-1)和Hsd3b2的表达。然而,与回肠相比,结肠中的糖皮质激素合成基因的改变更为显著。
HPA轴对糖皮质激素分泌的调节
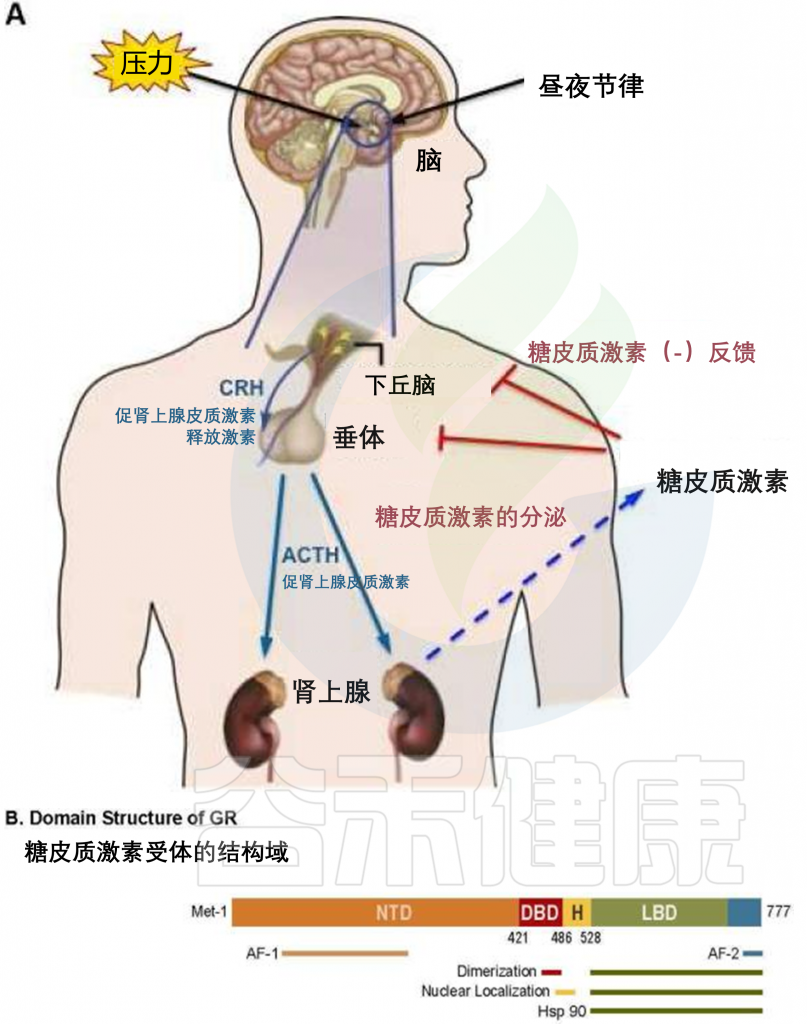
doi: 10.1016/j.rdc.2015.08.002
这些研究表明:肠道糖皮质激素合成可能是肠道细菌调节应激反应的关键途径。肠道细菌如何影响下丘脑-垂体-肾上腺轴(HPA轴)的精确机制尚不清楚。肠道外肾上腺糖皮质激素合成是一个值得研究的有希望的途径。
✓
乳杆菌(Lactobacillus)
鼠李糖乳杆菌 (L. rhamnosus GG)
鼠李糖乳杆菌作为益生菌已经使用了几十年。
鼠李糖乳杆菌GG减轻了母体分离大鼠和高脂饮食小鼠中急性约束应激诱导的皮质酮。
鼠李糖乳杆菌JB-1通过膈下迷走神经以应变依赖的方式降低急性约束应激诱导的皮质酮水平。母鼠分离后,幼鼠立即表现出高皮质酮水平。
口服鼠李糖乳杆菌R0011菌株(95%)和瑞士乳杆菌R0052菌株(5%)可以防止皮质酮水平增加。
除了鼠李糖物种外,副干酪乳杆菌、植物乳杆菌、干酪乳杆菌和其他物种已被证明在各种模型中调节应激。
副干酪乳杆菌 (L. paracasei )
副干酪乳杆菌Lpc-37的给药慢性降低了由慢性日常约束应激引起的皮质酮水平。
副干酪乳杆菌HT6有效地防止了早期生活压力社会经历引起的大脑GR表达变化。
副干酪乳杆菌PS23和植物乳杆菌PS128降低了早期生活压力诱导的皮质酮水平。
干酪乳杆菌(L. casei)
干酪乳杆菌Shirota菌株降低了大鼠WAS诱导的皮质酮水平和人类学业压力诱导的皮质醇水平。
干酪乳杆菌DKGF7抑制慢性约束应激诱导的皮质酮。
植物乳杆菌 (L. plantarum)
植物乳杆菌CCFM8610和干酪乳杆菌M2-01-R02-S01(M2S01)在WAS和柠檬酸杆菌诱导的肠易激综合征(IBS)模型中抑制皮质酮水平。
植物乳杆菌LRCC5310、植物乳杆菌LRCC5314和天然乳杆菌BNR17抑制了慢性冷应激和高脂饮食诱导的皮质酮升高。
路氏乳杆菌(L. reuteri)
路氏乳杆菌胞外多糖抑制氨苄青霉素诱导的皮质酮。路氏乳杆菌ATCC-PTA-6475在伤口愈合过程中下调皮质酮水平。
路氏乳杆菌NK33、约氏乳杆菌(L. johnsonii)分离株、约氏乳酸杆菌BS15和粘膜乳杆菌NK41抑制固定应激诱导的皮质酮升高。
其他乳杆菌
发酵乳杆菌(L. fermentum)CECT 5716减轻了WAS和母体分离诱导的皮质酮水平。用热灭活的发酵乳杆菌和德氏乳杆菌(ADR-159)治疗可降低皮质酮的基线水平并增加社交能力。
L.helveticus NS8可减少慢性约束应激诱导的皮质酮。用L.farciminis ML-7处理成功抑制了部分约束应力诱导的HPA轴的激活。
然而,并非所有乳杆菌物种都对应激反应产生下调作用,包括副干酪乳杆菌N1115、植物乳杆菌LP12407、植物乳菌LP12418,唾液乳杆菌UCC118、干酪乳杆菌CRL431、唾液乳杆菌HA113。此外,与安慰剂组相比,著名的益生菌鼠李糖乳杆菌JB-1不能改变皮质醇并释放压力。
双歧杆菌 (Bifidobacterium)
除了乳酸杆菌外,双歧杆菌是另一个已被广泛研究用于应激调节的细菌属。
B. subtilis在GF小鼠中的单克隆作用减弱了约束应激诱导的ACTH和皮质酮水平的增加。
青春双歧杆菌NK98、青春双歧杆菌IM38和长双歧杆菌NK46抑制固定应激诱导的皮质酮升高。
假链状双歧杆菌B. pseudocatenulatum CECT 7765和双歧双歧杆菌B.bifidum G9-1(BBG9-1)减轻了母体分离引起的皮质酮水平升高。
B.breve菌株M2CF22M7和CCFM1025减少了UCMS诱导的皮质酮产生。
在一项临床研究中,长双歧杆菌1714的给药降低了应激诱导事件后的应激激素水平。
同样,并非所有双歧杆菌物种都参与应激调节,包括婴儿双歧杆菌35624、短双歧杆菌UCC2003、长双歧杆菌1714、短芽孢杆菌1205。
益生菌混合物
结合乳酸杆菌和双歧杆菌的益生菌混合物也具有应激调节作用。L.helveticus R0052和B.longum R0175(Probio’Stick®)降低了WAS诱导的皮质酮升高。
用益生菌瑞士乳杆菌、鼠李糖乳杆菌、干酪乳杆菌和长双歧杆菌联合治疗可抑制UCMS大鼠的ACTH和皮质酮水平。
然而,不同细菌相互作用的机制可能很复杂。相反,母体使用B. animalis subsp. actis BB-12®和Propionibacterium jensenii 702可以增加新生儿皮质醇的水平。
其他不常见的益生菌
其他不常用作益生菌的细菌也在较小程度上调节应激诱导的激素。GF小鼠中大肠杆菌而非脆弱拟杆菌的单克隆作用降低了皮质酮的基础水平。给予克雷伯菌和大肠杆菌增加了皮质酮的基线水平。吴等人用抗生素(氨苄青霉素、万古霉素和甲硝唑;AVM)联合治疗小鼠,发现与全谱ABX治疗的小鼠相比,社交行为得到了保护,应激反应受到了抑制。当将AVM肠道微生物群移植到GF受体小鼠时,保留的社会行为和减少的应激反应被转移,表明AVM微生物群中的肠道细菌发挥了积极作用。
粪肠球菌——在社交活动中促进社交行为并抑制皮质酮水平升高的关键细菌
粪肠球菌(Enterococcus faecalis)是一种乳酸菌,对抗生素和许多其他压力源具有耐药性。粪肠球菌在宿主中的功能作用是多方面的,并且具有菌株特异性。
粪肠球菌是一种常见于尿路感染的常见病原体。另一方面,粪肠球菌已被广泛用作益生菌或食品添加剂。
粪肠球菌在ABX和GF小鼠中的定殖可以促进它们的社交行为,但只抑制ABX小鼠的皮质酮水平,而不是GF小鼠。
粪肠球菌可以调节神经系统和宿主行为
粪肠球菌EC-12菌株降低了焦虑反应,并改变了前额叶皮层中去甲肾上腺素和加压素的受体。
粪肠球菌SF3B菌株和EF-2001菌株已被证明可以缓解结肠炎诱导的肠道神经传递和病理。此外,粪肠球菌可以合成酪胺和苯乙胺,这两种神经活性分子被称为微量胺,能够调节宿主神经系统。
物质P刺激粪肠球菌V583菌株中酪胺和乳酸的产生,并增强肠道体外模型中的细胞毒性和细菌移位。
粪肠球菌AG5可增加宿主中的长链和短链脂肪酸,这可能通过间接方式间接影响神经系统。
一份报告发现,小鼠感染致病性粪肠球菌菌株K9和CP-1,会急性增加皮质酮,这表明粪肠球菌可以改变宿主中的糖皮质激素信号。临床上,粪肠球菌存在于89.3%的健康对照组中,而只存在于58.3%的神经发育障碍、58.3%的混合特异性发育障碍和55.6%的表达和接受语言障碍。
此外,粪肠球菌的给药对母体免疫激活的后代的重复行为和焦虑样行为没有产生任何影响。
总之,肠道细菌对宿主情绪和应激反应产生影响的分子和细胞机制将在未来进行研究。尽管在动物研究中微生物群对HPA轴有显著影响,但还需要更多的临床研究来支持使用益生菌缓解人类压力水平的概念。
✓
益生元是从食物中提取的不可消化成分,用于促进微生物(主要在胃肠道中)的生长。
合生元是将益生元和益生菌结合起来,对宿主产生协同作用。
先前的研究表明,益生元和合生元治疗都可以改变啮齿动物模型中的皮质酮水平。很少有研究调查益生元和压力暴露之间的相互作用及其对控制皮质酮水平的影响。
低聚果糖和低聚半乳糖
低聚果糖(FOS)和低聚半乳糖(GOS)治疗对成年小鼠产生抗焦虑和抗抑郁作用。
此外,GOS 和 FOS + GOS的组合可有效下调强迫游泳试验引起的急性应激诱发的皮质酮。有趣的是,FOS 和 GOS 处理中 Akkermansia、Bacteroides、Parabacteroides 的相对丰度有所增加,而脱硫弧菌、瘤胃球菌、Allobaculum、Turicibacter、乳酸杆菌、双歧杆菌的相对丰度降低。
然而,另外两项使用不同益生元化合物的研究并没有对不可避免的压力(GOS、聚葡萄糖和糖蛋白乳铁蛋白)或社会破坏压力(人乳低聚糖3 ‘唾液酰基乳糖或6 ‘唾液酰基乳糖)引起的皮质酮产生抑制作用。研究人员推测各种化合物、治疗持续时间和治疗开始时间都会影响益生元的效果。
低聚甘露糖
除了压力暴露之外,低聚甘露糖 (MOS) 的长期治疗降低了 5xFAD 转基因阿尔茨海默氏病小鼠模型血清中皮质酮和 CRH 的基线水平,但野生型小鼠没有这种情况。此外还发现 MOS 会增加血清和粪便中的丁酸盐水平,并与血清皮质酮呈负相关。然而,另一项研究结果表明,MOS 治疗降低了野生型 Whistar 大鼠成年期的血浆皮质酮水平。有趣的是,药物诱导的便秘大鼠模型表现出较高的 ACTH 和较低的皮质酮水平,而菊粉和低聚异麦芽糖可使其正常化。
其他益生菌和益生元联合治疗
益生元和益生菌的合生治疗很复杂,并且有多种组合。迄今为止,还没有研究使用相同的细菌菌株和益生元化合物配方来调节压力。
在暴露于慢性日常约束应激的大鼠中,将麦芽糊精L. paracasei DKGF1与仙人掌提取物相结合,以时间依赖的方式抑制约束应激诱导的皮质酮水平。
益生菌L.gasseri 505抑制UCMS诱导的皮质酮。添加柘树叶提取物不会对皮质酮产生额外影响。在急性应激中,在大鼠中,L. casei 54-2-33和菊粉的联合治疗可以降低加迷宫诱导的皮质酮升高。很少有研究采用合生元策略来缓解应激反应和皮质酮,这可能是因为缺乏关于益生菌和益生元的机制观点的基础。
✓
类固醇生成是在宿主体内将胆固醇转化为类固醇的生物合成过程。在糖皮质激素中,胆固醇通过几种关键酶(包括孕烯醇酮、黄体酮和脱氧皮质酮)经过几个步骤转化为皮质酮。然后皮质酮代谢为醛固酮。有趣的是,一些研究支持本地微生物直接调节宿主体内类固醇合成的假设。本节讨论从头细菌将胆固醇转化为类固醇的潜在候选细菌,这可能会干扰糖皮质激素类固醇的合成。
细菌中类固醇的生物合成——是直接影响类固醇激素水平的一种方式
在瘤胃球菌 (R.) gnavus、Bacteroides (B.) Acidifaciens 和 Clostridium (C.) scindens 培养物中发现孕烯醇酮治疗无氧条件下48小时可合成雄激素类固醇;研究人员使用液相色谱-串联质谱测量了类固醇途径中间体的水平。
在细菌条件培养基中检测到羟基孕烯醇酮、孕酮、脱氢表雄酮、睾酮。同样,相同的细菌菌株可以在体外将羟基孕烯醇酮代谢为黄体酮、脱氢表雄酮和睾酮。然而,R. gnavus 和 B. Acidifaciens 没有表现出任何胆固醇、皮质醇或醛固酮的代谢能力。
此外,孕烯醇酮和羟基孕烯醇酮在其他共生菌株中的处理,包括粪肠球菌、阴沟肠杆菌、肺炎克雷伯菌27、奇异变形杆菌、粘质沙雷氏菌、溶血葡萄球菌、大肠杆菌,产生了阴性结果,表明细菌在类固醇中间体代谢中的特异性。
类固醇激素代谢——可能是细菌影响宿主激素水平的另一种途径
从水甲虫的肠道中分离出两种芽孢杆菌菌株,HA-V6-3 和 HA-V6-11,并表明它们能够代谢孕烯醇酮。
其他证据表明,吲哚和粪臭素这两种肠道细菌衍生的色氨酸发酵代谢产物,可以抑制类固醇生成的限速酶CYP11A1,从而减少孕烯醇酮。
此外,睾酮缺乏与抑郁症状有关。最近发现从抑郁症患者中分离出的新金分枝杆菌(Mycobacterium neoaurum)可以将睾酮降解为雄烯二酮。
在新金分枝杆菌中鉴定出一个编码3β-羟基类固醇脱氢酶的基因,该基因可降解睾酮。将3β-羟基类固醇脱氢酶引入大肠杆菌中,产生3β-羟类固醇脱氢酶产生菌。产生3β-羟基类固醇脱氢酶的大肠杆菌在ABX小鼠中的定殖诱导了抑郁样行为。
最近进行的一项研究调查了给C57BL/6小鼠服用Thauera菌株GDN1的效果。GDN1是一种具有分解代谢睾酮能力的β变形菌。结果表明,GDN1菌株的给药导致血清雄激素水平显著降低,并在粪便提取物中检测到雄激素环切割代谢产物,这表明能够分解代谢雄激素的肠道细菌,可能调节宿主循环雄激素水平,并有可能作为益生菌用于高雄激素血症的替代治疗。
C. scindens细菌,通过侧链裂解将糖皮质激素皮质醇转化为雄激素
尽管没有研究表明特定细菌的能力可以直接影响皮质酮水平,但弗吉尼亚联邦大学的 Hylemon 实验室发现,C. scindens 是一种从人类粪便中分离出来的细菌,可以通过侧链裂解的机制将糖皮质激素皮质醇转化为雄激素。
C. scindens通过desD编码的钠依赖性皮质醇转运蛋白将皮质醇转运到细菌中。然后,皮质醇可以通过甾体-17,20-脱模酶(一种由desAB编码的假定转酮醇酶)代谢为11β-OHA(11β-氢-氰基甾-4-烯-3,17-二酮)。11βOHA可以通过ABC转运蛋白被泵出细胞。目前尚不清楚是否有细菌具有将皮质酮转化为其他类固醇的相似机制。
微生物参与皮质酮合成的新机制
Hylemon实验室的另一项研究在Eggerthella lenta(以前称为真杆菌)的细胞提取物中发现了一种皮质类固醇21-羟化酶。有趣的是,21羟化酶使用脱氧皮质酮、脱氧皮质醇、脱氢皮质酮和皮质酮作为底物。这可能是哺乳动物中微生物将类固醇转化为皮质酮的另一种机制。
然而,在压力暴露后的啮齿类动物中均未报告C.scindens和Eggertella lenta,也没有发现它们在微生物组缺乏的啮齿动物的肠道中定植。
从皮质酮的生物合成和代谢角度来看,一些细菌可以促进皮质酮前体,而另一些细菌则有相反的效果。因此,确定一种单一的途径来阐明在无菌和ABX(类似的广谱抗生素药物组合)治疗的小鼠中发现的高皮质酮仍然具有挑战性。该领域在肠道中面临着一种高度复杂的情况,这种情况会调节应激激素和应激诱导的行为异常。
✓
与压力相关的疾病,例如肠易激综合症(IBS),通常涉及微生物失调。 IBS 是一种胃肠道并发症,其特征是腹部不适、排便方式改变以及伴随的焦虑等症状,影响着 5-10% 的人口。尽管进行了广泛的研究,IBS 的确切病因仍然难以捉摸,公认的危险因素包括遗传、饮食、心理压力和肠道微生物组组成。
IBS患者中微生物代谢物的改变
研究表明,与健康对照相比,IBS 患者的 α 多样性降低,并且 21 种细菌种类的相对丰度存在显著差异。 IBS 亚型在肠道微生物群衍生的代谢物中表现出明显的改变:
微生物群在IBS患者中的作用
研究表明,IBS-D 中的特定细菌,如瘤胃球菌Ruminococcus gnavus,可以通过产生苯乙胺和色胺来刺激血清素生物合成,从而加速肠道蠕动。
研究发现,在一些 IBS 患者中发现的产气克雷伯菌(Klebsiella aerogenes)可增强组胺的产生,通过组胺 4 受体信号传导导致内脏痛觉过敏。
值得注意的是,可以通过人类 IBS 患者的粪便微生物群移植 (FMT) 到 GF 接受者来建立 IBS 动物模型。这种方法有效地复制了在人类 IBS 患者中的胃肠道和焦虑症状。
IBS 患者的应激反应激素水平
压力暴露是发生 IBS 的已知危险因素,通常影响肠道蠕动和 HPA 轴。 IBS 患者的应激反应激素水平与健康人不同。
研究表明,急性精神压力会导致 IBS 患者血浆 CRH 和 ACTH 显著升高。此外,Dinan组显示,CRH输注后,IBS患者的ACTH和皮质醇释放增加。
结肠扩张(CRD)是一种检测内脏敏感性的方法,在IBS动物模型中,导致PVN CRH神经元中c-Fos表达升高,血浆CRH、ACTH和皮质酮水平升高。
总之,这些发现表明,与健康受试者相比,患有IBS的个体表现出应激激素分泌增加和微生物群失调。
✓
应对压力是动物面对可能对身体有害的危及生命事件的重要策略。压力失调与情感性疾病密切相关。新冠肺炎大流行使压力相关疾病的全球流行率急剧上升,这对社会产生了深远影响。
最近的研究表明,肠道微生物群不仅出现在压力暴露的背景下,而且还起着“活性调节剂”的作用,调节神经和内分泌系统。这也就是我们开头提到的“胃里有蝴蝶”的扑腾感源于肠道微生物。
肠道微生物直接和局部调节类固醇生成,可能改变应激激素水平。然后,应激激素信号可以通过确定的途径、肾上腺外类固醇生成、自主神经系统和各种细菌成分传播到大脑。最终,大脑接收来自微生物的信息,并对PVN和其他大脑区域做出充分反应。
此外,大脑决定的应对和适应机制可以改变基于行为和内分泌功能的输出。然后,微生物可以进一步适应宿主在压力下的生理机能。这种从肠道微生物群开始的控制环路是基于目前对肠道微生物和压力之间相互作用的理解。
肠道微生物调节行为的分子和细胞机制、途径和回路在很大程度上尚未完全探索。识别导致和影响应激反应的关键细菌和细菌相关因素,将有利于使用基于微生物组的疗法进行替代医学创新。
随着对微生物与宿主交互作用机制的深入研究,肠-脑轴在心理疾病发生和治疗中的重要性将越来越受到重视。肠道菌群将成为预防和干预这类疾病的新靶点。
主要参考文献
Lai TT, Liou CW, Tsai YH, Lin YY, Wu WL. Butterflies in the gut: the interplay between intestinal microbiota and stress. J Biomed Sci. 2023 Nov 28;30(1):92.
Ramamoorthy S, Cidlowski JA. Corticosteroids: Mechanisms of Action in Health and Disease. Rheum Dis Clin North Am. 2016 Feb;42(1):15-31, vii.
Huang ST, Wu K, Guo MM, Shao S, Hua R, Zhang YM. Glutamatergic and GABAergic anteroventral BNST projections to PVN CRH neurons regulate maternal separation-induced visceral pain. Neuropsychopharmacology. 2023 Nov;48(12):1778-1788.
Avery S N, Clauss J A, Blackford J U. The human BNST: functional role in anxiety and addiction[J]. Neuropsychopharmacology, 2016, 41(1): 126-141.
谷禾健康
由于肉类和肉制品含有丰富的脂质和蛋白质,因此易于发生氧化反应。脂质氧化会产生一系列氧化衍生物,主要影响食物的颜色和风味,同时也会导致肌肉蛋白质的功能和稳定性丧失。同样,蛋白质容易被活性氧化物质(ROS)和氧化应激的次级副产物氧化。蛋白质氧化是一个会导致构象和结构改变、聚集和断裂的过程,从而损害蛋白质的功能特性,进而影响肉品的质量。
脂质氧化-蛋白质氧化在肉类中是密不可分的,因为脂质氧化可能会诱发蛋白质氧化。这些积累的氧化蛋白质和脂质,包括它们的次级产物,被认为在食用肉制品和随后的消化时对人类构成健康风险。因此,控制肉类和肉制品中的蛋白质氧化非常重要。
已经进行了许多消化率试验和相关研究,以揭示与肉类蛋白质相关的分子变化和潜在风险。蛋白质消化是一种复杂的过程,涉及消化酶对膳食蛋白质的集体作用。这个过程受多种因素的影响,包括蛋白质的饮食来源、消化酶的活性、胃肠道菌群,pH值、肠道蠕动和内源性分泌物等。通过动物模型的研究,已经证明摄入过多的加工肉类可能会促进与非酒精性脂肪肝和肥胖相关的蛋白水解细菌的增殖。
另外,大量摄入红肉后会刺激结肠中的氧化反应。过多摄入肉类氧化产物可能会导致肠道菌群失调,损害结肠的代谢能力,并对结肠粘膜产生有害影响。
前不久南京农业大学食品科技学院李春保教授等人发表了题为“Molecular Changes of Meat Proteins During Processing and Their Impact on Quality and Nutritional Values”的综述论文。重点介绍了肉类蛋白质的分子变化、对新鲜肉类和加工肉类营养价值的影响、肉类蛋白质的消化和吸收、与高肉类摄入量相关的健康风险以及为改善这些情况而采取的预防策略。希望能够对生活中肉类的食用有一定帮助。

编辑
肉质通常会受到理化和代谢变化的影响。这些变化主要包括pH值下降、肌原纤维蛋白降解、蛋白质氧化和蛋白质翻译后修饰。
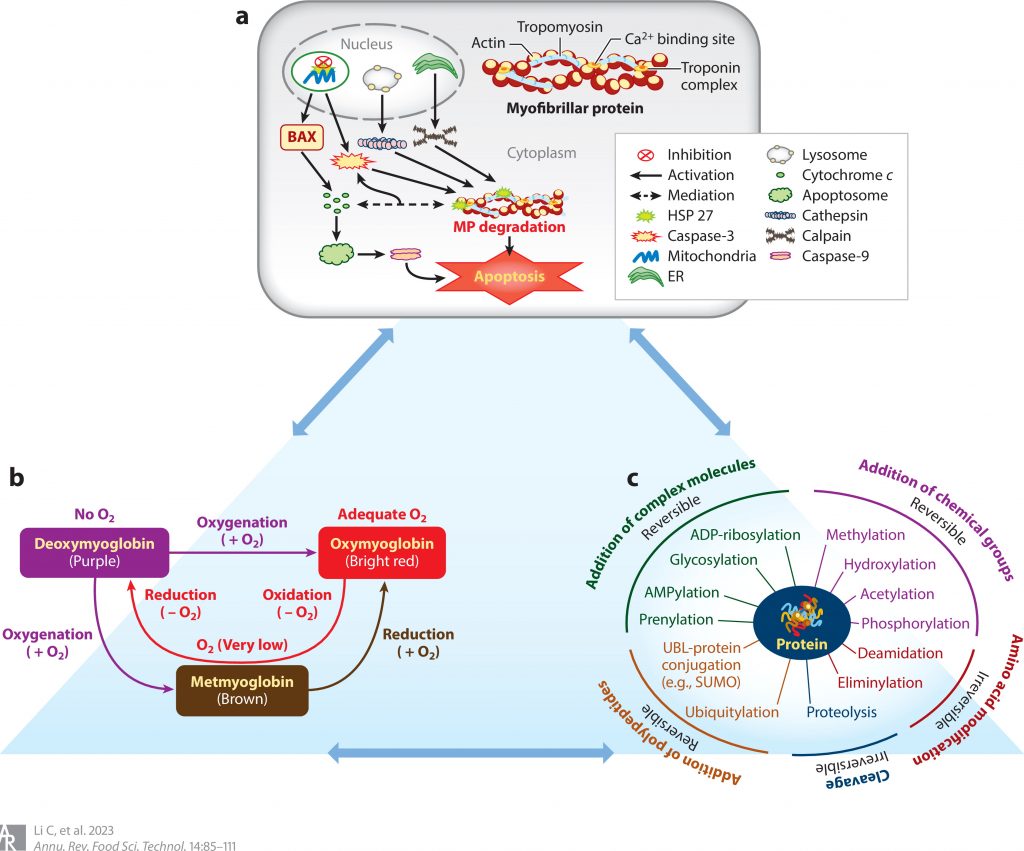
Li C,et al.Annu Rev Food Sci Technol.2023
肉的颜色是一个不可或缺的质量属性,也是消费者的第一判断点,其变化与购买的可能性密切相关。
鲜肉颜色通常由着色剂的浓度和所需颜色在储存期间或零售展示期间的稳定性为特征。色调和色度由肌红蛋白主导,其变化取决于其生化状态,特别是氧化或还原程度。
注:肌红蛋白是肌肉中主要的携氧血红素蛋白,而血红蛋白则在血液中输送氧气。肌红蛋白比血红蛋白具有更高的氧亲和力。
•在空气中暴露不同的时间导致产生不同颜色
肌红蛋白与空气中的氧气接触后,随着时间的推移,新鲜肉呈紫红色;暴露在空气中30~40min后,颜色变为鲜红色;最后肉类自身存在的耗氧酶会消耗掉渗入肉中的氧气使肉质产生褐变。
颜色变化的具体机制
最近,已经进行了生化研究来阐明肉的颜色机制。利用从深色牛肉中分离的线粒体(肌肉pH>6)注意到,深色牛肉的电子损失高于对照(即从正常牛肉肌肉中分离出的线粒体,pH=5.6)。
这表明线粒体较多的肌肉,如腰大肌,可以产生活性氧并发生氧化变化。在大腰肌中检测到高丰度的次黄嘌呤和肌苷,而在腰最长肌中观察到腺苷、肌肽和l-组氨酸,表明不同肌肉类型之间的嘌呤代谢率不同。
另一项研究同样报道了深色牛肉中较高的线粒体蛋白质和DNA含量。这一发现也证实了最近的一项蛋白质组学研究,该研究表明,与线粒体电子传输和糖原分解相关的蛋白质的变化分别促进了高氧摄入和pH值的增加,从而抑制了深色肉中的肌红蛋白氧化。
肌原纤维蛋白占肌肉总蛋白的60-70%,肌原纤维蛋白的分子内和分子间相互作用,例如氢键和疏水性、离子性和范德华相互作用,极大地影响肉的质地、结构和品质。
胶原蛋白浓度和溶解度、肉收缩的程度以及肌原纤维蛋白在死后老化过程中的蛋白水解变化是与肉嫩度相关的整体因素。老化最初提供了对机械应力的缓冲。它通过肌肉组织的酶促结构降解和随后的肌肉细胞肿胀来冷冻。肌纤维中的大部分水分被限制在肌原纤维中,肌原纤维蛋白降解可能与水分减少有关。
最近研究报道,溶酶体Fe2+促进脂质过氧化和线粒体肿胀,导致线粒体功能障碍,随后促进细胞色素c氧化和线粒体Ca2+积累,从而诱导肌原纤维蛋白变性。
线粒体是最初因死后变化而受损的主要细胞器,并且是进一步细胞反应和最终死后肉嫩化不可或缺的一部分。活性氧在死后衰老过程中不可避免地产生,并通过多不饱和脂肪酸的过氧化进一步侵入线粒体膜,导致线粒体损伤。
此外,离子强度和pH值可能会导致蛋白质结构发生改变,使疏水残基暴露于表面并影响表面疏水性。研究表明,在冷冻过程中,未冷冻水中的pH值降低,离子强度增加,导致肌球蛋白丝的内部结构破坏,并有助于其构象变化以及随后的溶解和变性。
氧气损失导致死后早期从有氧代谢转变为无氧代谢。伴随着这样的过程,会发生以下复杂的变化。
磷酸化是一种蛋白质翻译后修饰,在死后肌肉质量属性中发挥着不可或缺的作用。特别是,蛋白质磷酸化是苏氨酸、丝氨酸和酪氨酸残基的主要蛋白质翻译后修饰,调节信号转导、代谢和其他重要的生物过程,例如分化和增殖。它通过调节肌动球蛋白解离、蛋白质变性和宰后μ-钙蛋白酶活性来影响肉质。
先前的研究表明,肌浆蛋白和肌原纤维蛋白的磷酸化状态会在死后发生变化,这种变化与肉质有关。在一项磷酸蛋白质组学研究报告中称,季节也会导致肉质变化,包括pH值和保水能力下降。季节转换期间的温度变化可能会通过某些伴侣蛋白的过度表达导致猪的冷应激或热应激,从而导致肉质苍白、柔软、渗出性缺陷。
酶在肉类的生化变化中至关重要,例如碳水化合物降解、蛋白水解、氨基酸降解反应(脱氨、转氨基、脱羧)、Strecker 降解、美拉德反应、脂肪分解和 脂质氧化。在死后老化过程中,通过内源性蛋白水解作用使肉中的细胞骨架肌原纤维蛋白变性,可显著提高质量,例如多汁性、风味或嫩度。
钙蛋白酶和组织蛋白酶是参与死后蛋白水解的酶系统。钙蛋白酶系统的两种μ/m同工型在死后老化过程中均被激活,并且通过降解结蛋白和肌钙蛋白-T对肉嫩化至关重要。
另一方面,随着死后肌肉pH值降低,溶酶体膜减弱,组织蛋白酶从溶酶体中释放出来,使它们能够水解肌原纤维蛋白。因此,与死后肌肉储存过程中发生的肌原纤维蛋白相比,组织蛋白酶处理的肌原纤维蛋白具有不同的变性模式。
•热休克蛋白可以抑制肉的老化
热休克蛋白(HSP)家族以其在细胞保护中的重要性而闻名。高丰度的热休克蛋白是肌肉抗细胞凋亡不可或缺的一部分,并可能抑制肉的老化,影响肉的嫩度。
据报道,小热休克蛋白可以保护死后肌肉中的细胞骨架结构,例如肌联蛋白、肌动蛋白和肌钙蛋白。它们可以模拟肌动蛋白单体并与肌动蛋白-肌动蛋白结合位点结合,有效阻碍肌动蛋白聚合。μ-钙蛋白酶的蛋白水解活性通过小热休克蛋白的抑制调节而受到最终肉类pH值的影响。
HSP27是一种分子伴侣蛋白,大量存在于骨骼肌中,与肉品质相关。研究发现HSP27似乎抑制蛋白水解的启动,但随着肉在屠宰后7至14天的老化过程中,HSP27的存在有助于肉的嫩化。HSP27可能通过与酶、底物或两者的相互作用来调节蛋白水解活性。
在肉类加工过程中,根据叠加的时间温度条件和工艺流程,不同程度地发生多种结构变化。肌肉细胞在机械变化过程中被氧气降解和包裹,从而增加了脂质氧化-蛋白质氧化的可能性。
热处理会导致主要蛋白质成分发生严重变化,这是由蛋白质变性、氨基酸氧化和聚集过程引起的。
谷禾将不同加工方法及其对肉品质影响的优缺点简单总结在了下表:

Li C,et al.Annu Rev Food Sci Technol.2023
1
加热
加热引起构象变化,称为蛋白质变性。它还诱导蛋白质之间相互作用,导致蛋白质聚集。熟肉中蛋白质聚集体的形成主要影响产品的技术和营养特性。
粉碎产品和非粉碎产品的差异很大。例如,与煮熟的火腿相比,乳化香肠含有更多的不溶性蛋白质残留物。胶原蛋白的存在意味着高分子量蛋白质聚集体,可能是热处理和不同工艺过程的结果。这些聚集体的特征是肌原纤维与肌浆的比例很高,这表明肌原纤维蛋白,特别是肌动蛋白,在构建热诱导的超分子蛋白质组装过程中相互作用。
在研究烹饪对肉类蛋白质的影响时,观察到氨基酸氧化变化呈上升趋势,特别是在可溶性胶原蛋白中,而不溶性部分中的肌球蛋白最容易受到其他热诱导的修饰(例如美拉德反应)的影响。
注:最近发现范德华力和氢键产生肌球蛋白-醛结合相互作用,从而为维持/控制肉味的机制提供了新的见解。
•胶原蛋白在长时间加热后有更好的提取性
一项蛋白质组学研究表明,干燥加热有助于一些三维结构显著变化的肉类蛋白质的聚集,例如肌酸激酶M型和肌球蛋白重链。
尽管在尿素-硫脲溶液中检测到总蛋白质可提取性显著损失,但与其他蛋白质相比,骨骼肌中的主要胶原蛋白(I型和III型)在长时间烹饪后表现出相对更好的可提取性。
•低温加热可能拥有更好的抗氧化特性
据报道,在较低温度下热诱导的I型胶原蛋白部分解折叠或氧化可能会暴露出更多胃蛋白酶消化的活性位点,而过热会导致蛋白质聚集、聚合、交联和氧化,从而诱导对酶降解的抵抗。
真空低温烹调12小时增加了抗氧化活性肽的数量,这可以显著减少脂质氧化和蛋白质氧化的发生。这表明具有潜在抗氧化特性和生物活性、可穿透细胞的肽可能在低温和短时间下产生。
2
烘干
干肉和肉制品通常通过热风干燥技术来实现。一般来说,肉类蛋白质的连续和密集加工会因必需氨基酸的损失和消化率受损而导致营养变化。
•烘干会导致蛋白质消化率下降
为了揭示脂质氧化、肌原纤维氧化和蛋白质消化率的变化,研究发现风干牦牛肉蛋白质消化率的下降归因于氧化诱导的蛋白质交联和羰基化,影响了底物与胃蛋白酶的结合。
•冷干燥可以减少氧化,延长保质期
另外,据报道,冷干燥可以通过降低温度来减少热敏食品的快速氧化过程,从而延长保质期。冷风干鱼的蛋白质降解和脂肪酸氧化下降。
注:在4°C下冷干燥的凤尾鱼和鳟鱼样品比在恒定气流下在10°C、15°C和20°C下干燥的样品具有更高的功效。
3
加盐
盐是加工产品中不可或缺的成分。它促进肉类蛋白质的溶解,并充当肉类和脂肪之间的粘合剂,改善质地、嫩度、香气和适口性。
感知到的咸味主要归因于Na+阳离子,风味强度取决于盐含量。盐水平在肌动球蛋白的氧化、结构和消化中发挥着重要作用,表明对肉类营养和蛋白质消化率有很大影响。
•施用盐影响了蛋白质的消化率
由于肌红蛋白的刚性和消化效率低,氯化钠处理降低了蛋白质结合能力,导致肌红蛋白被胃蛋白酶快速消化。尽管如此,过量的氯化钠会损害蛋白质功能,因为肌原纤维蛋白具有高度盐溶性。
最近有报道称,氯化钠处理会改变血红素结构和肌红蛋白疏水腔,降低蛋白质消化率。因此,氯化钠减少对蛋白质氧化的影响各不相同,这与氯离子与蛋白质残留、蛋白水解和肌原纤维状况之间的关联有关。
•盐类物质可能改变了蛋白质的结构
人们对各种氯化物盐如氯化钙、氯化钾和氯化镁进行了研究,以确定它们作为肉制品中氯化钠替代品的效力。因此,观察到一价(钾)和二价阳离子(镁和钙)在凝胶化过程中增强了肉蛋白的功能。
为了评估用KCl、MgCl2或CaCl2部分替代NaCl对肌原纤维蛋白的影响,证明NaCl能够展开肌原纤维蛋白结构并增强凝胶质量,而25%KCl增加肌原纤维蛋白疏水性和粒径、二硫化物和羰基含量,在类似离子下比其他盐组合更好地增强凝胶结构和凝胶强度力量。尽管MgCl2或CaCl2导致蛋白质不溶和聚合,但前者极大地影响凝胶性质。
4
包装
人们开发了各种包装技术,以减少氧化过程并提高肉质。
•低氧浓度抑制蛋白质氧化,利于保持肉的嫩度
数据发现低氧浓度(10%O2/90%N2)抑制蛋白质氧化,而高氧包装增加了肉的损失并降低了肉的嫩度。
在高氧浓度(80%O2/20%CO2)包装牛肉中,揭示了高分子量蛋白质聚合物的生成,表明高氧化和交联导致高氧包装肉中的肌球蛋白聚合。
•富含天然抗氧化性的物质对健康有益
通过将食品、保鲜和包装系统纳入可食用、可生物降解和防潮薄膜中,可食用薄膜或涂层显著改善了包装。这种包装可以防止颜色退化、氧化变化和异味,延长保质期,并赋予肉类及其衍生物功能。
据报道,富含天然抗氧化剂的提取物、多糖和精油的不同涂层可增强膜的机械性能,并抑制肉制品中高铁肌红蛋白的形成和脂质氧化–蛋白质氧化过程的形成。
注:先前关于天然抗氧化剂在肉类工业中应用的研究也进行了综述。这些生物活性物质一旦摄入,也会对消费者的健康有益。
5
其他处理方法
最近,消费者的注意力已逐渐从营养转向对身体产生积极生理反应的食品。超声波处理、高压处理和脉冲电场(PEF)等创新工具已被证明可以增强肉类和肉制品的基本生化和功能特性。
•高压处理可以降低烹饪损失,改善消化
人们发现高压处理可以通过形成丝状网状网络(甚至在肌肉纤维内)来改善肉糊的质量,从而降低烹饪损失。
在一项蛋白质组学研究中,揭示高压处理加速蛋白水解,表明肌动蛋白与肌球蛋白不同,存在差异性变性。他们还证实,高压处理对直链醛(己醛、壬醛、庚醛和戊醛)的形成有积极影响,从而改善干腌火腿的味道和香气。高压处理和脉冲电场也被证明对消化过程中肉蛋白的酶水解产生积极影响。
•超声辅助可以改善肉的质地和结构
最近还发现,超声辅助的表没食子儿茶素没食子酸酯共价反应基团表现出更高的消化率,这与消化蛋白酶的可及性和减少肌原纤维蛋白聚集有关。证明集成超声波和低温短时加热(40kHz,0.2W/cm2,55°C,15分钟)协同促进必需肉类蛋白酶(组织蛋白酶 B、钙蛋白酶和总蛋白酶)的失活。这显著减少了肉类蛋白质的降解,改善了肉的质地和微观结构。
同样,液相色谱与串联质谱联用(LC-MS/MS)的结果表明,超声处理可促进小分子量肽的生成,这可能与线粒体能量代谢的上升趋势有关。
肉类是膳食蛋白质的重要来源,是具有良好平衡性和高生物利用度的必需氨基酸。然而,蛋白质氧化和肉类结构的变化会影响蛋白质的水解速率,从而影响蛋白质的生物利用度。
•烹饪程度影响蛋白酶的敏感性
虽然轻度蛋白质氧化可能诱导部分解折叠,促进蛋白水解酶与蛋白质底物的结合,但严重的蛋白质氧化条件会改变蛋白质与蛋白水解酶的结合位点,并通过聚合使蛋白质结构变得紧凑,从而降低蛋白酶敏感性。
一个例子如美洲鸵鸟肉,烹饪抑制了肌原纤维蛋白对胃蛋白酶活性的敏感性。烹饪后胰酶的蛋白水解率增加支持了蛋白质聚集在肉类蛋白质营养特性中的重要性。
•肉的类型影响蛋白质成分和消化敏感性
肉的营养质量取决于多种因素,从动物和肌肉类型到成熟度和烹饪条件。在蛋白质组研究中,证明猪肉中的股二头肌对消化的敏感性最高,相互作用分析表明差异蛋白主要与糖酵解和肌肉收缩有关。
因此,蛋白质成分和消化敏感性的变化可能归因于肉质类型。然而,营养质量还必须考虑肉的脂肪酸组成和可能产生有害物质的过氧化过程。
加工过程中生肉和预煮肉制品中蛋白质氧化衍生物含量较高可能归因于高脂肪-蛋白质比率、低蛋白质水平或强烈的切碎和烹饪方法。烹饪过程中蛋白质氧化的发生以及进一步消化过程中氧化序列的释放导致生物活性变化,主要来自胶原蛋白和肌联蛋白检测到的肽以及消化过程中新产生的肽。最近证明,蛋白质来源和脂肪含量影响食品基质的流变特性和微观状态,进一步影响蛋白质水解程度和消化率。
•低消化率会降低蛋白质的生物利用度
还有研究证明肌动球蛋白在100°C下持续加热会导致二硫键的形成、聚集和残基氧化,从而通过破坏部分切割位点导致消化率降低,很大程度上改变了蛋白质消化物中的肽组成。肌红蛋白在胰酶和胃蛋白酶消化中的水解程度也相对较低。
低消化效率可能会抑制蛋白质的生物利用度及其在盲肠和结肠中的积累,蛋白质或其片段在盲肠和结肠中由肠道微生物群发酵。
评估了胃肠道消化对肉和肉制品产生的生物活性肽的影响。胃肠道中生物活性肽的模拟研究表明,肉蛋白是抗二肽基肽酶活性、血管紧张素转换酶抑制和一定程度抗氧化活性的来源。
因此,为了使肉释放生物活性肽并表现出生理效应,肽必须完整地通过胃肠道。
饮食成分和饮食习惯对肠道健康很重要,包括肠道微生物成分和平衡。在消化和吸收过程中,胃肠道从食物中提取营养物质,以提供基本营养和健康。虽然有些食物富含抗炎生物活性化合物,但其他食物则含有促炎化合物。
定期摄入富含抗炎生物活性化合物的食物有益于肠道微生物的流行,将食物成分降解为代谢物以维持体内平衡。相比之下,摄入富含促炎化合物的饮食可能导致生态失调,产生可能对健康构成风险的致病代谢产物。
过量摄入高脂肪肉类带来的不利影响
Li C,et al.Annu Rev Food Sci Technol.2023
•合理的肉类摄入有助于平衡肠道微生物以及抗氧化作用
在均衡饮食中,摄入肉类蛋白质已被证明可以对肠道微生物成分、能量代谢和抗氧化产生积极影响。报道称,在长期食用蛋白质来源(酪蛋白、牛肉、鸡肉和大豆等)的过程中,肉类蛋白质保持了更平衡的肠道微生物成分,并降低了肠道微生物对宿主的抗原负荷和炎症反应。
有研究称,与大豆相比,肉类蛋白质有利于新陈代谢和生长。最近声称,与牛肉、猪肉和大豆蛋白相比,鸡肉和鱼肉蛋白显著诱导肝脏抗氧化应激。因此,在推荐剂量下,肉类蛋白质可以促进甘油三酯分解和胆固醇降解,并将能量平衡维持在健康水平。
不同肉类摄入对健康的影响


Li C,et al.Annu Rev Food Sci Technol.2023
•膳食酪蛋白有助于减少肥胖
此外,由于氧化应激是肥胖、糖尿病等慢性疾病的主要推动因素,在不损害营养价值的情况下合理摄入肉类可以改善健康风险。
几项研究证明了积极的发现。在一项研究中与膳食鸡肉相比,膳食酪蛋白增加了实验动物盲肠中乳酸乳球菌(Lactococcus lactis)的丰度,并上调了与肥胖抑制相关的基因表达。
一致地,在喂养小鼠的膳食蛋白质来源(大豆、牛肉、去皮鸡肉、酪蛋白、鳕鱼片或猪肉)中,与其他饮食相比,酪蛋白饮食大大减少了脂肪量并防止了肥胖的发展。
富含酪蛋白的食物:酪蛋白是一种乳清蛋白,主要存在于乳制品中。例如牛奶、酸奶、奶酪、奶粉,以及一些鱼类、豆类。
•天然益生元通过肠道菌群有利于健康
在喂食富含菊粉的加工猪肉产品(发酵香肠和煮熟的火腿)的两种结直肠癌动物模型中,息肉减少,抗炎和纤维发酵微生物群呈上升趋势。
值得注意的是,与淀粉不同,菊粉等果聚糖在到达结肠之前不会在人类胃肠道中发生改变,在结肠中它们被双歧杆菌、乳酸杆菌和其他短链脂肪酸产生物种等益生菌用作能量和碳源。
富含角豆果提取物的肉制品还被发现可以在晚期2型糖尿病模型中提供预防和治疗作用,例如诱导更高的肠道微生物丰富度、增强结肠屏障完整性、促进足够的短链脂肪酸水平以及在远端结肠粘膜。
丁酸盐是结肠细胞的首选能量来源,具有促进结肠健康和抗肿瘤的功效,支持粘膜完整性,并通过其对表观遗传调节、免疫和基因表达的影响来预防炎症和癌变。
需要注意的是,过量地摄入肉类也不利于人体健康。
•高肉高脂肪饮食会损害神经递质平衡
报道称,高猪肉或高鸡肉饮食(40%或以上)和高脂肪含量会损害谷氨酸能系统和神经递质平衡,并对海马谷氨酸能系统和肠胶质细胞之间的关联产生巨大影响。
同样,高脂饮食中过量的肉类蛋白摄入可能会促进代谢紊乱、全身炎症、中枢神经系统功能的改变,并最终导致神经退行性疾病的发展。
•高肉类饮食影响肠道微生物组成
高脂饮食中的蛋白质显著影响肠道微生物组成、肝脏代谢特征、肠道炎症基因表达和血清内毒素水平,其中许多代谢物影响血清内毒素、炎症和肠道通透性。
研究表明,肠道炎症通过高脂饮食引起的肠道血管屏障功能障碍诱导肝损伤,强调了肠道微生物、肠道屏障和非酒精性脂肪性肝病发展的饮食之间的关系。
特定的肠道微生物可能通过调节宿主消化蛋白的分泌来影响蛋白质的消化和吸收,特别是在长期食用肉类蛋白之后。研究表明,与替代品相比,高膳食肉类蛋白会降低肝脏中吲哚和粪臭素代谢酶的活性。
还报道了饮食蛋白质喂养的大鼠盲肠微生物群组成的快速变化。说明了肉类摄入量与肠道微生物之间的关联,表明过量肉类消费会增加心血管疾病风险和动脉硬化。
•高肉饮食会导致糖代谢紊乱
最近评估了不同氧化损伤程度的熟猪肉对小鼠葡萄糖代谢的影响,并得出结论,摄入高氧化损伤猪肉饮食会损害葡萄糖耐受性,并导致低胰岛素血症和高血糖,表明葡萄糖代谢紊乱。
•高肉饮食影响胆汁酸的分泌与合成
肠道微生物还会在肉类和其他膳食来源中产生三甲胺。三甲胺被肝脏含黄素单加氧酶氧化为氧化三甲胺,可能抑制胆汁酸的合成和分泌,还会促进动脉粥样硬化。
胆汁酸对于宿主改变肠道微生物组成至关重要,因为它与左旋肉碱有很强的相互作用,次级胆汁酸(如石胆酸和脱氧胆酸)的升高归因于摄入高脂饮食,这可能会引发结直肠癌风险。
研究发现肉类诱导的氧化三甲胺形成受到肝脏基因表达水平变化以外的机制的调节,可能涉及肠道微生物改变。重要的是,该研究表明,将富含红肉的饮食改为白肉或非肉类蛋白质来源(但保持相同的卡路里和膳食蛋白质比例)会导致氧化三甲胺水平在几周内大幅下降。
研究了膳食蛋白质来源与血浆氧化三甲胺水平的相互作用,以及通过饮食改变降低氧化三甲胺水平所需的时间,强调除了数量之外,饮食成分(与蛋白质来源有关,但与饱和脂肪含量无关)影响氧化三甲胺的整体代谢。
推荐剂量的肉类蛋白质可以增强胆固醇降解和甘油三酯分解,并保持能量合成处于健康状态。
以下几点可以使肉类在保存和加工过程中保持较好的营养价值和肉质:
采用低温保存,控制温度在0-4°C范围内,可以减缓肌红蛋白的氧化程度,保持肉类颜色。
选择肌肉类型含有较高线粒体的肉类,如鸡肉,可以减少肌红蛋白的氧化程度。
适度使用盐可以改善肉质,但不要过量,以免损害蛋白质。
避免高温长时间加工,如不必要的高温煮熟,可以防止蛋白质变性和聚集。
选择加工方法温和的肉制品,如熏肉比烤肉更易保留营养。
适量食用肉类,可以促进肠道微生物平衡,但过量可能影响健康。
食用肉类时搭配蔬菜水果等食物,可以提供抗氧化营养成分,共同保护肉类免受氧化损害。
总体来说,低温保存、温和加工和适量食用,可以最大限度地保留肉类的营养价值和肉质质量。
主要参考文献:
Li C, Bassey AP, Zhou G. Molecular Changes of Meat Proteins During Processing and Their Impact on Quality and Nutritional Values. Annu Rev Food Sci Technol. 2023 Mar 27;14:85-111.
谷禾健康
支原体:小而隐蔽
支原体(Mycoplasmas)是一种特殊且独特的细菌,是地球上已知的最小的自由生活的生物体。由于其体积较小,早些时候被误解为病毒。它小到用普通显微镜无法定位。事实上,大约 4000 个支原体可以轻松容纳在红细胞内。
支原体既作为膜表面寄生虫存在于细胞外,也存在于细胞内,作为“沉默的寄生虫”成为细胞内的居民。
支原体与其他细菌之间的主要区别在于:
当我们使用抗生素治疗某些细菌感染时,是通过破坏细菌细胞的外壁来起作用的。由于支原体没有外壁,所以大多数抗生素如青霉素等对它不起作用。因此支原体是小且隐形的病原体。
支原体首次被描述于 100 年前。迄今为止,已鉴定出 210 多种广泛分布于人类、动物、昆虫和植物中的物种。其中23种可以感染人类;一般免疫系统健康的人不会受到支原体细菌的影响,但免疫力低下的人很容易被感染,并可能出现严重的健康问题。支原体可以通过飞沫感染、昆虫叮咬、性接触、口腔摄入、吸入以及开放性伤口转移到另一个身体。
主要致病支原体包括:
• 肺炎支原体(Mycoplasma pneumoniae)
• 生殖支原体(Mycoplasma genitalium)
• 发酵支原体(Mycoplasma fermentation)
• 人型支原体(Mycoplasma hominis)
• 穿透支原体(Mycoplasma penetrans)
• 梨支原体(Mycoplasma pirum)
• 解脲支原体(Ureaplasma urealyticum)
这些支原体被确定与人类和动物的呼吸道和泌尿生殖系统疾病有关。
支原体是操纵和战胜宿主免疫系统的大师。它的一半基因组成专门用于这一独特目的。虽然它几乎没有能力造成直接伤害,但它可以利用宿主的免疫功能来发挥其优势:支原体产生慢性低度炎症并从体内窃取重要的营养物质。
事实上,这种隐形微生物生存所需的一切——维生素、矿物质、脂肪、碳水化合物和氨基酸,包括胆固醇、氨基酸、脂肪酸都要从宿主身上获取。它们尤其在富含胆固醇和精氨酸的环境中茁壮成长。支原体为什么大部分存在于呼吸道粘膜中,因为它们需要胆固醇来维持细胞膜的功能和生长,并且呼吸道的支气管中含有丰富的胆固醇。
广泛感染的很大程度上可能受宿主免疫功能状态的影响。如果免疫功能最佳,初次感染后微生物就会被遏制,不会发生长期危害。大约 30-70% 的人携带至少一种支原体,但没有症状。
截止2023年6月柳叶刀在全球范围内的统计显示,在COVID-19之前,肺炎支原体是呼吸道感染的常见原因,通过直接测试方法测得,2017年至2020年全球发病率为8.61%(Lancet Microbe. 2023 Oct;4(10):e763)。
然而,针对 COVID-19 的非药物干预措施大大降低了肺炎支原体的传播。短期内,2020-2021 年间,发病率下降至 1.69%。当时还观察到其他病原体的传播率较低。
然而,柳叶刀刚刚12月又发表统计结果表明:肺炎支原体:COVID-19 大流行限制后延迟重新出现。文章指出:
2023年4月1日至9月30日(6个月期间),前瞻性监测数据是从欧洲、亚洲、美洲和大洋洲这四个联合国区域的 24 个国家的 45 个地点获得的。在所有四个联合国区域均通过 PCR 检测到肺炎支原体。通过 PCR 检测,6 个月内肺炎支原体的平均发病率为4.12%,较COVID-19期间显著上升,推测可能出先新的亚型。也表明该病原菌容易逃脱免疫系统。
与病毒不同,支原体可以在组织液(血液、关节液、心脏、胸腔和脊髓液)中生长,并且可以在任何活组织细胞内生长而不杀死细胞,就像大多数正常细菌和病毒一样。
实际上,大多数支原体物种都不是正常菌群,它们只是在等待立足的机会。如果免疫功能由于某种原因而下降,慢性、全身性感染就可能发生。有些支原体种类还具有完全逃避免疫系统的独特能力。
一旦它们附着在体内的宿主细胞上,它们独特的血浆和蛋白质涂层就可以模仿宿主细胞的细胞壁,并且免疫系统无法将支原体与人体自身的宿主细胞区分开来。然后慢慢的,支原体开始窃取重要的营养物质,并引起一系列与最初感染无关的症状。支原体等隐形微生物对组织的普遍破坏会加速衰老过程,并且可能是少数慢性退行性疾病的主要因素。
据《国际免疫学评论》和《英国医学从业者杂志》等出版物分别报道,几种支原体与许多慢性退行性疾病(如多发性硬化症和阿尔茨海默病)密切相关。
每年关于肺炎支原体疾病期间“肺外症状”的报告数量一直在增加。临床和流行病学数据表明,症状来自皮肤和粘膜、中枢神经系统、心脏。
在患有脑膜炎和脑炎、癫痫、肌萎缩侧索硬化(ALS)、阿尔茨海默病和其他中枢神经系统感染、疾病和紊乱的患者的脑脊液中发现了它。
而且,肺炎支原体以及至少 7 种其他支原体现已被认为是许多慢性疾病的直接原因或重要辅助因素,包括类风湿性关节炎、阿尔茨海默病、多发性硬化症、纤维肌痛、慢性疲劳、糖尿病、克罗恩病、ALS、非淋菌性尿道炎、哮喘、狼疮、不孕症、艾滋病以及某些癌症和白血病。
在肺炎支原体大流行的情况下,本文汇编查阅了许多研究资料,带大家了解下关于支原体这个神秘的细菌,菌属特性,致病性以及传播和防御等。
支原体(Mycoplasmas)是最小、最简单的自我复制细菌。支原体细胞含有生长和复制所必需的最小细胞器:质膜、核糖体和由双链环状DNA分子组成的基因组。与所有其他原核生物不同,支原体没有细胞壁,因此它们被归类为单独的。
支原体细胞薄片的电子显微照片
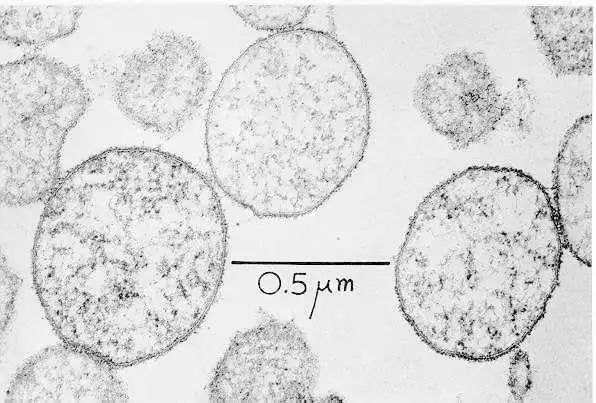
Baron S, editor. Medical Microbiology Chapter 37, Mycoplasmas
细胞以单层膜为界,在剖面上显示出特征性的三层形状。细胞质包含代表染色体切片的细线和代表核糖体的深色颗粒。
支原体的快速增殖,可以作为污染物抢占空间
支原体可以作为污染物出现在细胞培养中,支原体在细胞培养物中也会快速增殖,并与培养的细胞竞争营养和空间,从而导致培养失败或研究结果的偏差。
支原体:微小无细胞壁
支原体直径为 0.1-0.3 μm,长度可达 98 μm。它们在自然界中无处不在,广泛存在于人类、哺乳动物、爬行动物、鱼类、节肢动物和植物中。它们生活在呼吸道和泌尿生殖道的粘膜表面、眼睛、消化道、乳腺和关节中,通常表现出器官和组织特异。支原体属于 Mollicutes 类(拉丁语中 mollis = 软,cutis = 皮肤),它们体积小且没有细胞壁,这有助于将它们与其他细菌区分开来。
进化假设:寄生生活方式导致细胞壁的退化
一种假设(还原进化或退化进化)指出,支原体通过采用寄生生活方式而失去了细胞壁和其他生物合成途径。根据这一假设,寄生生活方式使得细胞壁的存在变得一次性。因此,支原体逐渐失去了合成构建细胞壁所需的聚合物所需的基因。
通过在宿主环境中作为寄生虫生活,这种发展并没有导致进化劣势。作为一种权衡,支原体依赖于其宿主来满足许多基本营养需求,这阻碍了它们在培养物中的生长,从而阻碍了对其致病决定因素的详细研究。
支原体具有较小的环状双链基因组
同一物种的菌株之间存在差异,范围从小于600 kb到2200 kb,并且它们合成的蛋白质数量相对较少;因此,代谢能力有限。
支原体膜简单坚固,复制存活依赖宿主或培养基
支原体的膜非常简单、坚硬、薄而坚固,由甾醇(脂肪酸、胆固醇或复合脂质)组成。这些分子是从周围环境中吸收的,而不是由这些微生物合成的;它们的复制和存活取决于宿主产生的因子或生长培养基吸收的因子。
支原体可寄生于细胞外或内
支原体物种既存在于细胞外作为膜表面寄生虫,也存在于细胞内,在细胞内它们成为细胞内居民作为“沉默的寄生虫”。其他数据显示艾滋病患者细胞样本中发酵支原体的细胞内定位,并且支原体(名为穿透支原体)能够在体内和体外进入许多不同的人类细胞。
最近,共聚焦显微照片证明了肺炎支原体结合和内化的能力,具体取决于细胞类型。
支原体是多种疾病的病原体和辅助因子
通常引起慢性感染,其大多数致病决定因素的身份和作用机制尚未完全了解。由于粘膜表面破坏、局部创伤、手术、组织坏死和无菌部位清除受损,支原体倾向于定殖、损伤和侵入深层组织。由于它们可以在厌氧环境中生长,这可能会导致局部感染。
支原体常作为局部感染的病原体,RT-PCR提高了检测效率
事实上,在许多情况下,支原体是这些局部感染的病原体,并且通过实验室实践对其进行分离和鉴定的困难可能导致这些关联被低估。最近添加的带有特异性引物的实时聚合酶链反应(RT-PCR)可以特异性确定感兴趣位点中是否存在支原体。例如,RT-PCR 是生殖支原体的首选诊断方法,但由于生长极慢,在常规培养中无法检测到支原体。在其他情况下,例如解脲脲原体和人型支原体,它们的快速生长允许使用常规培养来确定它们的存在,在这种情况下,RT-PCR 可以用作确认性和更快的检测方法。
蛋白质序列技术的进步,已经能够鉴定人类(例如肺炎支原体、生殖支原体和发酵支原体)和动物(鱼类中的移动支原体、猪肺炎支原体和猪中的絮状支原体)支原体致病性的潜在决定因素。
因此,一些数据表明,支原体膜上存在的脂质蛋白与单核细胞/巨噬细胞相互作用,调节免疫反应,有时导致免疫系统逃避。
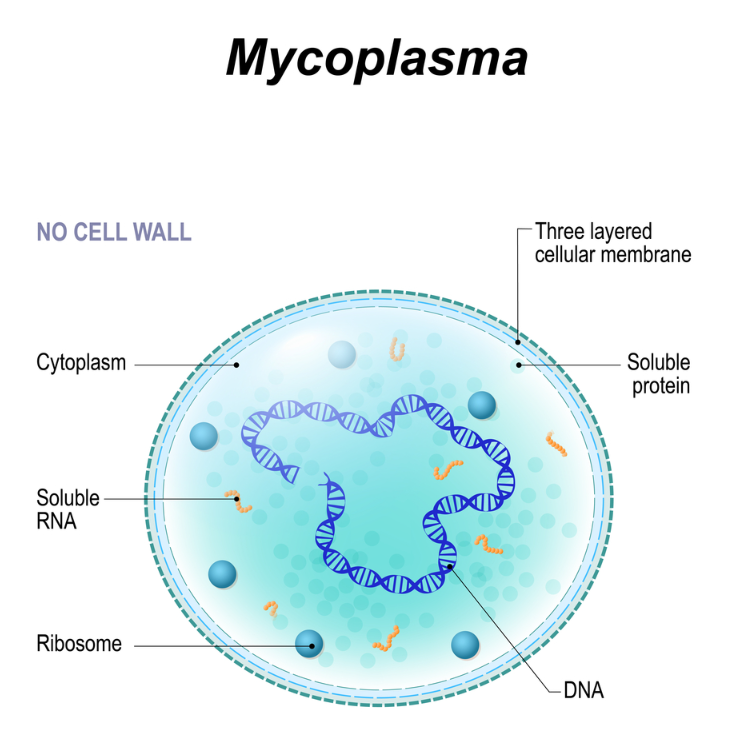
std-gov
支原体的基因组特征和形态多样性
支原体基因组的鸟嘌呤-胞嘧啶(G + C)含量较低,并且相应地优先使用含有腺嘌呤和尿嘧啶的密码子,特别是在第三位。最有趣的是在许多支原体中使用通用终止密码子 UGA 作为色氨酸密码子,这是迄今为止仅在支原体和非植物线粒体中发现的罕见特性。
其变异性是由于重复元件(由大小和数量不同的基因片段组成)或插入序列元件(IS)造成的。它们的形状由细胞骨架的存在控制,细胞骨架也有助于细胞分裂(通过二元裂变进行繁殖)和支原体的运动。支原体的主要形状是球形,但它们可以有小的球虫体、肿胀的环状形式和长度可变的丝状分支形式。
支原体的繁殖方式和生长特点
支原体通过细胞出芽和二元横向裂变进行繁殖。支原体没有鞭毛,不产生孢子,并且是革兰氏阴性。几乎所有寄生在人类和动物身上的支原体以及所有腐生支原体都可以在或多或少复杂的人工营养培养基上生长,在其中它们产生微小的菌落,通常具有特征性的“煎蛋”外观。
支原体繁殖模式的示意图
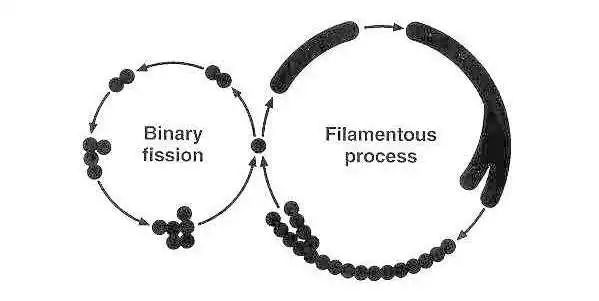
细胞可以通过二元裂变分裂或首先伸长为多核丝,随后分裂为球状体。
支原体附着细胞器
一些支原体具有独特的附着细胞器,在肺炎支原体和生殖支原体中形状为锥形尖端。肺炎支原体是呼吸道病原体,主要通过附着细胞器粘附在呼吸道上皮上。有趣的是,这两种人类支原体在液体覆盖的表面上表现出滑动运动。尖端结构始终领先,再次表明其在附着中的重要性。
煎蛋菌落形状
支原体最有用的显着特征之一是其独特的煎蛋菌落形状,由嵌入琼脂的中心生长区和琼脂表面的外围生长区组成(下图)。
典型“煎蛋”支原体菌落的形态
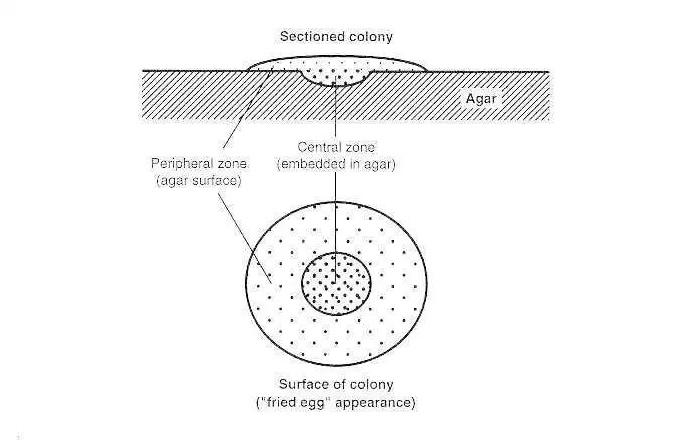
支原体膜的组成和特点
缺乏细胞壁和胞质内膜有利于以相对纯净的形式分离支原体膜。分离的支原体膜与其他原核生物的膜相似,由大约三分之二的蛋白质和三分之一的脂质组成。除了需要甾醇的支原体中含有大量胆固醇之外,支原体脂质与其他细菌的脂质相似。
支原体的抗原决定簇和血清学测试
暴露在细胞表面的膜蛋白、糖脂和脂聚糖是支原体的主要抗原决定簇。含有针对这些成分的抗体的抗血清抑制支原体的生长和代谢,并且在补体存在的情况下引起生物体裂解。这些特性用于区分支原体种类和血清型的各种血清学测试,并检测患者血清中的支原体抗体。
支原体的来源和对抗生素的敏感性
支原体主要从健康和/或患病动物以及患有呼吸道和泌尿生殖道疾病的人类中分离出来;它们与动物的一些关节炎和神经疾病有关;有些被发现以腐生菌的形式存在。
大多数支原体对青霉素完全耐药;支原体RNA聚合酶对利福平的抗性是支原体区别于传统真细菌的另一个特性。然而,它们对四环素和氯霉素敏感,有些对红霉素和某些其他抗生素敏感。
支原体的系统发育
作为最小和最简单的自我复制原核生物,支原体提出了一个有趣的问题:它们是否代表了在基于肽聚糖的细胞壁发育之前就存在的极其原始的细菌的后代,或者它们是否代表了已经失去其原始特征的进化退化的真细菌形式。
分子进化研究主要依靠核糖体RNA(rRNA)序列,特别是16S rRNA序列的比较。rRNA序列高度保守,比较结果支持支原体从其他细菌进化而来的假说。根据科学家的研究,支原体是从低鸟嘌呤加胞嘧啶革兰氏阳性菌分化出来的一个分支。支原体与两种梭状芽胞杆菌Clostridium. innocuum和Clostridium ramosum的关系最为紧密。也有科学家认为支原体的起源是单系的,并通过该群体的快速进化过程来解释支原体的多样性。
在大多数情况下,体内可能存在的 200 多种不同种类的支原体是完全无害的。然而,有五种特定形式可能导致感染和严重的健康问题:
这些细菌生活宿主呼吸系统中,可能导致肺部感染。通常也被称为“行走性肺炎”。
大多数感染这种细菌的人都会患上轻度肺炎。症状包括疲劳、喉咙痛、头痛和慢性咳嗽。在某些情况下,可能会出现支原体皮疹(类似于荨麻疹的红色皮疹)。
注:“行走性肺炎”是一种肺部感染,约三分之一的支原体感染者受到影响。这种感染也被称为行走性肺炎,在儿童中,它被称为支气管炎。
症状
肺炎支原体可引起上呼吸系统感染,也可模仿常见的肺炎支原体感染。与这种感染相关的一些常见症状包括干咳、不适、持续发烧和轻度呼吸急促。在极少数情况下,这种感染还可以通过损害中枢神经系统和心脏而达到临界水平。
与这种感染相关的一些常见疾病包括溶血性贫血、肾衰竭、关节炎、心包炎、中毒性表皮坏死松解症和格林巴利综合征。
常见的治疗方案
大多数时候,专家建议使用抗生素治疗这种感染。对于儿童,阿奇霉素、红霉素、克拉霉素足够有效,而对于成人,专家建议四环素、强力霉素、莫西沙星等。
当抗生素无法控制疾病时,专业人士可能还会建议使用泼尼松龙等皮质类固醇来治疗支原体。
预防肺炎支原体的小贴士
这种感染的风险在冬季和秋季会增加。这种感染可以在人与人之间传播,因此最好避免去拥挤的地方。一些预防技巧包括:保证充足的睡眠,每晚至少 6-8 小时,均衡饮食。最好避免与已经感染肺炎支原体的人接触。与人互动后和吃饭前切勿忘记洗手。
这些细菌生活在宿主的尿道和生殖器内及其周围。如果与已感染细菌的人发生性行为,就会发生这种细菌感染。
症状
可怕的事实是,生殖支原体并不总是伴随症状。许多报告表明,即使一个人根本没有感觉到任何症状,这种疾病也存在。症状因性别而异。
一般来说,女性的症状包括:
如果是男性,症状可能是:
研究表明,百分之一的成年人可能患有这种性病。有些人还尝试将支原体和衣原体联系起来;然而,它们是两种不同的性传播疾病,几乎没有共同的症状,但原因和治疗方法却不同。
常见的诊断治疗
医疗卫生专家认为,生殖支原体是一个严重的问题,诊断也很棘手。几种可能的选项是:
医生可能会推荐阿奇霉素,但如果没有任何改善,那么可能需要服用莫西沙星。继续服用该药物至少一个月后,需要再接受一次医学检查,看看感染是否消失。但都需要根据症状和感染程度采取特定的治疗。
这些细菌主要影响女性并生活在女性的生殖系统和泌尿道中。对于大多数健康人来说,这些细菌从未被注意到,而且几乎没有症状。这种细菌感染的迹象通常只出现在怀孕和分娩期间,因为细菌可以从感染该细菌的母亲那里传染给新生儿。它可能导致流产或早产,以及新生儿发烧。
症状
常见的诊断、治疗、预防
➤ 4
解脲支原体
(Ureaplasma Urealyticum)
小脲原体(Ureaplasma parvum)
这些细菌生活在男性的尿道和女性的子宫颈或阴道中。几乎所有健康的成年人都有这种细菌,但如果它导致感染,就会造成健康问题。
很少有男性携带这种细菌,但大多数健康女性都有。细菌在性交过程中传播,并可能导致阴茎或阴道分泌物、腹痛和生殖器异味等症状。
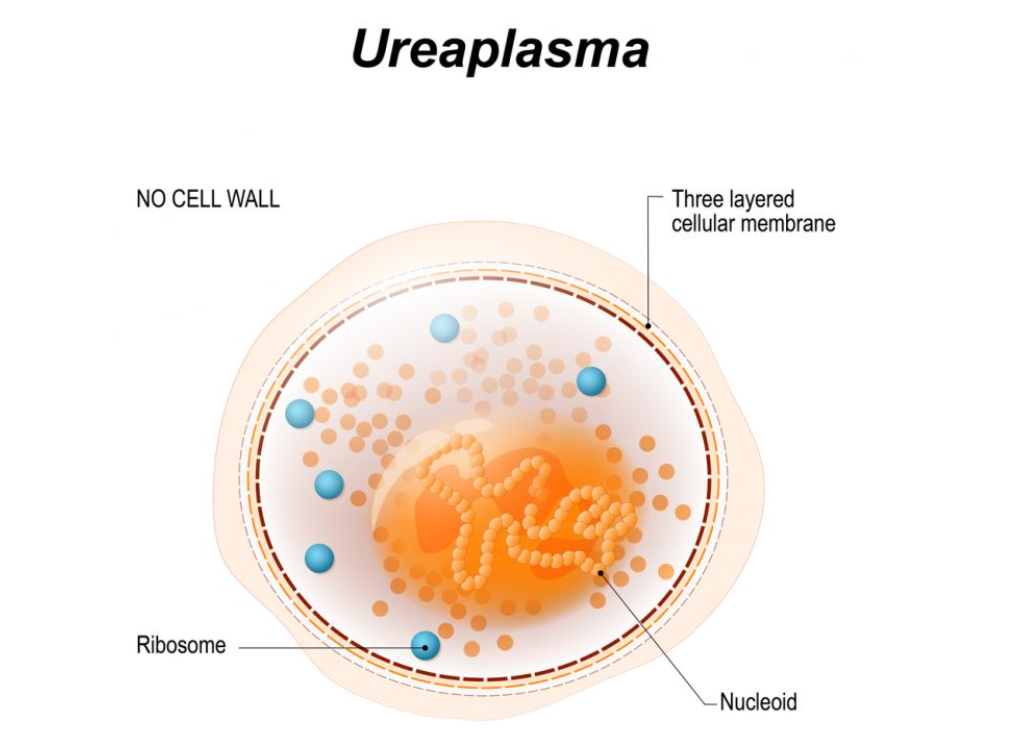
健康女性也可能感染解脲支原体和微小解脲支原体。虽然它们不会对你的身体产生任何重大影响,但几乎观察不到基本的疼痛和分泌物症状。支原体和解脲支原体之间有一个共同的关系,即它们都存在于男性和女性的生殖道中。如果出现一些疾病症状,最好就医。
大多数人类和动物软体动物属于支原体科的解脲支原体属。支原体是由革兰氏阳性细菌进化而来的,这些细菌经历了显着的基因组缩减;因此,该属的大多数成员表现出宿主和组织特异性,并且复制和生存的代谢选择有限,迫使它们适应从宿主获取代谢前体。
支原体的入侵
为了完成这一致病过程,支原体必须首先克服几个障碍才能成功侵入宿主的防御并达到完整的生命周期。
支原体的侵袭性是由表面粘附素及其辅助蛋白、荚膜多糖、侵袭酶和生物膜介导的,所有这些都确保了支原体在体内的建立、繁殖和传播。
在寄生周期中,支原体吸收宿主细胞的营养物质并释放大量代谢物,如过氧化氢(H2O2)、氨(NH3)和硫化氢(H2S),造成局部组织损伤。
支原体分泌毒素,表达多种致病酶
某些支原体还可以分泌一些外毒素,包括社区获得性呼吸窘迫综合征毒素(CARDS TX)和溶血素。这些毒素具有不同的功能,可以通过多种方式影响宿主细胞功能。
支原体还可以表达多种致病酶,如脂肪分解酶、肽酶、磷酸酶、胞外ATP酶、细胞毒性核酸酶和核苷酸酶,被认为是重要的支原体致病因子。
此外,支原体细胞膜上的一些固有成分,例如脂质、膜脂蛋白,甚至关节炎支原体产生的超抗原,都可以通过各种策略引发炎症反应。
支原体毒力因子示意图
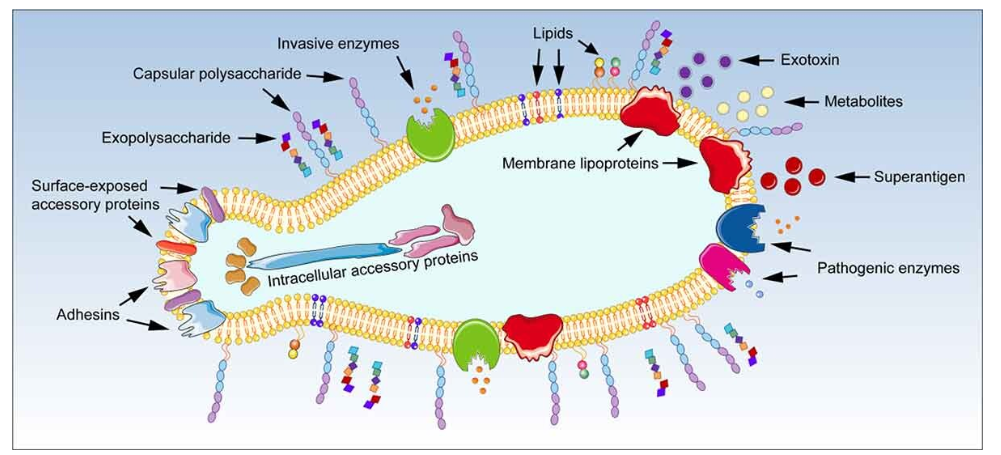
doi: 10.1080/21505594.2021.1889813
支原体的毒力因素包括侵袭性、类毒素物质、外毒素、致病酶和一些膜成分。
注:类毒素物质主要包括增殖过程中产生的代谢产物,如H2O2、NH3、 H2S等。
支原体附着的受体位点(宿主细胞膜)
肺炎支原体和生殖支原体的附着受到神经氨酸酶预处理宿主细胞的影响,宿主细胞膜的唾液酸糖蛋白和/或唾液酸糖脂似乎是这些支原体的受体位点。
有证据表明,几种肺炎支原体膜蛋白充当粘附素,并且它们对宿主细胞上肺炎支原体的特异性受体具有高亲和力。其中一种蛋白质,即蛋白质 P1的单克隆抗体可抑制支原体的附着。抗体的铁蛋白标记表明 P1 集中在支原体的尖端结构上,这一发现进一步支持了尖端作为附着细胞器的观点。
支原体粘附蛋白P1和MgPa
对肺炎支原体(P1) 和生殖支原体(MgPa)主要粘附素的基因进行了克隆和测序,从而能够表征这些蛋白质。这两种粘附素在许多方面都很相似,并且实际上包含广泛的同源性区域,这也通过共享表位来表达。这两种蛋白质可能是经历水平基因转移事件的祖先基因的产物。
支原体代谢产物过氧化氢对宿主细胞的损伤
损害支原体感染粘膜表面的毒性因子的性质仍不清楚。因此,研究人员考虑支原体代谢的终产物是否导致组织损伤。过氧化氢(H2O2)是支原体呼吸的最终产物,自从在体外被证明它能导致支原体裂解红细胞以来,它就被认为是主要的致病因素。
然而,H2O2的产生本身并不能决定致病性,因为肺炎支原体毒力的丧失并不伴随着H2O2产生的减少。为了使 H2O2发挥其毒性作用,支原体必须足够紧密地粘附在宿主细胞表面,以维持有毒的稳态 H2O2浓度,足以对宿主细胞造成直接损害,例如脂质过氧化。
粘附肺炎支原体对宿主细胞造成氧化损伤的机制
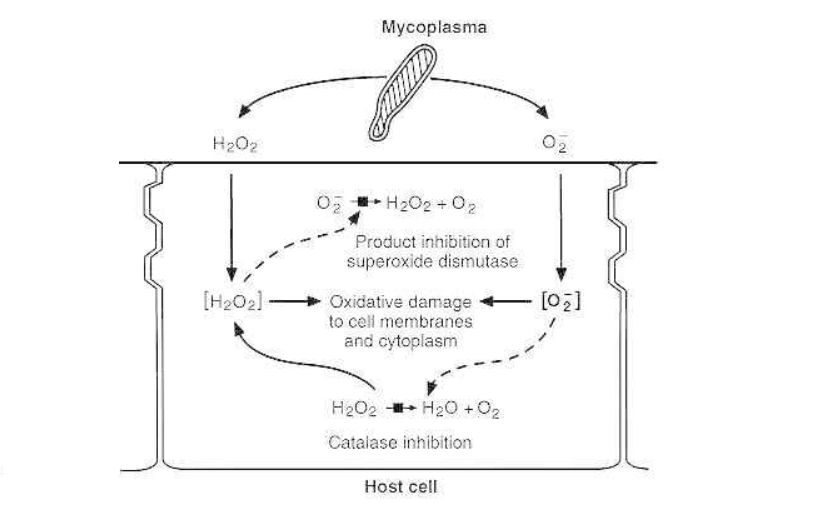
Medical Microbiology. Chapter 37, Mycoplasmas
生物体相关因素和宿主相关因素均参与支原体感染的发病机制
支原体激活巨噬细胞,诱导细胞因子产生和淋巴细胞增殖;大鼠病原体关节炎支原体可产生有效的超抗原。因此,就肺炎支原体而言,宿主可能通过对支原体产生局部免疫反应而对肺炎负有主要责任。
鼻内接种肺炎支原体的仓鼠显示出由单核细胞浸润组成的斑片状支气管肺炎病变。实验感染前胸腺功能的消融可防止特征性肺部浸润的发展,但会延长微生物从肺部分离的时间。当胸腺动物恢复并再次感染时,就会发生夸大和加速的肺炎过程。
流行病学数据还表明,在出现症状性疾病之前,需要在人类中反复感染:大多数 2 至 5 岁的儿童中都可以发现肺炎支原体的血清抗体,尽管这种疾病在 5 至 15 岁的个体中发生的频率最高。
免疫病理学机制可解释一些肺炎支原体并发症
免疫病理学机制也可以解释一些感染肺炎支原体的患者出现影响远离呼吸道的器官的并发症。在许多此类患者的血清中检测到了各种自身抗体,包括与红细胞 I 抗原反应的冷凝集素,以及与淋巴细胞、平滑肌细胞以及脑和肺抗原反应的抗体。
肺炎支原体与脑和肺抗原之间的血清学交叉反应已被证明,这些抗原可能与肺炎支原体膜的糖脂有关,这种糖脂也存在于大多数植物和许多细菌中。显然,宿主反应差异很大,因为只有大约一半的患者出现冷凝集素,并且即使在具有抗组织球蛋白的个体中,并发症也很少见。
肺炎支原体肺炎最令人费解的特征之一是患者的年龄分布。1964年至1975年间对西雅图地区10万多人进行的一项调查显示,5岁至9岁儿童的特定年龄发病率最高。最年轻年龄组(0至4岁)的肺炎支原体肺炎发病率约为学龄儿童的一半,但明显高于成人。
肺炎支原体肺炎在6个月以下的婴儿中很少观察到,这表明母体赋予了免疫力(下图)。肺炎支原体占学龄儿童所有肺炎的 8% 至 15%(科研统计数据,只做参考)。在年龄较大的儿童和年轻人中,约 15% 至 50% 的肺炎是由这种微生物引起的。
肺炎支原体感染全年都会在世界范围内发生,但在较冷的月份中更容易发生,这显然是因为通过飞沫感染传播的机会更大。肺炎支原体似乎需要密切的个人接触才能传播;成功的传播通常发生在家庭、学校和机构中。潜伏期为2至3周。
传播途径
风险因素
对于大多数人来说,这些细菌是他们日常生活中无害且始终存在的一部分。然而,如果有以下风险因素,那么发生感染的风险就会增加:
支原体脂蛋白与宿主细胞相互作用
支原体附着在外部细胞膜上,一方面导致某些细菌蛋白(脂蛋白LP/脂肽或特定附着细胞器)与靶细胞表面上的特定细胞受体之间的相互作用手。为此,许多研究已经鉴定出几种支原体的脂蛋白可以与宿主生物体的上皮细胞和白细胞相互作用。
免疫细胞的炎症反应与支原体脂蛋白结合的关系
当这些细菌蛋白与免疫细胞中表达的特定受体(模式识别受体PRR)结合时,就会发生炎症反应。更详细地说,病原体相关分子模式 (PAMP) 通过与专门的 PRR——Toll 样受体 (TLR) 和核苷酸结合寡聚化结构域蛋白 (NOD) 样受体相互作用,被先天免疫系统的细胞识别。
支原体的脂蛋白可以通过与TLRs 1、2、4和6结合来引发炎症反应。其中第一个被鉴定的脂肽是支原体的巨噬细胞活化脂肽-2(MALP-2),它可以通过与TLRs结合来激活细胞和表达NF-κB。
三酰基或二酰基脂肽分别与TLR 1/2或TLR 2/6异二聚体结合。TLR2基因敲除小鼠无法通过MALP-2来激活信号通路,这进一步证实了该信号通路的重要性。支原体感染可引发乳腺炎、输卵管炎、尿道炎、非典型肺炎等发炎性疾病。
支原体影响参与炎症和细胞转化的细胞途径
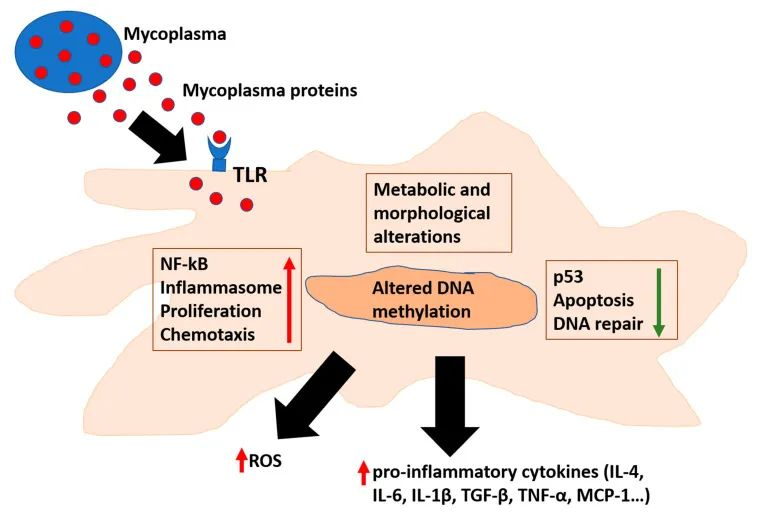
Benedetti F et al. 2020. Microorganisms
支原体的蛋白质与 TLR 相互作用或进入细胞,在那里它们可以改变负责炎症和 DNA 修复的多种途径。此外,影响细胞 DNA 甲基化会导致细胞表观遗传景观的改变。TLR:Toll 样受体;ROS:活性氧。TGF:转化生长因子;TNF:肿瘤坏死因子;和MCP-单核细胞趋化蛋白。
支原体感染引起的炎症反应和免疫细胞迁移
这些炎症反应是由支原体感染后上皮细胞和白细胞分泌的免疫因子所致,其后会激活NF-κB和多种细胞因子,从而引起粒细胞、巨噬细胞和淋巴细胞的迁移,使它们集中到感染部位。
个别脂肽如三酰基脂肽也能在没有完整支原体的情况下促进白细胞浸润。TLRs与NF-κB的结合可能会启动细胞的抗凋亡程序,最终产生促癌作用。
总而言之,研究数据表明支原体配体在进化上具有保守性,能够引发相同的细胞信号传导反应。值得注意的是,支原体中的单个脂肽可以诱导炎症,与整个微生物分离,这表明可能对细胞产生旁分泌作用。
慢性阻塞性肺病(COPD)可能与支原体有关
虽然支原体水平高和炎症水平升高可以很容易地解释其致病性,但在某些情况下,其负面影响的机制尚不清楚。
一个例子是慢性阻塞性肺病 (COPD),其两种病理表现(慢性支气管炎和肺气肿)是发病率和死亡率日益增加的原因(全世界有 130,000 人死亡)。
COPD病因和症状
长期接触刺激物(主要是吸烟和空气污染物)会引发肺部炎症反应,导致小气道狭窄、肺组织破裂和进行性肺泡破坏(肺气肿),并出现呼吸困难、咳嗽等症状和痰液产生。
尽管呼吸道症状是慢性阻塞性肺病的标志,但非肺部症状也经常出现。因此,心血管、内分泌和肌肉骨骼合并症的风险增加。
炎症对COPD患者的全身影响
这些非肺部表现很可能是由炎症过程引发的免疫功能障碍介导的,炎症过程最初在肺部触发,并在全身传播,导致并加重合并症。
在这方面,在 COPD 患者中观察到的循环炎症生物标志物水平升高是这些全身效应的潜在介质。此外,COPD 患者的循环功能性 T 调节细胞 (Treg)、骨髓源性抑制细胞 (MDSC) 和衰竭性程序性死亡 (PD) 1 + 细胞的水平也显着升高,这些细胞会导致效应 T 细胞功能障碍和降低他们抵抗感染的能力。
COPD患者肺部微生物变化
与健康人中观察到的微生物群相比,肺部微生物群的特征发现多样性显着减少。特别是,在慢性阻塞性肺病患者中,微生物群的组成似乎仅限于包括潜在致病微生物的门,例如肺炎支原体,它也与急性加重有关。
包括支原体在内的许多细菌的病原体与癌症的精确关系在很大程度上仍然难以捉摸。
我们特别注意到,一些细菌能够通过侵入宿主细胞并在很长一段时间内不被免疫系统检测到来建立持续的慢性感染。它们产生的蛋白质会干扰和改变重要细胞途径的功能,如细胞周期控制、细胞凋亡、DNA 修复。这与这些病原体诱导能够增加 DNA 损伤的物质的能力有关,可能会增加异常细胞的生长和转化。
▼
许多研究已经在慢性炎症、肿瘤进展和 p53 之间建立了牢固的联系,鉴于 p53 在保持基因组稳定性方面的核心作用,p53 无疑是人类最重要的肿瘤抑制蛋白。支原体感染细胞后,由于p53功能受损无法适当修复DNA损伤,从而潜在增加癌症发生和发展风险。
注:p53是一种蛋白质,也被称为肿瘤蛋白53。它在细胞中起着关键的调控作用,主要参与细胞周期的调控、DNA修复和细胞凋亡等过程。p53蛋白质的功能异常与多种癌症的发生和发展有关。正常情况下,p53可以抑制异常细胞的生长和分裂,促进DNA修复,或引导细胞进入凋亡途径。当细胞受到DNA损伤或其他应激刺激时,p53蛋白质会累积并激活一系列的反应,维护基因组的稳定性。然而,当p53基因发生突变或其他异常时,p53功能可能受到抑制或失调,导致异常细胞的增殖和存活,从而增加患癌风险。p53是癌症研究中的重要研究对象,也是潜在的治疗靶点。
慢性炎症通过NF-κB降低p53的活性:细胞命运的关键决定因素之一
NF-κB 降低 p53 的活性,抗凋亡 NF-κB 和促凋亡 p53 之间的相互调节是细胞命运的主要决定因素之一。事实上,在不同肿瘤细胞系中对持续活化的 NF-κB 进行遗传或药理学抑制会导致 p53 功能激活,并通过 p53 依赖性细胞凋亡导致肿瘤细胞死亡。
鉴于炎症可以降低 p53 的活性,慢性炎症可能通过 NF-κB 的激活降低 p53 的活性;因此,促进细胞转化。
p53参与DNA损伤响应和细胞周期调控
DNA 损伤和其他应激信号后,细胞 p53 蛋白的低水平增加,导致生长停滞、DNA 修复或细胞凋亡。细胞周期的中断可防止受损 DNA 的复制,从而使 p53 能够激活参与 DNA 修复的蛋白质的转录。
另一方面,当该途径受到损害时,细胞会激活导致细胞凋亡的途径,这是避免含有异常 DNA 的细胞增殖的选择机制。
p53活性受严格调控
由于这些原因,p53的细胞浓度和活性必须受到严格调节,p53的主要调节因子是Mdm2,它通过将p53保留在细胞质中并通过泛素系统激活其降解来发挥作用。
Mdm2 通过反馈机制受到 p53 以及参与生长停滞、DNA 修复和凋亡的基因(例如 p21、Gadd45、BAX 和 PUMA)的调节。
▼
骨形态发生蛋白 2 (BMP2) 是一种重要的生长因子和形态发生素,与癌症的促进和生长有关。
事实上,已经表明,穿透支原体、发酵支原体和人型支原体的感染会在通常不表达此类蛋白的细胞,包括 BEAS-2B 细胞(永生化细胞)中诱导 BMP2 RNA 表达以及成熟 BMP2 蛋白的分泌。
人支气管上皮细胞、A549 细胞(肺腺癌细胞)以及其他几种不同来源的细胞系(间质细胞、上皮细胞和骨髓细胞)。
支原体感染细胞中 BMP2 表达的增加,主要是通过调节 RNA 稳定性来实现的,而不是影响转录水平。此外,研究表明,BMP2 刺激慢性支原体感染转化的 BEAS-2B 细胞的增殖,表明支原体感染对 BMP2 调节途径的深远影响,包括参与细胞增殖、分化和凋亡的途径。
▼
将支原体与致癌作用联系起来的进一步研究通过其参与 DNA 甲基化模式的变化来说明。DNA 甲基化(即胞嘧啶向 5-甲基胞嘧啶的转化)是转录调控的重要元素,也是主要的表观遗传机制之一。
许多应激或 DNA 损伤实际上会干扰 DNA 被 DNA 甲基转移酶 (DNA-MTase) 在 CpG 二核苷酸处甲基化的能力。当特定支原体MTase 在人类细胞系中表达时,已观察到它们易位至细胞核。结果是人类基因组甲基化格局发生了变化,因为这些细菌酶使人类基因组 DNA 中各自 CG 和 GATC 位点内的胞嘧啶甲基化,从而刺激促癌途径。
▼
其他报告强烈表明支原体在细胞转化中的作用,目前正在积极研究支原体与癌症之间的联系。为此,许多研究证明支原体对细胞系的影响,表明支原体可能促进肿瘤发生,例如在口腔组织中、在人前列腺细胞、胃癌细胞和宫颈细胞中两者都是体外的。
在体内,一些研究报道了在各种肿瘤组织和体液中分离出支原体,特别是在癌前病变以及胃癌、结肠癌、卵巢癌、肺癌和肝细胞癌患者的恶性组织中发现了支原体,尽管迄今为止尚未证明与细胞转化有直接的因果关系。尽管如此,所有概述的研究和支原体的特性都强烈表明这些物质是癌症促进因素。
▼
将患者痰液、脓液等样本接种到适当的细胞培养基中,观察是否有支原体在细胞内生长繁殖。这是一种传统的诊断方法。
采用聚合酶链反应(RT-PCR)技术检测支原体DNA特异序列。PCR检测灵敏度高,能快速检出病原体。
高通量测序技术可以应用于支原体的检测和鉴定。支原体是一类细菌,通过对其基因组进行测序分析,可以确定其物种、亚型和耐药性等信息。
ELISA法检测患者血清中支原体特异性抗体IgM和IgG。这种方法操作简单、成本低。
采用荧光标记的抗支原体抗体对病理切片或细胞培养物进行染色,观察荧光信号判断是否感染。
HE染色下观察支原体在组织中的形态和位置。电镜下可以直接观察到支原体细胞内的结构。
检测CRP、ESR等炎症标志物升高情况,辅助支持支原体感染的诊断。
这是一种传统的方法,通过细胞形态学特征判断是否感染支原体。
▼
不建议对支原体感染进行化学预防,并且没有可用的疫苗。先前的自然感染似乎提供了最有效的抵抗力;然而,有证据表明肺炎支原体感染每隔几年就会复发。这些观察结果表明,对单一自然感染的免疫力相对较短。
以下方式可能有助于预防,仅供参考:
▼
支原体感染的治疗通常采用抗生素进行,具体的药物选择和治疗方案会根据支原体的种类、临床症状和耐药性等因素进行确定。
常用的治疗支原体感染的药物:
• 大环内酯类抗生素
如红霉素、四环素、阿奇霉素等,常用于治疗肺炎支原体感染。
• 四环素类抗生素
如多西环素、米诺环素等,常用于治疗衣原体感染。
• 喹诺酮类抗生素
如氧氟沙星、左氧氟沙星等,常用于治疗衣原体、肺炎支原体感染。
• β-内酰胺类抗生素
如头孢菌素、阿莫西林-克拉维酸等,常用于治疗鸡眼支原体感染。
• 大环内酯类和四环素类联合治疗
对于一些耐药性较强的支原体感染,联合使用大环内酯类和四环素类抗生素可能会提高疗效。
需要注意的是,抗生素的使用应该根据医生的建议和具体情况来确定,遵循药物的使用指南和剂量要求。
此外,抗生素治疗支原体感染时,应严格按照疗程完成,以避免耐药性的产生。
除了抗生素治疗外,还可以辅助应用一些支持疗法,如充分休息、保持良好的营养状态、健康胃肠道生态、增强免疫力等,以帮助更快地康复。最终的治疗方案应根据临床情况和医生的建议来确定。
注:本账号内容仅作交流参考,不作为诊断及医疗依据。
主要参考文献
Bajantri B, Venkatram S, Diaz-Fuentes G. Mycoplasma pneumoniae: A Potentially Severe Infection. J Clin Med Res. 2018 Jul;10(7):535-544.
Meyer Sauteur PM, Beeton ML; ESGMAC the ESGMAC MAPS study group. Mycoplasma pneumoniae: gone forever? Lancet Microbe. 2023 Oct;4(10):e763.
Meyer Sauteur PM, Beeton ML; European Society of Clinical Microbiology and Infectious Diseases (ESCMID) Study Group for Mycoplasma and Chlamydia Infections (ESGMAC), and the ESGMAC Mycoplasma pneumoniae Surveillance (MAPS) study group. Mycoplasma pneumoniae: delayed re-emergence after COVID-19 pandemic restrictions. Lancet Microbe. 2023 Nov 23:S2666-5247(23)00344-0.
Kashyap S, Sarkar M. Mycoplasma pneumonia: Clinical features and management. Lung India. 2010 Apr;27(2):75
Siles-Guerrero V, Cardona-Benavides I, Liébana-Martos C, Vázquez-Alonso F, Expósito-Ruiz M, Navarro-Marí JM, Gutiérrez-Fernández J. Recent clinical relevance of mono-genital colonization/infection by Ureaplasma parvum. Eur J Clin Microbiol Infect Dis. 2020 Oct;39(10):1899-1905.
sciencedirect.com/topics/medicine-and-dentistry/mycoplasmataceae
Yiwen C, Yueyue W, Lianmei Q, Cuiming Z, Xiaoxing Y. Infection strategies of mycoplasmas: Unraveling the panoply of virulence factors. Virulence. 2021 Dec;12(1):788-817.
Benedetti F, Curreli S, Zella D. Mycoplasmas-Host Interaction: Mechanisms of Inflammation and Association with Cellular Transformation. Microorganisms. 2020 Sep 4;8(9):1351.

谷禾健康


DNA 测序是测量各种生命形式主要特性的基础。自20世纪50年代发现DNA双螺旋结构后,全世界科学家们就开始致力于确定不同物种基因组的原始序列。这一任务被称为基因组测序,旨在揭示不同生物的基因组组成和基因的排列顺序。
现代基因组研究标志之一是生成大量原始序列数据。这项工作的重要性在于,基因组序列的破译可以提供关于生物的遗传信息,包括基因功能、遗传变异和进化关系等方面重要线索。
在过去的几十年里,随着测序技术的不断发展和突破,测序的速度和精度都得到了显著提高。早期的测序方法主要依赖于Sanger测序技术,该技术基于DNA链延伸的原理,通过测量DNA链延伸反应中释放的荧光标记物来确定DNA序列。然而,由于其低通量和高成本的限制,Sanger测序逐渐被新一代测序技术(NGS)所取代。
随着新一代测序技术的兴起,如Illumina的高通量测序和454 Life Sciences的Roche测序平台以及华大基因(BGI)DNBSEQ-T7等测序平台,基因组测序进入了一个全新的时代。这些技术利用并行测序的原理,可以同时测序数百万个DNA片段,大大提高了测序的速度和效率。同时,这些技术也在成本和准确性方面取得了显著的突破,使得大规模基因组测序成为可能。
随着测序技术的不断进步,越来越多的原核和真核基因组序列被测序出来,并存储在公共数据库中,目前四个主要的数据库是:
它们目前拥有丰度的实验以及样本核苷酸序列原始数据,此外还有蛋白质序列或大分子结构数据。这些数据库为科学家们提供了宝贵的资源,可以用于研究和比较不同物种的基因组,从而增进对生物多样性、进化和基因功能的理解。
除了基因组序列的分析,还需要开发各种生物信息学工具和数据库,以帮助解释和注释基因组数据。这些工具可以用于预测基因的功能、识别调控元件、比较不同物种之间的基因组差异等。
随着计算机技术的不断进步,测序数据也逐渐借助人工智能和机器学习等技术。这些技术可以辅助人们更快速、准确地分析和解释基因组数据,发现隐藏在数据中的模式和关联。机器学习算法可以用于预测基因的功能、识别基因组中的重要调控区域或精确区分相近物种等。
例如谷禾肠道菌群16S数据库,是谷禾健康从几十万人肠道菌群检测数据中提取的16S序列库,通过宏基因组匹配数据和模型构建,重新完成到种的物种注释。进一步通过基因组数据研究和分析,为您提供深度个性化的健康检测方案。
目前测序技术已经在许多应用领域产生了广泛的影响。例如,基因组测序为研究人类遗传疾病的发病机制提供了重要线索,短读长、高测量通量和低成本为个体化医学和精准治疗奠定了基础。此外,测序还广泛应用于农业、环境科学和生物工程等领域,为改良作物、保护环境和生产高效生物工艺提供了有力支持。
因此本文和大家分享DNA测序相关的知识以及测序技术的发展和测序的注意事项等。
DNA(脱氧核糖核酸)是遗传物质,存在于所有生物的细胞中,以双螺旋结构存在,由核苷酸组成,包括磷酸基团、脱氧核糖和四种氮含基(腺嘌呤A、胸腺嘧啶T、胞嘧啶C和鸟嘌呤G)。DNA负责存储遗传信息,指导蛋白质合成,并在细胞分裂时复制自身,确保遗传信息传递给子代。
不同物种的DNA在结构上非常相似,但在序列和组织上存在差异
人类DNA包含约30亿个碱基对,组成大约2万到2万5千个基因,这些基因分布在23对染色体上。人类DNA中的遗传信息决定了我们的外貌、生理功能和健康状况。人类的遗传多样性虽然存在,但所有人类的DNA序列大致相似,约有99.9%的相似度。个体基因组有 3-400 万个碱基对位置存在差异。这些变异可在单核苷酸多态性(SNP)中捕获 ,但也存在一些称为结构变异(SV) 的较大变异。

大多数病毒基因组有 10000 个 bp;某些植物的基因组长达数千亿个碱基对。细菌,通常具有较小的基因组,可以从几百万到几千万碱基对不等。细菌的DNA通常是单个环状染色体,而不是多条线性染色体。此外,许多细菌还含有质粒,这些是小型的DNA分子,可以在细菌之间进行转移,促进基因的水平传播,这是细菌适应环境和抗药性发展的重要机制。
总之,不同物种的DNA在功能上都是遗传信息的载体,但是在大小、形态和序列上存在差异,这些差异导致了物种之间的多样性。
不同物种个体基因组差异的产生有两个原因:
➼ 随机突变,在进化过程中发生,因为自然选择有利于某些表型。这些主要是由于细胞分裂期间 DNA 复制过程中的“错误”造成的。大多数突变都是有害的,会导致有害的表型变化并导致细胞死亡。有时,自然选择有利于某些突变,而这些突变会保留在种群中。
➼ 重组,发生在哺乳动物等高等生物的繁殖过程中。在重组过程中,亲本生物体传递给子代的遗传物质是来自亲本生物体的遗传物质的混合物。
DNA双链碱基互补
DNA 是双链的,并以双螺旋形式构建,其中核苷酸对作为螺旋的“横档”(因此称为“碱基对”)。腺嘌呤总是与胸腺嘧啶发生化学结合,而胞嘧啶总是与鸟嘌呤发生化学结合。换句话说,A 与 T互补,类似地 C 与 G 互补。AT 和 CG 对称为互补对。
DNA的结构如下所示:
图源:medlineplus
DNA序列通常以5’端(头部)到3’端(尾部)的方向展示或写入。当我们有一条 DNA 链时,由于知道互补对,所以可以推断出另一条链是第一条链的反向互补链。
为了获得反向互补,可以反转原始字符串中核苷酸的顺序,然后互补核苷酸(即,将 A 与 T 互换,将 C 与 G 互换)。
下图显示了 DNA 片段及其反向互补链的示例。
DNA 补体
DNA复制
DNA 是细胞复制的基础。当细胞进行细胞分裂(也称为有丝分裂)时,细胞核中的 DNA 会被复制,并通过下图所示的一系列步骤,一个亲代细胞产生两个相同的子细胞。
有丝分裂的图
图源:wikipedia
有丝分裂过程中涉及多种生物分子,我们在这里对有丝分裂过程进行了高度简化的解释。
在图中,我们从两条染色体开始:红色和蓝色。
首先,DNA 被复制,产生更熟悉的 X 形染色体。通过生物分子信号的复杂级联和细胞内重组,(现已复制的)染色体在细胞中部排列。对于每条染色体,两半被拉开,两个子细胞中的每一个都会收到原始染色体的副本。这会产生两个与原始亲本细胞在遗传上相同的子细胞。
DNA 复制是这张图中最重要的部分;这是用来进行测序的基础过程。DNA复制如下图所示:
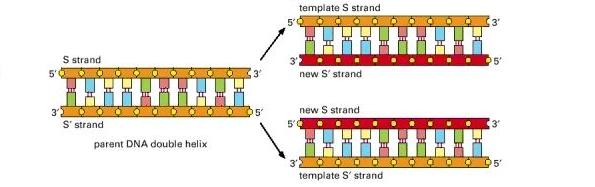
在 DNA 复制过程中,两条 DNA 链首先被解压缩,产生两条单链,每条链都充当复制的模板。然后将短 RNA 引物附着到 DNA 上的特定位点;引物中的碱基与位点中的碱基互补。酶促进(或“催化”)化学反应,而DNA 聚合酶是催化新核苷酸与延伸结合引物的模板 DNA 互补配对的酶。
DNA 聚合酶用来延伸链的核苷酸称为dNTP(脱氧核苷酸三磷酸)。从生物化学角度来看,它们与核苷酸略有不同,因为它们在 DNA 复制过程中更容易使用。对应于A、C、G和T的dNTP分别是dATP、dCTP、dGTP和dTTP。
获取DNA序列主要依赖于测序技术。常用的测序技术有桑格测序法和下一代测序法。下一章节会详细介绍。
图源:praxilabs
桑格测序法准确度高但通量低。下一代测序法代表了高通量测序技术,它实现了并行化操作,大大提高了序列通量,降低了测序成本和时间,因此适合进行整个基因组或transcript组的测序。这些技术使大规模、高精度的DNA测序分析成为可能。
DNA 测序方法的发展在 2000 年左右达到顶峰,主要基于四位研究人员的贡献。
➛
Allan Maxam 和 Walter Gilbert 在 70 年代开发了一种 DNA 测序的化学方法,其中末端用放射性磷标记的 DNA 片段经过碱基特异性化学切割,并通过凝胶电泳分离反应产物。
➛
1977 年,Frederick Sanger采用另一种方法,通过使用链终止双脱氧核苷酸类似物完善了测序方法,该类似物导致引物DNA合成的碱基特异性终止。在这种方法中,引物通常用放射性磷标记。
➛
Leroy Hood 与他的同事 Michael Hunkapiller 和 Lloyd Smith 在1986年通过使用荧光标记的双脱氧核苷酸将 Sanger 方法修改为更高的通量配置。这种方法可避免使用寿命有限的放射性化合物,而是使用稳定的荧光探针。此外,所有核酸碱基的分析可通过仅读取一个而不是四个电泳泳道来完成,并且读取过程可以自动化。
这种高通量配置用于第一个人类基因组的测序,该测序于 2003 年通过人类基因组计划完成,该计划历时 13 年。

由于方法的改进和自动化,2008 年,另一个人类基因组在 5 个月的时间内完成了测序。第一份人类基因组草图的完成只是现代 DNA 测序时代的开始,它带来了更多的发明和新的、先进的高通量 DNA 测序策略,即所谓的下一代测序 (NGS)。
NGS 策略的发展正在满足我们对测序通量和成本的需求,从而在基因组研究中实现多种当前和未来的应用。这些先进方法需要开发新的生物信息学工具,作为分析过程中产生的大量数据的必要先决条件。
第一代测序——桑格测序
Fred Sanger 及其同事开发了一种基于放射性标记部分消化片段检测的相关技术。
著名的桑格测序起源于 20 世纪 70 年代末,当时桑格开发了一种基于凝胶的方法,将 DNA 聚合酶与标准核苷酸和链终止核苷酸 (ddNTP) 的混合物结合起来。将 dNTPS 与 ddNTP 混合会导致 PCR 期间测序反应随机提前终止。四个反应并行进行,每个反应包含一种版本的链终止核苷酸。使用凝胶电泳可视化该过程使得能够逐个碱基读取序列。在当时,这项技术是革命性的。它能够对 500-1,000bp 片段进行测序。
图源:praxilabs
桑格方法的一种变体——加减法,由桑格和艾伦·科尔森开发,于1977年获得了第一个DNA基因组序列,即噬菌体φX174。
图源:pixels
两年后,艾伦·麦克萨姆和沃尔特·吉尔伯特发表了他们的化学裂解技术,该技术成为第一个广泛采用的 DNA 测序方法。

到了 20 世纪 80 年代,桑格最初的方法已经自动化(毛细管电泳)。大块凝胶被更细的丙烯酸毛细管取代,结果可以在电泳图上查看。这项技术对于 2003 年人类基因组计划的完成至关重要。尽管如此,即使在人类基因组计划之后,毛细管电泳的成本仍然过高,无法实现大规模测序项目。
到 2000 年代中期,人们做出了一些努力来降低测序成本。世界各地的实验室正在测试用于更高通量筛选的新方法和技术。
第二代测序技术
第二代测序,也称为下一代测序(NGS)。简单来说,二代测序是依靠PCR文库构建和激光探针荧光信号读取的短读长测序。
目前最常见的平台有Illumina和华大基因(BGI)。
Illumina测序平台
由 illumina 等公司开发的第二代 NGS 技术可分为两大类:杂交测序或合成测序。
华大测序平台
华大基因测序化学方法被称为组合探针锚定合成(cPAS)。它采用Phi 29 DNA聚合酶进行滚动圆环复制,合成一条长的单链DNA,自组装成约300纳米大小的纳米球。然后进行鉴定以确定 DNA 序列。
随着大规模双脱氧测序技术的进步,一项新技术的出现奠定了下一代测序(NGS)技术的基础。这项名为焦磷酸测序的方法,利用DNA合成过程中焦磷酸盐产生的光信号来确定核苷酸序列。在这个过程中,模板DNA被固定在一个固相表面上,随着每个核苷酸的加入,通过检测焦磷酸释放的光信号来推断DNA的序列。此技术后续还引入了珠子,以便更有效地附着DNA分子。
焦磷酸测序技术被454 Life Sciences公司所开发,并最终被罗氏公司收购,成为市场上第一个取得重大成功的商业化NGS平台。
乳液PCR
在这一平台中,DNA文库通过油包水乳液PCR技术附着在微小珠子上。在测序过程中,当较小的珠联酶和dNTPs被引入到反应板上时,便可进行焦磷酸测序。这种高度并行化的方法显著提高了测序的吞吐量,实现了数量级的提升。
桥式放大
继454测序技术取得成功之后,许多新的并行测序技术相继出现。其中最显著的是Solexa测序技术,该技术后来被illumina公司收购。
Illumina测序平台因此成为了第一个实现商业化的高通量并行测序技术。

其他
随着时间的推移,新技术不断涌现,其中包括:
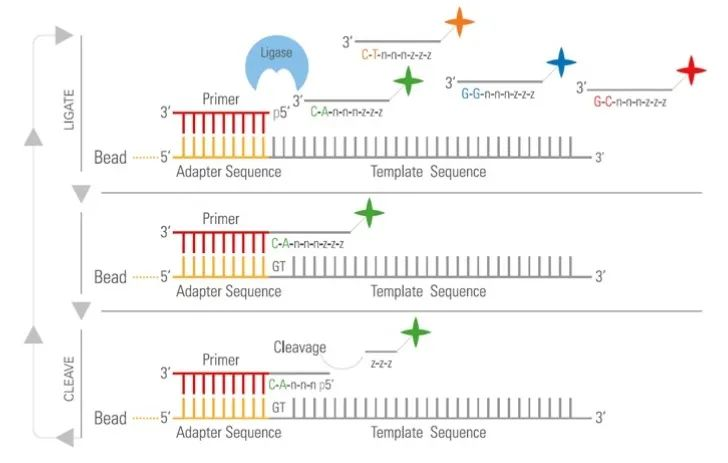
图源:slideserve
这些创新技术已经成为下一代测序(NGS)技术领域的一部分。NGS平台目前是主流的测序技术,它们可以以相对较低的成本进行高通量的测序工作。然而,这些平台的读长通常有限,一般产生的读长在50到500碱基对(bp)之间。
本文我们主要介绍Illumina和华大这两个平台的测序原理。其他的简单说明一下。
Illumina测序平台介绍
Illumina 的首个测序平台是通过收购Solexa公司获得的,被命名为基因组分析仪,并于2007年开始商业化运作。这台设备能够在每个测序通道中对600万个扩增的DNA片段进行测序,最初每个片段的读取长度大约是30个碱基。Illumina 不久后提升了这一读取长度,增至100多个碱基对。同时,流动池中扩增片段的数量也得到了提升,使得基因组分析仪的输出能力达到了80吉字节的碱基信息。
注:吉字节(GigaByte),又称千兆字节,是计算机存储容量单位,简称GB。
2010年,Illumina 推出了其第二代NGS设备——HiSeq。这款设备配备了两个流动池:
紧接着,Illumina 又发布了HiSeq X10,该设备通过使用图案化的流动池凹坑(代替了传统的随机扩增簇),进一步提高了可分析片段的数量。
目前,Illumina 提供了多种测序设备,包括NextSeq 和 NovaSeq 系列,以及适用于不同规模需求的台式测序仪,如iSeq100和 MiniSeq。

NextSeq
NextSeq 500 于 2014 年推出,采用两种染料测序技术,而不是其前代产品使用的四种染料测序技术。仅拍摄红色和绿色图像,从而显着缩短周期和数据处理时间。该仪器能够在大约 30 小时的运行时间内读取4亿个碱基对。
NextSeq 1000 和 2000 机器于2020年发布,旨在通过提供机载信息学和基于云的技术来简化工作流程。P3 流动池扩展了 NextSeq 2000 仪器的范围,在单次测序运行中提供 11 亿次读取。
NovaSeq6000
NovaSeq 6000于2017年发布。它能够运行三种不同的芯片,并且可以生成100 GB的序列输出,价格仅为375美元——这个价格仅适用于测序,不包括DNA分离、文库制备、测序分析或数据贮存。
本质上,该机器每次运行能够对多达 48 个完整人类基因组进行测序,这可能需要长达 44 小时。其他关键应用包括单细胞分析、转录组测序和宏基因组分析。

HiSeq X 系列
HiSeq X Ten 测序仪是一种高性能的测序系统,它能够在单次运行中产生高达16 Tb的序列输出。使用该系统,可以以不到1000美元的价格对人类基因组进行30倍或更高倍数的测序,并且每年可以提供超过18,000个人类基因组的测序数据。每个流动池可以生成多达520亿次的读取,最长运行时间为48小时。
该系统具有超越人类物种的全基因组测序能力,并且还可以用于全外显子组测序、转录组测序、单细胞分析和多组学研究。
华大测序平台(BGI) 介绍
华大基因集团成立于1999年、参与人类基因组计划的中国公司。华大基因于2012年收购了Complete Genomics,其产品由子公司(华大智造)销售。
DNBSEQ-T7
DNBSEQ-T7于2019年推出,旨在支持健康项目和临床研究的一系列大规模测序应用。据报道,与百万基因组整体解决方案软件和硬件一起,DNBSEQ-T7 每年可以对多达 800,000 个样本进行测序。

硬件解决方案包括自动化文库制备系统,这意味着测序机可以24小时运行,无需人工干预,每天可以完成60个人类全基因组测序。其商业化预计将把个人全基因组测序的成本降低至 500 美元以下,从而改变测序格局。
华大基因测序化学
BGI的测序化学方法被称为组合探针锚定合成(cPAS)。它采用Phi 29 DNA聚合酶进行滚动圆环复制,合成一条长的单链DNA,自组装成约300纳米大小的纳米球。荧光探针被结合在其中,纳米球被连接到硅片流动池上,选择性地与带正电的材料高度有序地结合。然后,荧光发射被成像和测量,以记录碱基位置。
与所有短读取测序方法一样,BGI平台主要缺点是无法获得长的DNA序列。然而,基于cPAS的测序的一个重要优势是Phi 29 DNA聚合酶的高准确性,确保了环状模板的准确扩增。此外,由于DNA纳米球在流动池上保持不动,它们不会产生光学重复,并且不会干扰相邻的DNA。
DNBSEQ-G99(G99)
DNBSEQ-G99(以下简称“G99”)基因测序仪采用的是基于聚合酶链式反应(PCR)原理的测序技术。测序过程中,首先使用特定的引物引导 DNA 序列进行体外扩增,然后添加一种含有测序所需的四种不同颜色的 dNTP(脱氧核苷酸)和荧光标记物。当引物与待测序列结合后,聚合酶开始合成新的链,同时荧光标记物被激活并发出不同颜色的荧光。通过记录这些荧光信号,并使用计算机进行数据分析和解码,最终确定每个碱基的序列。

G99突破性地实现了12小时可完成PE150测序,从用户需求出发,在提供高质量的测序数据的同时,做到快速、简单、灵活,能为测序工作带来更好的体验,应用场景大大扩展了。
而且DNBSEQ-G99获批国家药品监督管理局(NMPA)医疗器械注册证(国械注准20233221289)。此次获批意味着,中小通量测序仪中的“速度王者”DNBSEQ-G99被准许在国内市场应用于临床,将能够充分发挥其快速、灵活的优势,服务于临床方向的应用需求。
第三代测序技术
第三代测序技术的原理主要基于单分子测序或合成测序方法,通过直接读取DNA分子的序列来进行测序。
单分子测序:通过将DNA固定在表面上,并使用荧光染料或其它探针进行测序。
单分子实时测序(SMRT):使用PacBio公司的SMRT技术,通过监测DNA聚合酶在DNA模板上的荧光信号来进行测序。
纳米孔测序(Nanopore):使用Oxford Nanopore Technologies(ONT)的纳米孔测序技术,通过将DNA分子通过纳米孔,测量通过纳米孔的电流变化来进行测序。

合成测序:通过在反应体系中逐步合成DNA序列,并使用荧光标记的核苷酸来标记每个碱基。第三代测序技术通常具有较长的读取长度,可以读取数千到数百万个碱基。
第三代测序技术的不断发展和改进,为基因组学研究提供了更多的可能性,可以更好地解析复杂的基因组结构和功能。适用于长片段的测序,如全基因组测序、长读段转录组测序、甲基化测序等。然而,第三代测序技术也面临一些挑战,如测序错误率、数据处理和分析等方面的问题,需要进一步的研究和改进。
市场上的其他三代测序平台:
MinION:MinION设备是一种便携式的纳米孔测序仪器,可以实现实时测序,并且具有较小的体积和较低的成本。
GridION:GridION设备是一种高通量的纳米孔测序仪器,可以同时进行多个样品的测序。
PromethION:PromethION设备是一种高产量的纳米孔测序仪器,可以进行大规模的基因组测序。
此外国内目前也有多家公司已推出或正在开发三代测序仪,包括真迈生物,齐碳科技等。
测序将继续变得越来越高效和经济实惠,彻底改变与基因组学相关的多个领域。目前,所有高通量测序(NGS )方法都需要文库制备。该方案发生在 DNA 片段化之后,其中接头连接到每个片段的末端。接下来通常是 DNA 扩增步骤,以产生一个文库,然后可以通过 NGS 平台进行测序。
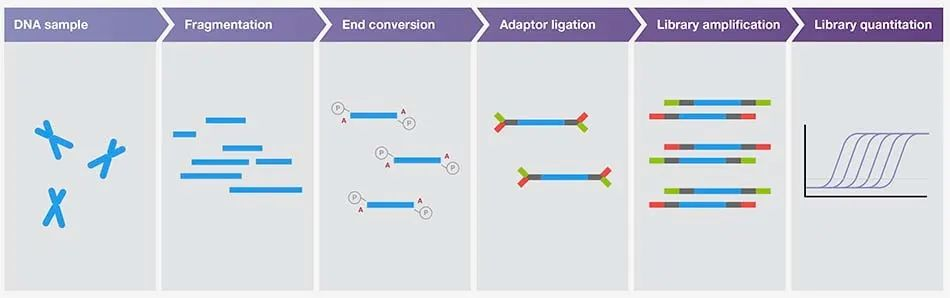
➦1
样品制备的本质是将生物样品中的核酸混合物转化为不同类型的文库,以准备进行NGS技术所需的测序步骤。如果未正确遵循方案,测序将会受到影响。每个准备步骤都是基础性的,并且根据样本和NGS平台的类型有不同的考虑因素。因此,在开始实验之前,考虑如何执行最有效的方案以确保最高质量的结果非常重要。
样品制备的一般步骤如下:
步骤1:提取遗传物质
这是每个样品制备方案的第一步。从各种生物样品中提取核酸(DNA 或 RNA)。
步骤2:文库准备
生成文库需要一系列步骤,最终目标是将提取的核酸转换成适合所选测序技术的格式。这是通过将目标序列片段化至所需长度,然后将特定的接头序列连接到这些目标片段的末端来完成的。
适配器还可以包括条形码,识别特定样品并允许多重分析。片段化可以通过物理或酶促方法完成。
步骤3:放大
这是一个可选步骤,但通常也是必需的。这取决于 NGS 的应用和样本量。扩增对于获得足够的覆盖范围以对含有少量起始材料的样品进行可靠测序至关重要。聚合酶链式反应(PCR) 是增加 DNA 量的常用方法。有关可实现小样本核酸检测的 PCR 方法出现的更多信息。
步骤4:纯化和质量控制
此步骤是必要的,以去除可能阻碍测序的任何不需要的材料。一些NGS平台可能对尺寸要求较窄,因此丢弃太大或太小的片段可以提高测序效率。最佳文库大小由测序应用决定。这种“清理”通常通过基于磁珠的清理或琼脂糖凝胶来完成。
质量控制是进行测序之前的最后一个过程。确认 DNA 的质量和数量可以提高测序数据的可信度。后续的实验既耗时又昂贵,因此需要严格的质量控制步骤以确保所有样品都适合其应用。
➦2
挑战 1
许多样本是从有限数量的样本或甚至单个细胞中提取的。它们本身并不能提供足够的遗传物质,因此需要进行 PCR。然而,该扩增步骤很容易给样品引入偏差。PCR 重复是指存在完全相同的 DNA 片段的多个拷贝。太多的 PCR 重复会导致实验的测序覆盖率不均匀。
解决方案 1:消除所有偏差来源有些不可能,但了解偏差发生的位置并采取所有实际步骤将其最小化非常重要。高 PCR 重复率表明文库制备需要进行一些修改,可能需要提高 NGS 文库的复杂性。
许多程序都可以删除 PCR 重复项,最常用的是 Picard MarkDuplicates 和 SAMTools。此外,特定的PCR 酶已被证明可以最大限度地减少扩增偏差。最终,文库制备的目标是最大限度地提高样品的复杂性,并最大限度地减少扩增造成的偏差。
挑战 2
建库效率低下是样品制备过程中面临的问题。具有正确适配器的片段比例较低反映了这一点。其后果是获得的测序数据量减少,嵌合片段数量增加。嵌合读数源自基因组中彼此不相邻的部分,并且是测序期间错误的来源。
解决方案 2:据报道,PCR 产物的有效A 加尾可防止嵌合体形成,该程序是通用的,可应用于多种不同的文库构建技术。此外,链分割伪影读数 (SSAR) 已被建议减少样本中嵌合伪影的数量,并且嵌合体检测程序可用于过滤原始序列,以实现仅 1% 的总体嵌合率。
挑战 3
样品污染是一个固有的问题,因为单独的文库通常是并行制备的。最可能的主要污染源是预扩增,这是一种在 PCR 之前增加核苷酸序列量的方法。
解决方案 3:可以通过质控,阴性对照,设置重复等步骤识别污染,确保在样品制备过程中使用无菌技术和无菌实验条件,以防止外源性污染的引入。
此外使用独特的条码和标签对样品进行标识(谷禾所有样本全程唯一条形码溯源识别管理),以避免混淆和交叉污染。最后,做好定期清洁和消毒:定期清洁和消毒实验室设备和工作区域,以减少污染的积累和传播。
挑战 4
文库制备的巨大成本主要归因于实验室设备、需要经过培训的人员和试剂成本。
解决方案 4:通过优化实验步骤和条件,可以减少试剂的使用量和浪费,从而降低成本。确保实验室人员接受适当的培训和技术支持,以提高实验的效率和准确性。与其他实验室或研究团队合作,共享设备和资源,共同承担成本和实验负担。随着自动化技术变得越来越流行,样品制备的准确性和效率可能会提高。
什么是碱基平衡?
测序中一个不可忽视的原则就是碱基平衡,是指测序过程中,每个循环中A、C、G和T四种碱基,比较均匀地存在。需要兼顾的平衡度与复杂度。在测序过程中,保持碱基平衡是非常重要的,可以确保测序结果的准确性和可靠性。
什么是碱基不平衡文库?
就是扩增子产生的文库,扩增子的特点是有特定的起始位点的。反应到测序图像上,就会呈现一张照片特别亮,光点很多,而其他三张照片就特别暗。这时软件做空间上比对就比较难。结果是判断的可靠性比较差,导致对于碱基的判读就会出现错误,从而导致测序质量值大幅度下降。一般添加诸如基因组DNA文库,或掺入大量的平衡碱基文库,包括phix文库等。同时,也可以尽量多掺入不同类型的扩增子文库。
此外,碱基平衡还涉及到检测和纠正测序过程中的碱基偏差。在测序过程中,可能会出现碱基的插入、缺失或错误,这些错误会影响测序结果的准确性。为了纠正这些错误,开发了各种生物信息学工具和算法,例如质量控制和碱基校正等。
文库长度含两侧测序接头和插入目标片段,整个文库的长度范围不能过宽,一般建议在250bp-450bp之间比较好,超过600bp以上就会造成一些不利影响。
过长的文库长度会降低测序效率
在Illumina测序等高通量测序平台上,测序片段长度会影响测序的质量和效率。过长的文库长度会增加测序过程中的错误率,并且会导致测序片段的读取长度变短。这会降低测序的可靠性和准确性,影响后续的生物信息学分析和数据解读。但是如果文库片段过短的话,该短片段测序到后期,就是要测接头序列了,有的时候连接头序列都测完了,那就没有信号了,后续会读取一些假信号,降低测序质量值。
过长的文库长度会降低簇密度
簇密度是基于单次只测一个碱基的边合成边测序原理,要求对各个分子簇的反应时间要求一致。也就是各个分子簇必须同时进行反应。理想状态当然是如此,但是实际PCR反应过程中,各个分子的反应时间还是不尽相同的(一般体系和酶要控制好)。因此,会产生有的分子簇内的分子反应的快,有的慢的情况过长的文库长度会降低簇密度。在Illumina测序中,DNA片段会被固定在测序芯片上的聚合酶链反应(PCR)产物中,形成簇。过长的文库长度会导致PCR扩增效率降低,从而降低簇密度。低簇密度会减少每个簇中的测序片段数量,进而降低测序的覆盖度和深度,影响后续的数据分析和解读。
过长的文库长度可能导致碱基偏移
在测序过程中,由于DNA聚合酶的滑动等原因,长片段的文库容易出现碱基偏移的情况。
数据量(yield)
数据量指一次测序所获得的PF数据的总量。注意,是PF数据(PF数据是指通过滤波后的有效测序数据,即通过质量控制筛选后的测序片段),而不是原始数据。数据量当然越多越好,实际成绩与测序仪型号有关,不同的机器,产量不一样。
PF数据的总量是衡量测序深度和测序质量的一个重要指标。较高的PF数据总量表示测序过程中得到了更多的有效测序片段,可以提供更高的测序覆盖度和深度,从而提高后续数据分析的可靠性和准确性。
Q30
Q30是指在测序过程中,质量值(Quality Value,QV)大于或等于30的碱基。质量值是根据测序仪器对每个碱基的测量结果和信号峰值计算得出的,用来表示该碱基的质量好坏。Q30值越高,表示测序数据中高质量的碱基比例越高。
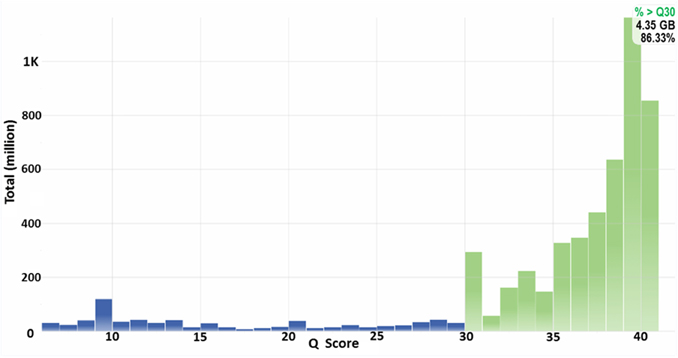
by:Alexander William Eastman
需要注意的是,Q30的大小与测序片段(read)的读长有关。如果读长较长,即测序片段包含的碱基数较多,那么要求每个碱基的质量值都达到或超过30就更加困难,因此平均%Q30可能会降低。相反,如果读长较短,即测序片段包含的碱基数较少,那么要求每个碱基的质量值达到或超过30就相对容易,平均%Q30可能会提高。
比对率(mappingrate)
将测序数据与参考序列(reference)进行比对,是测序数据分析中的一项重要步骤。比对率是指在比对过程中,测序数据与参考序列完全一致的碱基占测序数据总碱基数的比例。比对率越高,表示测序数据的准确性和可靠性越高。在细菌16S测序中,可以根据具体的需求选择适合的比对工具。
常用的比对工具是基于Smith-Waterman算法的BLAST(Basic Local Alignment Search Tool)和基于Burrows-Wheeler变换的Bowtie、BWA等工具。高比对率是测序数据质量好的重要指标之一。它表示测序数据的准确性和可靠性较高,能够提供更准确的基因组信息和变异位点等重要信息(在谷禾16s测序中,尤其粪便样本约超过 70%能比对到种)。在后续的数据分析和解读中,高比对率的测序数据更有助于准确地进行变异检测、基因表达分析、功能分析等。
需要注意的是,比对率受到多种因素的影响,包括测序数据质量、参考序列的准确性、数据库以及比对算法的选择等。在进行测序数据分析时,需要综合考虑比对率、测序数据质量和其他相关指标,以获得准确可靠的分析结果。
覆盖度(coverage)
由于测序数据的生成过程中存在一些技术和生物学上的随机性,导致不同区域的测序数据的覆盖深度(coverage depth)是不一样的。
覆盖深度是指在某个特定位置的测序数据的读段数目或测序碱基数目。覆盖深度越高,表示该位置的测序数据越丰富,测序结果的准确性和可靠性越高。
需要注意的是,覆盖深度的均匀性和高低受到多种因素的影响,包括测序深度、测序技术、样本质量等。
重复率(duplicationrate)
在二代测序文库的构建过程中,除了无PCR流程(PCR-free approach),其他方法都需要进行PCR扩增。PCR扩增会导致染色体的不同区域放大程度不一致,有部分序列被过度放大。这是一种人为引入的偏差。重复率与文库构建试剂的质量有关,对于人类全基因组测序来说,通常<10%。
捕获率(capturerate)
杂交捕获建库是通过探针杂交捕获来从基因组文库中富集相应序列的,探针杂交捕获存在着捕获效率高低的问题,因此考察、评价这一步骤成败、好坏的参数就是捕获率,越高越好。捕获率与所用的捕获试剂有关,不同的试剂,捕获率不同。
高通量测序操作包括样品准备、文库构建、PCR扩增、测序仪器运行等。只有按照标准SOP规范操作,才能保证实验的准确性和可重复性。新测序仪平台和技术的不断出现和改进,使得高通量测序无论通量、质量、速度和成本都在快速进步,高通量测序的应用范围也大大得到拓展,不久的将来有望以低成本随时随地的开展高通量测序应用。

谷禾健康
你敢相信吗?从粪便中提取出具有强烈粪臭味的物质,当用酒精稀释上千倍后,脱胎换骨变成了一种香味。这就是一种吲哚衍生物——3-甲基吲哚(又名粪臭素)
吲哚,是所有花香类原精的关键成分,这种物质在低剂量1-3%浓度时,具有放松的作用,协同苯基酯,能强效抗痉挛,增添感官愉悦和魅力。然而,如果简单将吲哚等同于香水,未免太少看它的功效。
在谷禾特定肠道菌群检测报告中炎症部分或毒素部分有吲哚这个指标。
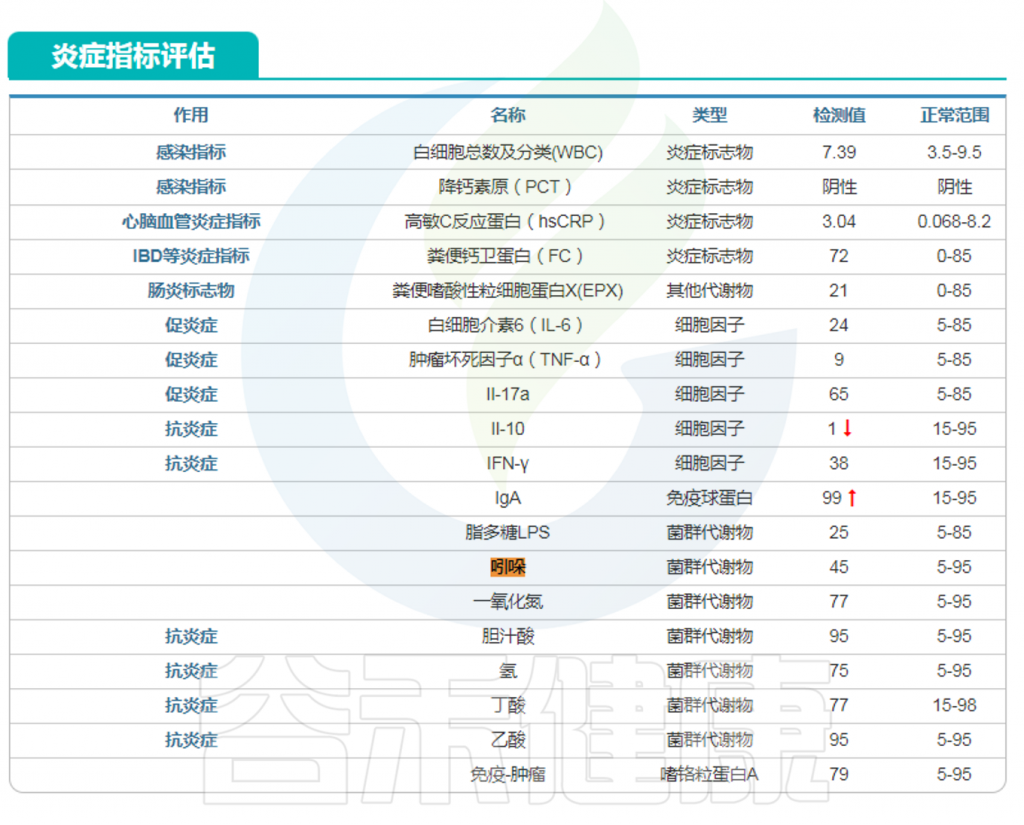
<来源:谷禾肠道菌群检测数据库>

<来源:谷禾肠道菌群检测数据库>
很多人可能对与菌群和健康相关的血清素,色氨酸,脂多糖,组胺等指标比较熟悉,但是对吲哚还是比较陌生,实际上吲哚与肠道微生物群和人类健康和生活关系密切,例如,吲哚衍生物可以影响胃肠道的蠕动,可能会影响便秘或腹泻等状况;吲哚及其衍生物已被证明可以加强肠道屏障功能,有助于防止病原体和有害物质进入血液。
事实上,由吲哚衍生而来的各种化学物质,对人体神经调节和抗病原微生物,有极为积极的作用。在人体中存在着许多这样神奇的吲哚类物质。内源性吲哚及其衍生物是源自肠道微生物群的色氨酸代谢物,具有一系列生物活性。
最近的研究表明,吲哚有助于维持人体肠道的生物屏障,主要通过激活AhR和PXR受体来发挥抗炎活性,从而影响免疫系统的功能,显著改善肠道健康(炎症性肠病、出血性结肠炎、癌症结直肠癌),并进一步促进人类健康(糖尿病、中枢系统炎症和血管调节)。
作为细胞间信号分子,吲哚还控制细菌生理学的各个方面,例如产吲哚细菌的孢子形成、质粒稳定性、耐药性、生物膜形成和毒力。相比之下,许多不产生吲哚的细菌、植物和动物产生多种加氧酶,可能会干扰吲哚信号传导。
吲哚还是身体神经系统与内分泌系统正常与否的风向标。所谓的体臭,其实就是人体内吲哚含量增高的表现。吲哚在细菌生理学、生态平衡以及人体健康中发挥着重要作用。
然而,吲哚的毒性影响也不容忽视。过量的吲哚会对情绪和行为产生不利影响,硫酸吲哚酚是一种吲哚衍生物,具有肾毒性和心血管毒性。羟吲哚还可能降低神经元兴奋性。
吲哚与肠道微生物群之间的相互作用以及吲哚对人类健康的影响是一把双刃剑。本文主要讲述了内源性吲哚的生物合成、吲哚对肠道功能及微生物群的影响,为吲哚与疾病的相关性提供了科学依据。
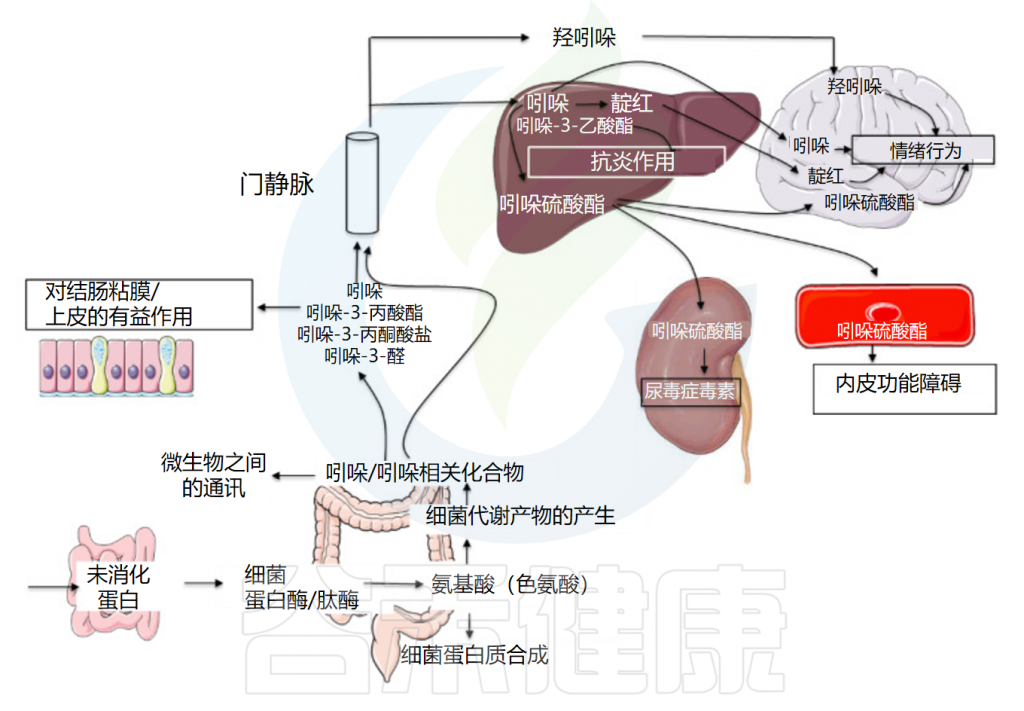
Tennoune N,et al., Microorganisms.2022
吲哚(Indole)是一种有机化合物,是吡咯与苯并联的化合物,又称苯并吡咯。吲哚及其同系物和衍生物广泛存在于自然界,主要存在于天然花油,如茉莉花、苦橙花、水仙花、香罗兰等。
而内源性吲哚及其衍生物是来源于肠道菌群的色氨酸代谢物,具有一系列生物活性。内源性吲哚类物质包括吲哚本身以及吲哚的代谢产物,如吲哚-3-甲酸、吲哚-3-丙酸、硫酸吲哚酚等。
吲哚相关化合物的结构
Lee JH, Lee J.FEMS Microbiol Rev.2010
肠道菌群产生吲哚类物质
细菌蛋白酶和肽酶从可用的膳食中释放氨基酸,包括色氨酸。而色氨酸是吲哚的主要前体。大部分色氨酸在小肠中被吸收,约4%~6%的色氨酸在结肠中被肠道菌群分解代谢为吲哚。
色氨酸作为一种必需氨基酸,主要从富含蛋白质的食物中获取,是唯一含有吲哚的氨基酸。
肠道中的色氨酸通过三个主要途径进行代谢:1) 犬尿氨酸途径(摄入的色氨酸的95%);2)细菌色氨酸代谢(4-6%);
3) 血清素途径 (1-2%)
✦ 肠道菌群主要通过色氨酸酶将色氨酸转化为吲哚
肠道细菌主要通过色氨酸酶(TnaA)的作用将色氨酸转化为吲哚;色氨酸酶TnaA可逆地将色氨酸降解为吲哚、丙酮酸和氨。
值得注意的是,在哺乳动物中,吲哚完全来源于细菌代谢活动,因为宿主细胞不具有产生这种化合物的代谢能力。
色氨酸也可以通过肠道微生物群直接或间接转化为几种吲哚相关化合物,包括吲哚-3-丙酮酸、吲哚-3-乳酸、吲哚-3-丙酸、吲哚-3-乙酰胺、吲哚-3-丙烯酸、吲哚乙醛、吲哚-3-乙酸、3-甲基吲哚(粪臭素)等。
✦ 内源性吲哚产生的机制
大肠杆菌(E. coli)是研究最广泛的吲哚合成生物体,它可以使用多种机制(抑制、转录衰减和反馈抑制)来调节色氨酸代谢中的色氨酸操纵子(Trp operon)和tna操纵子的表达。
在色氨酸缺乏的条件下, trp操纵子的表达升高,而由TnaC(24个氨基酸前导肽,以前称为TnaL)、TnaA(色氨酸酶)和TnaB(通透酶)组成的tna操纵子的表达受到抑制,因为转录终止因子(Rho)依赖性终止发生在tna操纵子中。
结果,当色氨酸水平较低时,TnaA和TnaB的表达以及吲哚的产生受到抑制。在富含色氨酸的条件下,转录终止因子依赖性转录终止被消除,因此吲哚产量增加。
因此,细胞外色氨酸和其他氨基酸直接影响吲哚的产生。
// 拓展:渗透酶通过转运也会影响吲哚水平
此外,三种渗透酶(Mtr、TnaB和AroP)在不同环境的色氨酸转运中发挥不同的作用,因此可以直接影响吲哚的水平。
Mtr通透酶主要负责转运吲哚,而TnaB通透酶对于色氨酸的摄取至关重要。尽管色氨酸的生产成本高昂,但细胞仍然利用色氨酸途径大量生产和分泌吲哚。
由色氨酸产生的吲哚可以通过细胞膜蛋白进行转运。例如,大肠杆菌和霍乱弧菌在丰富的培养基中可以分泌高达0.6 mM的吲哚。
在大肠杆菌中,外排蛋白AcrEF部分负责吲哚的输出,因为AcrEF突变体的吲哚排泄低于其野生型菌株。Mtr通透酶主要负责输入吲哚,因为吲哚不被mtr突变体吸收。
然而,最近有人提出,吲哚由于其疏水性,可能可以直接扩散通过细胞膜。因此,有必要清楚地了解吲哚的进出口情况。
大肠杆菌中吲哚生物合成和吲哚信号传导
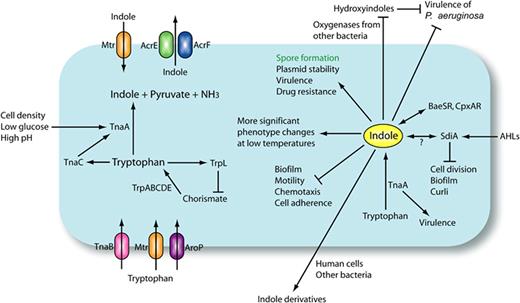
Lee JH, Lee J.FEMS Microbiol Rev.2010
人体粪便中吲哚的浓度较高,通常范围为0.25毫摩尔浓度~1.1毫摩尔浓度,但可能由于人群之间肠道微生物群组成的差异以及膳食蛋白质消耗水平不同而存在差异区别。
注:由于吲哚似乎是通过结肠上皮吸收的,结肠中吲哚的浓度可能高于粪便中记录的浓度,远端大肠的浓度高于近端部分的浓度。
尽管大多数生物体都含有色氨酸代谢途径,但只有能编码tnaA的细菌才能合成吲哚。表达色氨酸酶(tnaA)的细菌,例如大肠杆菌(E. coli)、梭菌属(Clostridium spp.)、拟杆菌属(Bacteroides spp.)和普通变形杆菌(Proteus Vulgaris),可催化色氨酸直接转化为吲哚。
✦ 梭状芽孢杆菌代谢产生吲哚-3-丙酸
已经证明吲哚-3-丙酸是由梭状芽孢杆菌(Clostridium sporogenes)产生的。粪臭素(3-甲基吲哚)是由梭菌属和拟杆菌属产生的。
此外,产孢梭菌(C. sporogene)可以将色氨酸转化为吲哚-3-丙酮酸(IPyA),并在吲哚-3-丙酮酸脱羧酶的催化下产生吲哚-3-乙醛(IAAld)。然后,IAAld 被厚壁菌门、变形菌门、拟杆菌门和放线菌门的成员脱羧,产生吲哚乙酸 (IAA)。
✦ 一些病原菌也可以产生吲哚
色氨酸代谢很复杂,许多菌株参与吲哚衍生物的制造。总的来说,数据支持迄今为止有至少85种细菌已被证明可以产生吲哚。
这些细菌包括许多病原菌,例如创伤弧菌、流感嗜血杆菌、多杀巴氏杆菌(pasteurella multocida)、四种志贺氏菌菌株、植物克雷伯菌(Klebsiella planticola)和普通变形杆菌。值得注意的是,一些革兰氏阳性菌株,包括B.alvei和粪肠球菌(E.faecalis)也产生吲哚。
✦ 一些拥有tnaA基因的细菌却无法产生吲哚
有趣的是,一些细菌,如杀鲑气单胞菌(Aeromonas salmonicida)、宋氏志贺氏菌(Shigella sonnei)、克氏耶尔森氏菌(Yersinia kristensenii)和Seudovibrio spp.,已经失去了合成吲哚的能力,尽管这些菌株的染色体上有一个tnaA基因同源物。
例如,与正常志贺氏菌菌株不同,一些在tna操纵子中具有点突变、插入或缺失的志贺氏菌不产生吲哚,这可能是由于某些适应性优势。虽然只是推测,但一些个体可能通过利用当地细菌群体的信号来避免产生吲哚的成本。
其他可以产生吲哚的细菌:
Alkaliphilus metalliredigens
Bacillus alvei
Clostridium novyi
Clostridium limosum
Clostridium tetani
Corynebacterium acnes
Desulfitobacterium hafniense
Nocardioidessp.
Oribacterium sinus
Propionibacterium acnes
Enterococcus faecalis
Symbiobacterium thermophilum
Aeromonas hydrophila
Aeromonas liquefaciens
Aeromonas punctata
Bacteroides thetaiotaomicron
Bacteroides sp.
Brachyspira hyodysenteriae
Burkholderiasp.
Chromobacterium violaceum
Chryseobacterium gleum
Citrobactersp.
Citrobacter intermedius
Citrobacter koseri
Desulfovibrio vulgaris
Edwardsiella tarda
Enterobacter liquefaciens
Escherichia coli
Escherichia fergusonii
Flavobacteria bacterium
Flavobacteriumsp.
Fusobacterium nucleatum
Fusobacterium polymorphum
Haemophilus influenzae
Haemophilus somnus
Haloarcula marismortui
Halogeometricum borinquense
Hyphomonas neptunium
Klebsiella ornithinolytica
Klebsiella planticola
Micrococcus aerogenes
Morganella morganii
Paracolobactrum coliforme
Pasturella multocida
Pasturella pneumotropica
Photobacterium harveyi
Porphyromonas asaccharolytica
Porphyromonas endodontalis
Porphyromonas gingivalis
Prevotella intermedia
Proteus inconstans
Proteus vulgaris
Providencia alcalifaciens
Providencia rettgeri
Providencia rustigianii
Providencia stuartii
Rhizobium leguminosarumbv.trifolii
Saccharomonospora viridis
Shigella boydii
Shigella dysenteriae
Shigella flexneri
Sphaerophorus varius
Stigmatella aurantiaca
Treponema denticola
Vibrio alginolyticus
Vibrio cholerae
Vibrio fluvialis
Vibrio harveyi
Vibrio hollisae
Vibrio mediterranei
Vibrio nigripulchritudo
Vibrio orientalis
Vibrio parahaemolyticus
Vibrio splendidus
Vibrio tapetis
Vibrio tubiashii
Yersinia frederiksenii
Yersinia intermedia
吲哚类物质的生成可能受到环境因素的严重影响,例如膳食成分、肠道微生物群、碳源、温度、pH值以及生理状态。
★ 肠道微生物群和饮食对吲哚生成有重要影响
吲哚类物质是肠道微生物群产生的膳食色氨酸的代谢产物。因此,肠道微生物组成以及饮食模式的变化会影响其产生。
研究发现吲哚-3-丙酸(IPA)与肠道微生物群的α多样性呈正相关,肠道微生物组的组成可以解释大约20%的IPA循环水平的变化。
高纤维和富含多酚的饮食可以提高吲哚含量
多项研究发现,吲哚-3-丙酸的循环水平与纤维摄入量相关,这可能是通过肠道微生物群的变化来解释的。
此外,富含多酚的饮食会导致肾功能正常受试者的血清IPA显著增加,但肾功能受损的受试者不会显著增加。与此相一致的是,饲喂菊粉60天的猪的盲肠和结肠中微生物组组成的β多样性显著增加,同时IPA含量也显著增加。
地中海饮食会导致吲哚-3-丙酸水平升高
不同的饮食结构也会改变吲哚的水平。例如,地中海饮食仅治疗4天后就导致吲哚-3-丙酸水平升高,而快餐(即薯条和汉堡)则引起相反的结果。
此外,正常饮食喂养的同窝小鼠的血浆IPA水平显著高于生酮饮食喂养的小鼠。一项针对117名超重成年人的研究发现,摄入炸肉会降低肠道微生物群落的丰富度,并导致IPA浓度下降。
发酵食品可能会降低吲哚含量
通过对14名健康男性进行随机交叉研究设计,探讨了发酵乳制品对人类血清代谢组的影响。与牛奶组相比,酸奶摄入组餐后血中吲哚-3-丙酸和吲哚乙酸浓度较低。
同样,C反应蛋白水平轻度升高的健康超重男性在接受抗炎膳食混合物后,饮用500mL餐后奶昔(由300mL蛋奶冻、150mL奶油干酪和50mL鲜奶油组成)。受试者在后期表现出吲哚-3-丙酸血浆浓度降低。
总之,我们推测不同的饮食结构会改变肠道微生物的组成,从而导致吲哚类物质产生的个体差异。
• 细胞群体密度影响吲哚分泌
细胞外吲哚浓度取决于细胞群体密度,其中大肠杆菌和霍乱弧菌在指数早期开始产生吲哚。浓度在固定相中达到最高水平(在富集培养基中高达0.6mM吲哚),并在固定相期间稳定维持。
• 碳源影响吲哚产量
其次,有报道称葡萄糖抑制吲哚生物合成。TnaA的分解代谢抑制得到证实,因为tnaA基因的转录在碳限制期间受到抑制。
此外,tnaAB操纵子被大肠杆菌中的分解代谢调节蛋白环AMP复合物激活。因此,当大肠杆菌数量较多且碳源减少时,会产生相对大量的吲哚。
• 温度和pH影响吲哚生物合成
温度和pH也是影响大肠杆菌吲哚生物合成的重要环境因素。温度从30°C转变到43°C会诱导大肠杆菌中tnaAB基因的表达,但大肠杆菌在44.5°C时失去吲哚生物合成的能力。
注:与37°C相比,吲哚信号传导在较低温度(30°C)下对大肠杆菌基因表达、生物膜形成和抗生素耐药性的控制作用更为显著。
此外,低pH值会抑制大肠杆菌中吲哚的产生,而TnaA是pH9.0时最容易诱导的蛋白质之一。
• 生理状态影响吲哚生成
例如年龄、性别以及个体的生理状态都可能影响内源性吲哚的产生。睡眠模式、压力水平和运动习惯等生活方式因素都可能对内源性吲哚的产生产生影响。
此外,特定的健康状况和疾病,如肠道炎症、肝脏疾病等,可能会改变内源性吲哚的生成或代谢。
• 药物的使用也会影响吲哚生成
一些药物可能影响吲哚类物质的生成。例如,一些抗生素和非甾体抗炎药物被发现可以改变肠道微生物群的组成和功能,从而影响吲哚类物质的生成。
此外,一些药物可能直接干扰色氨酸代谢途径,从而影响吲哚类物质的生成。
小结
因此,细胞群密度、碳源、温度和pH等环境条件直接会影响吲哚的浓度;而饮食、药物的使用以及生活方式可能通过影响肠道微生物群或其他器官间接影响吲哚的生成。
肠道屏障的功能与肠道健康密切相关,在健康中发挥着至关重要的作用。肠道屏障将宿主与肠腔中的微生物隔离,并限制微生物和分子从肠腔的运动。
一旦肠道屏障受损,肠道通透性增加,细菌及其产物(如内毒素)将跨膜并激活单核巨噬细胞系统,促进大量炎症因子的产生,如白细胞介素6和肿瘤坏死因子-α(TNF-α),导致慢性微炎症状态。
吲哚对于控制肠道屏障功效至关重要,包括调节炎症和免疫反应、影响上皮通透性、粘液产生和紧密连接(TJ)形成。
▼
✦ 吲哚减轻非甾体抗炎药引起的肠道炎症
研究了使用吲哚作为非甾体抗炎药肠病的治疗方法。吲哚降低粪便钙卫蛋白浓度以及吲哚美辛诱导的脾和肠系膜淋巴结中中性粒细胞的浸润。
吲哚与吲哚美辛联合使用可减少非甾体抗炎诱导的粘膜转录组变化。粪便钙卫蛋白是肠道炎症的中性粒细胞特异性生物标志物。而中性粒细胞被认为是非甾体抗炎肠病发病的关键因素,可通过释放多种炎症介质引起炎症和组织损伤。
这就说明吲哚能够减轻吲哚美辛引起的小鼠肠道炎症,对非甾体抗炎药肠病具有潜在的免疫调节作用。
此外还发现吲哚减少促炎细胞因子IL-8的产生和趋化因子TNF-α激活的核因子κB(NF-κB)的表达,并诱导抗炎细胞因子IL-10的分泌。
研究表明,共生菌可以限制鼠伤寒沙门氏菌诱导的NF-κB激活,减轻小鼠炎症。因此,吲哚可能是益生菌减少肠道炎症的信号。
✦ 细菌产生的吲哚衍生物增强免疫屏障
吲哚乙酸
卵形拟杆菌(Bacteroides ovatus)产生的吲哚乙酸(IAA)增加了CD103+ /CD11c+免疫群体的丰度,而 CD103+ /CD11c+免疫细胞对于维持肠道免疫稳态和诱导耐受性免疫反应至关重要。
此外,吲哚乙酸与树突状细胞上的芳基氢受体结合并驱动IL-22的产生。在炎症性肠病个体中,IL-22 刺激上皮再生并减少炎症,并且在肠道炎症的调节中发挥关键作用。
注:除卵形拟杆菌外,其他拟杆菌产生的吲哚也可以激活免疫细胞上的AhR,从而产生IL-22。
在脂多糖存在的情况下,肠上皮细胞中表达的Reg4通过增加乳酸菌及其代谢物吲哚乙酸的比例来维持免疫稳态,从而促进肠组织中IL-35 +B调节细胞的产生和积累。这些结果表明吲哚乙酸可以调节IL-35 的产生,并影响肠道免疫屏障。
吲哚-3-乳酸
罗伊氏乳杆菌(Lactobacillus reuteri)产生吲哚-3-乳酸(ILA),ILA是Trp的吲哚衍生物,可激活CD4+ T细胞中的AhR并下调转录因子ThpoK,使CD4 + T细胞分化为具有免疫调节功能的CD4+ CD8αα +双阳性上皮内T淋巴细胞(DPIELs)。通过这些机制,吲哚-3-乳酸可以促进肠道屏障功能并减少炎症。
生活在母乳喂养婴儿胃肠道中的婴儿长双歧杆菌亚种也可产生吲哚-3-乳酸,从而显著减弱TNF-α和脂多糖诱导的肠上皮细胞中促炎细胞因子IL-8的增加。吲哚-3-乳酸还增加AhR靶基因CYP1A1和NRF2靶向基因谷胱甘肽还原酶2、超氧化物歧化酶2和NAD(P) H脱氢酶的 mRNA 表达,这可能是母乳喂养婴儿肠道炎症的重要调节剂。
此外,另一项研究表明,婴儿双歧杆菌分泌的吲哚-3-乳酸对未成熟肠道具有抗炎作用。这些数据为生产吲哚-3-乳酸益生菌的生产和饮食建议提供了重要的见解。
吲哚-3-丙酸
吲哚-3-丙酸(IPA)下调肠上皮细胞介导的炎症细胞因子TNF-α,同时上调配体蛋白编码mRNA,从而调节肠道屏障功能,缓解肠道炎症。
吲哚-3-丙酸还通过激活AhR显著诱导肠上皮细胞上的IL-10受体配体结合亚基(IL-10R1)来调节肠道免疫稳态。
IL-10R1是抗炎细胞因子IL-10的受体,IL-10通过IL-10R1发送抗炎信号,抑制多种细胞(包括肠上皮细胞)过度释放促炎介质。这进一步说明了吲哚-3-丙酸对肠道免疫屏障的保护作用。
吲哚-3-醛
乳杆菌(Lactobacillus)产生的吲哚-3-醛(IAld)有助于AhR依赖性IL22转录,IL-22产生者抑制炎症并保护粘膜表面的免疫生理学。吲哚-3-醛可以替代益生菌,在感染或化学损伤期间保护和维持粘膜完整性。
吲哚-3-醛可用作菌群处理和肠道菌群失调期间的支持疗法。乳酸菌还通过IAld诱导的AhR刺激LPL分泌IL-22,从而激活pSTAT3通路并抑制IL-1β和TNF-α分泌,以保护粘膜免疫屏障.
吲哚对肠道免疫的影响

Ye X,et al.Front Immunol.2022
服用非甾体抗炎药时,肠道微生物组以革兰氏阴性菌为主,这种转变会导致肠道损伤。吲哚和吲哚美辛的共同给药可以维持甚至增加厚壁菌门的重要成员,特别是对肠道稳态至关重要的C. cluster XIVa和C. cluster IV。似乎它可以防止拟杆菌的任何增加,这种变化可以减少肠粘膜损伤。
吲哚可通过增殖MUC基因的表达来增加HCT-8细胞对去甲肾上腺素介导的肠出血性大肠杆菌(EHEC)沉降的抵抗力。
吲哚-3-醛(IAld)激活AhR以产生IL-22,IL-22调节粘膜反应,使混合微生物群落存活并抑制白色念珠菌(Candida albicans)定植
✦ 减轻克雷伯菌属的细胞毒性
吲哚调节多种肠道细菌的毒力因子。吲哚增强替利霉素向替利伐林的转化。两者均由克雷伯菌属(Klebsiella spp.)产生,不同之处在于替利伐林是一种细胞毒性降低的吲哚类似物,并且替利伐林与上调孕酮X受体(PXR)反应性解毒基因结合并抑制微管蛋白定向毒性。因此,吲哚以多种方式减轻克雷伯菌属的细胞毒性。
✦ 调节霍乱弧菌和大肠杆菌的毒力基因表达
肠道益生菌代谢产生的吲哚在肠腔内浓度较高,减少了肠道大肠杆菌和啮齿类柠檬酸杆菌(Citrobacter rodentium)致病基因的表达。
吲哚还用作ToxR激动剂,调节肠道内霍乱弧菌(Vibrio cholerae)的毒力基因表达和生物膜产生。吲哚、吲哚甲酸和吲哚乙酸还可下调致病性大肠杆菌菌株的毒力和志贺氏毒素的产生。
此外,吲哚和7-羟基吲哚显著改变铜绿假单胞菌(Pseudomonas aeruginosa)的毒力基因表达,从而降低毒力因子并降低集群运动性。
铜绿假单胞菌用7-羟基吲哚治疗后,胃肠道中有更好的清除率。因此,吲哚可能有助于作为针对肠出血性大肠杆菌等病原体的治疗剂。
✦ 增强肠道机械屏障
吲哚通过诱导许多涉及内皮细胞的基因的表达,包括紧密连接、粘附分子连接(AJ)、肌动蛋白细胞骨架和黏蛋白形成,在体外增强内皮细胞的屏障功能。
将人肠上皮细胞系HCT-8暴露于吲哚以测量其基因表达的变化。结果显示,高剂量的吲哚增强了肠上皮细胞HCT-8中与黏蛋白产生、粘膜屏障增强和上皮间抵抗相关的分子谱。因此,吲哚加强了机械屏障。
✦ 增强肠道粘液屏障
吲哚-3-丙酸通过增加MUC2和MUC4以及杯状细胞分泌产物(TFF3和RELMβ)来增强粘液屏障功能。
吲哚-3-丙酸还促进IL-10释放,IL-10的信号转导增强粘液屏障功能并维持上皮细胞的稳态。
细菌中的吲哚-3-丙烯酸(IA)可增强IL-10的产生和MUC基因的表达,而IL-10对于维持杯状细胞中MUC的产生至关重要。因此,吲哚-3-丙烯酸通过 IL-10间接影响粘液屏障。
吲哚通过AhR或PXR调节肠道屏障
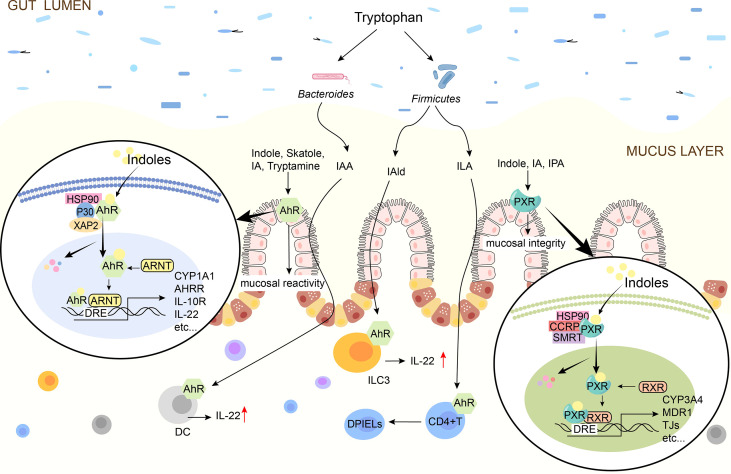
Ye X,et al.Front Immunol.2022
研究发现吲哚在细菌中发挥多种生物学作用,包括孢子形成、耐药性、毒力因子、质粒稳定性和生物膜形成等。
吲哚可以作为细菌间的信号分子
事实上,吲哚可以作为一种细菌信号,参与同一物种内以及不同物种之间的通讯。它在细菌生态系统中充当细胞间和细胞内信号。
注:吲哚还诱导了S. aurantiaca的孢子形成。
通过遗传筛选,在大肠杆菌中研究了吲哚作为细胞外信号分子的可能。结果,四个基因(astD、cysK、gabT和tnaB)被证明在稳定期通过自身产生的细胞外信号的积累而被激活。
在后续研究中,吲哚被证实是astD、gabT和tnaB激活所需的细胞外信号分子。
吲哚作为细胞间信号分子存在哪些争议?
吲哚是否是细胞间信号分子一直存在争议。已经提出了对群体感应信号分子要求的许多标准,如下所示:
1.假定的信号必须在特定阶段产生,吲哚主要在固定相中产生。
2.假定的信号必须在细胞外积累并被特定受体识别。吲哚的化学性质是众所周知的,并且在大多数吲哚调节情况下,化学互补被证明,其中吲哚在稳定期积累并且是已知的细胞外信号。该受体由AcrEF输出并由Mtr输入。
3.假定的信号必须累积并产生一致的响应,吲哚已被证明可以控制孢子和生物膜的形成。
4.重要的是,假定的信号必须引起超出信号代谢或解毒所需的生理变化的反应。吲哚已被证明可以控制毒力、生物膜和质粒稳定性,这些与吲哚无关代谢。
因此,根据这些标准,吲哚有可能成为群体感应分子。
由于上述四个标准仅适合规范的群体感应信号,例如AHL,Monds&O’Toole又添加了两个更普遍适用且具有实用价值的标准:
1.表型变化所需的生理相关信号浓度对细胞无毒。大肠杆菌和霍乱弧菌产生高达0.6mM的吲哚,对细胞无毒,并在该生理浓度下控制许多表型。
2.信号网络在社区层面是自适应的。尽管由于进化实验的困难而难以证明,但我们假设细胞间信号吲哚可能对微生物群落有益,尽管吲哚的生产对个体来说成本高昂。
例如,吲哚在产生吲哚的细菌大肠杆菌和霍乱弧菌中增加了质粒稳定性、耐药性。相反,在不产生吲哚的细菌中,吲哚通过干扰群体感应系统,降低了真菌的细胞生长和铜绿假单胞菌的毒力。因此,我们推测产生吲哚的细菌可能利用吲哚来对抗其他细菌和真核生物。
吲哚和tnaA基因也影响病原菌的毒力。色氨酸酶活性与致病性大肠杆菌有关,因为色氨酸酶活性是LEE1启动子完全激活所必需的。
吲哚会增加肠出血性大肠杆菌中毒力相关的EspA和EspB蛋白(LEE4基因产物)的分泌以及附着和消除病变的形成。
在霍乱弧菌中,吲哚和tnaA基因可能通过诱导毒力相关的分泌蛋白来增加对盘基网柄菌(Dictyostelium discoideum)的抗性。
此外,在流感嗜血杆菌分离株中,大多数血清型 (94-100%)呈吲哚阳性,而无害分离株中只有70-75%呈吲哚阳性。结果表明,吲哚的产生是必要的,但不足以对该菌株产生毒力。
值得注意的是,吲哚通过抑制毒素的产生来减轻克雷伯菌属(Klebsiella spp)的细胞毒性,还通过降低细胞运动性和聚集性来减少李斯特菌(L. monocytogenes)的相关毒力。
吲哚还通过改变基因表达来减少铜绿假单胞菌(不产生吲哚的细菌)中毒力因子的产生。
吲哚降低毒力基因的表达
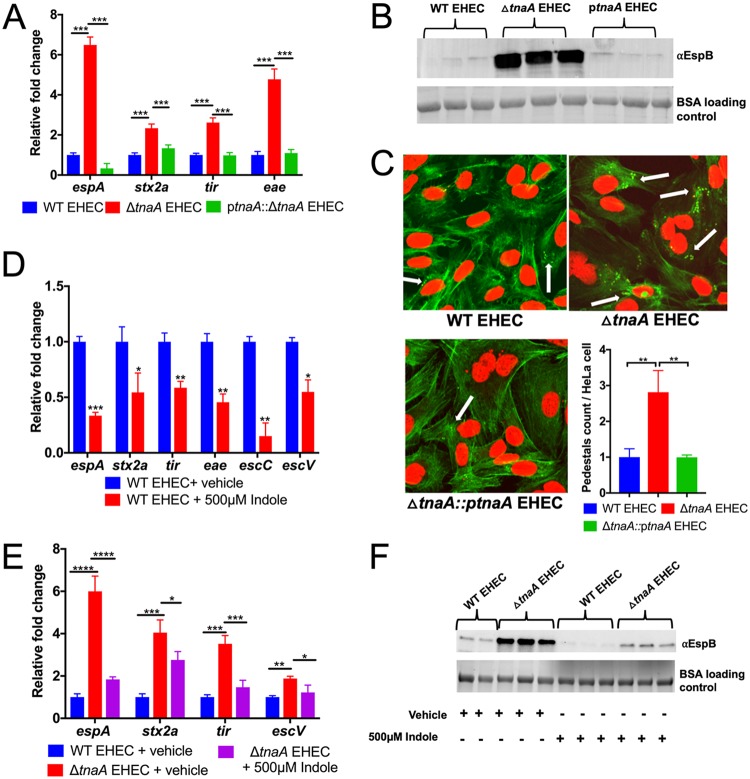
Kumar A,et al.mBio.2019
研究表明,吲哚通过在大肠杆菌中诱导输出基因(mdtEF和acrD)来增加耐药性,其中吲哚通过双组分信号转导系统(BaeSR和CpxAR)发挥作用。
这些双组分信号系统可以用作吲哚传感器。该结果证实了另一项研究,其中吲哚通过BaeSR和CpxAR诱导spy(球形体蛋白Y)基因的表达。
此外,有人认为GadX(AraC型转录因子)、Hfq(sRNA功能的全局调节因子)和RpoS(应激和固定相西格玛S)对吲哚诱导的mdtEF表达是必需的。因此,吲哚可能与多种调节因子相互作用。
增强大肠杆菌的质粒稳定性
吲哚增强大肠杆菌中的质粒稳定性。研究表明,来自大肠杆菌质粒ColE1的小非编码RNA与TnaA结合,有助于防止质粒丢失,而吲哚则延迟细胞分裂。
大肠杆菌中的吲哚还通过抑制gadABCEX、hdeABD和ymgB等耐酸基因来降低耐酸性。大肠杆菌可能会在弱碱性肠道菌群中存在吲哚的情况下关闭耐酸性基因,因为在通过酸性胃存活后不再需要耐酸性蛋白。
降低了运动性
此外,吲哚是一种化学排斥剂,会降低运动性,可能是由于细胞分裂干扰,而肾上腺素和去甲肾上腺素是一种化学引诱剂,会增加大肠杆菌O157: H7的运动性。
此外,吲哚降低细胞对上皮细胞的粘附,而肾上腺素和去甲肾上腺素增加细胞粘附。由于细菌粘附和定植到上皮细胞对于感染很重要,因此假设这也会对细菌毒力产生不同的影响。
吲哚对其他微生物的影响如下所示:

Lee JH, Lee J.FEMS Microbiol Rev.2010
吲哚类物质被肠上皮细胞吸收并扩散到血液中,从而循环到全身并影响各个系统,例如肠道及相关疾病的调节。
谷禾在这里总结了它们对炎症性肠病、出血性结肠炎、结直肠癌、糖尿病、中枢神经系统炎症和血管调节的影响。
Ye X,et al.Front Immunol.2022
炎症性肠病是克罗恩病、溃疡性结肠炎等一组疾病的总称,是临床上重要的慢性胃肠道炎症性疾病。大量研究表明,吲哚对缓解炎症性肠病症状有一定的作用。
✦ 吲哚类物质增加了抗炎细胞因子
炎症性肠病与粘液层有关,炎症性肠病患者的内部粘液层较薄,MUC2糖基化较低。结果表明,吲哚-3-丙烯酸甲酯(IA)处理小鼠结肠球体可显著增加AhR激活和MUC2基因表达。
即使在脂多糖刺激的共培养物存在下,IA仍能保持其对MUC2基因表达的影响并增加IL-10的产生。杯状细胞产生MUC依赖于IL-10。因此,细菌产生的 IA 有可能增加IL-10的产生和MUC基因的表达,这可能对炎症性肠病患者有利。
吲哚对于肠出血性大肠杆菌的发病机制至关重要。出血性结肠炎是一种出血性腹泻,可导致溶血性尿毒症综合征,由人类病原体大肠杆菌O157:H7引起。
✦ 吲哚减少了肠出血性大肠杆菌的定值
根据表面相关EHEC的DNA微阵列研究,吲哚降低了EHEC中与表面定植和致病性有关的基因表达。
吲哚可以被加氧酶氧化,产生新的种间生物膜信号,从而以多种方式影响相同的表型(生物膜产生)。大肠杆菌K-12和肠道内其他共生菌产生的吲哚可能会限制大肠杆菌K-12生物膜的产生,并通过改变SdiA的表达来降低非致病性大肠杆菌的运动性,从而影响EHEC趋化性和粘附力。粘附测定证实,吲哚可在体外减少EHEC与上皮细胞的附着。
总之,吲哚通过改变肠出血性大肠杆菌毒性基因的表达、减少其运动性和生物膜形成以及减少粘附来减轻肠出血性大肠杆菌感染并改善出血性结肠炎。
根据越来越多的数据,肠道吲哚,特别是色氨酸代谢物,似乎在结直肠癌中发挥着重要作用。吲哚刺激AhR,它可以直接作用于肠道干细胞,维持MUC合成并改善肠道屏障功能。
然而,在炎症相关结直肠癌发生模型中,阻断吲哚-AhR信号通路会显著增加TNF-α、IL-1β和IL-6 mRNA水平。
此外,吲哚衍生物色胺会抑制吲哚胺2,3-双加氧酶(IDO1),该酶参与肿瘤免疫耐受。这些发现表明肠道菌群产生的吲哚可能会减缓结直肠癌的进展。
与健康个体相比,结直肠癌患者的吲哚/色氨酸比率较低,犬尿氨酸(kyn)/色氨酸比率较高。在结直肠癌患者中,犬尿氨酸和IDO1的表达增加,吲哚的产生减少。
吲哚产生的下降削弱了对结肠癌的抑制作用。总体而言,有证据表明微生物色氨酸-吲哚代谢途径的改变在结直肠癌的发病机制中发挥着重要作用。
较高的吲哚-3-丙酸血清浓度可降低患2型糖尿病的可能性,并增强胰岛素分泌和敏感性。
研究发现,与喂食对照饮食的大鼠相比,喂食吲哚-3-丙酸饮食的大鼠的空腹血糖水平显著降低。根据其他研究,吲哚可以调节小鼠结肠成纤维细胞释放胰高血糖素样肽-1(GLP-1)。
✦ 吲哚调节胰高血糖素样肽-1的释放
吲哚在短时间内增加GLP-1的释放,在较长时间内减少其分泌。这些作用结果的产生是因为吲哚可以影响L细胞的两个关键化学途径。
在某种程度上,吲哚可以抑制电压门控K+通道,增加L细胞诱导的动作电位持续时间,并导致Ca2+进入大量增加,从而快速促进GLP-1的分泌。吲哚抑制了NADH脱氢酶,随着时间的推移减缓ATP的生成并降低GLP-1的释放。
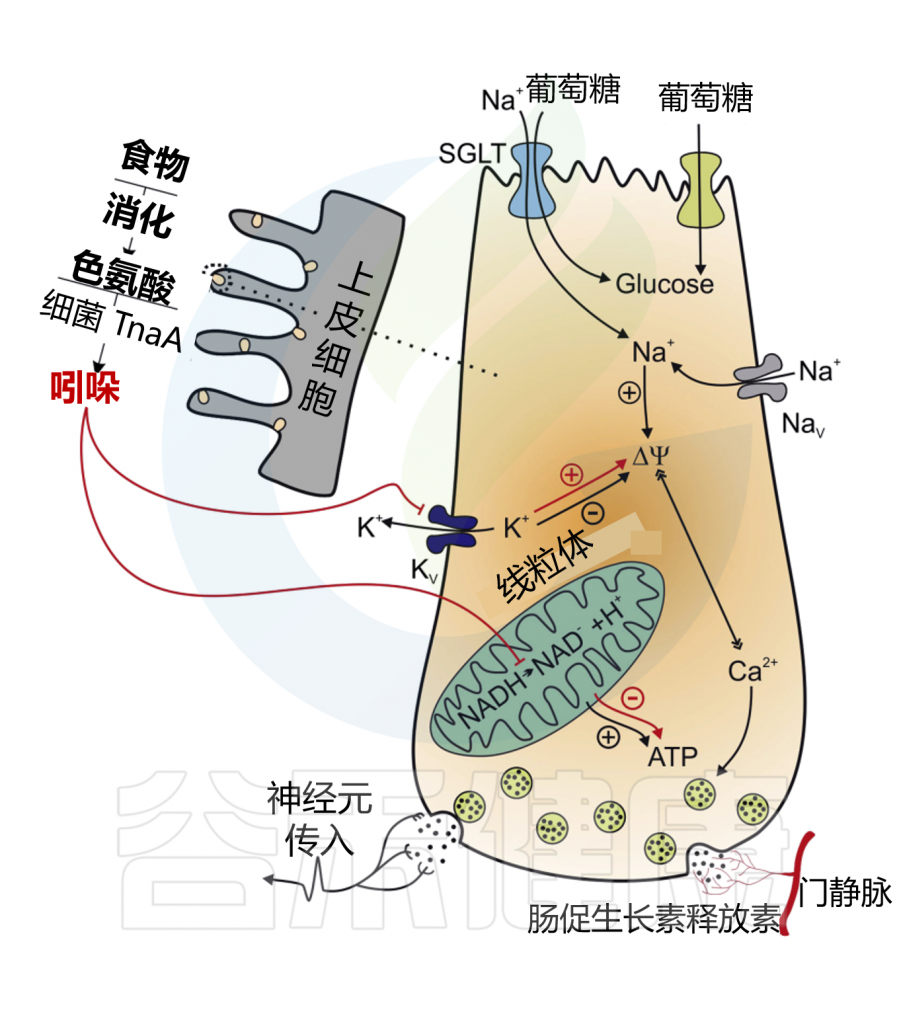
Chimerel C,et al.Cell Rep.2014
肠道内的吲哚可能还会影响食欲,同时,吲哚-3-丙酸具有强大的抵抗氧化应激的能力,这表明这种代谢物可以保护β细胞免受代谢和氧化应激相关的损伤以及淀粉样蛋白积累。因此,肠道微生物群代谢产生的吲哚-3-丙酸对2型糖尿病具有保护作用。
体外实验发现,吲哚丙酸也是嗜肺军团菌(Legionella pneumophila)生长的强效抑制剂,且抑菌效果随着吲哚丙酸浓度和暴露时间的增加而增强。因此,吲哚丙酸可以抑制细菌引起的肺部炎症。
HIV阳性患者通常更容易感染结核病,这是死亡率增加的重要因素,两种疾病的共同感染可能会因抗逆转录病毒药物和抗结核药物之间的相互作用而使治疗复杂化。
值得注意的是,吲哚丙酸可能是此类治疗中潜在的药物成分。有研究发现,接受抗逆转录病毒治疗的HIV患者体内吲哚丙酸含量显著下降,这是区分HIV感染者与健康人的重要标志物。
这种作用的机制尚不清楚,但部分原因是肠道屏障受损导致脂多糖和脂多糖结合蛋白易位,从而加剧全身炎症反应。此外,接受抗逆转录病毒治疗的HIV患者也容易出现非酒精性脂肪性肝病/非酒精性脂肪性肝炎,进一步说明了吲哚丙酸在各种疾病中的多种治疗潜力。
相比之下,吲哚丙酸是一种小分子代谢产物,具有良好的药代动力学特性,很容易被宿主吸收,充分发挥治疗作用。
未来,吲哚丙酸有望通过其作为抗结核病和抗逆转录病毒药物的补充的潜力来改善某些疾病的现有治疗选择。
吲哚丙酸可以触发孕烷X受体(PXR),一种生物量激活的核受体,存在于包括血管内皮在内的各种组织中,以调节内皮功能。
吲哚丙酸通过PXR调节主动脉和肺动脉导管中激动剂诱导的内皮依赖性松弛。这种调节主要是由于内皮一氧化氮合酶产生的一氧化氮的改变,而内皮一氧化氮合酶受到吲哚丙酸介导的PXR激活的抑制。
然而,破坏肠道微生物群并降低吲哚丙酸丰度的抗生素治疗会通过改变内皮PXR途径来改变吲哚丙酸的血管舒张作用。
而微生物代谢的吲哚丙酸补充可以提高全身吲哚丙酸水平并刺激PXR表达,从而逆转抗生素治疗引起的主动脉和肺动脉中激动剂诱导的内皮依赖性血管舒张的增强。
注:吲哚与血管的相互作用可能会显著改变传统传染病或结肠手术相关的抗生素治疗,导致微生物群落紊乱。
尽管吲哚对于改善肠道甚至全身疾病至关重要,但它们中的一些仍然会产生负面影响,例如硫酸吲哚酚(IS)、吲哚基-β-d-葡萄糖苷和吲哚乙酸。
硫酸吲哚酚是最重要的肾毒性代谢产物之一,其肾毒性已在临床研究中得到广泛证实。此外,一些吲哚类物质还会对胃肠系统、神经系统和心血管系统产生毒性。
越来越多的证据表明吲哚和吲哚化合物对大脑代谢、生理和宿主行为有影响。在拥有产生吲哚的大肠杆菌的大鼠中,吲哚的慢性过量产生已被证明会增强焦虑样行为和抑郁。
另一项研究还发现,给予常规大鼠肠道内吲哚可以激活一个称为迷走神经背侧复合体的大脑核。通过比较与非吲哚产生大肠杆菌菌株或吲哚产生大肠菌菌株单体相关的小鼠,发现肠道微生物群的长期高吲哚产生增加了慢性压力对整体情绪行为不利影响的敏感性。
✦ 可能影响儿童自闭症的发病率
在人类中,一项研究发现,患有自闭症谱系障碍的儿童在大便样本中吲哚水平较健康儿童低,而3-甲基吲哚水平升高。 这两种吲哚与患者中更常见的梭菌属细菌有关。
肠道微生物代谢活性产生的血浆吲哚升高与肝性脑病有关,这是由肝功能障碍引起的神经精神障碍,以及个体意识、行为和个性的变化。
此外,NutriNet-Santé观察性前瞻性研究的结果表明,尿液中吲哚和吲哚化合物浓度与反复发作的抑郁症状呈正相关。这种相关性提出了这样一个假设:肠道微生物过度产生这些化合物可能在人类情绪障碍的出现中起到作用。
硫酸吲哚酚由肠道菌群和宿主共同代谢。肠道菌群用酶分解食物中的色氨酸生成吲哚,然后由门静脉携带至肝脏,在细胞色素P450酶和磺基转移酶的作用下转化为硫酸吲哚酚。
硫酸吲哚酚通过上调IRF1表达、抑制动力相关蛋白1表达并干扰线粒体自噬通量来诱导肠上皮细胞(IEC)损伤。硫酸吲哚酚通过以浓度依赖的方式增加活性氧的释放,在IEC-6细胞中引起氧化应激。
硫酸吲哚酚还抑制NRF2激活,降低抗氧化防御细胞系统并抑制血红素加氧酶-1、NAD(P)H 脱氢酶和超氧化物歧化酶的表达。
在IEC-6细胞中,硫酸吲哚酚处理可显著增加TNF-α的释放、环氧合酶-2和诱导型一氧化氮合酶的产生以及硝基酪氨酸的合成,表明肠上皮细胞是硫酸吲哚酚诱导的肠道炎症的靶点。
与硫酸吲哚酚一起培养的结肠显示出收缩性降低,这表明该毒素可能对结肠平滑肌细胞产生有害影响并导致肠道蠕动受损。
微生物群代谢物硫酸吲哚酚在中枢神经系统疾病的发生中也具有致病作用。众所周知,慢性肾病和心血管疾病常常是由硫酸吲哚酚引起的。此外,肾功能不全会导致尿毒症毒素在大脑中积聚,导致中枢神经系统功能异常。
✦ 损伤神经元和神经干细胞
其次,硫酸吲哚酚可损伤神经元和神经干细胞,损害神经营养因子和神经递质,诱发氧化应激和神经炎症。例如,通过作用于中枢神经系统胶质细胞,硫酸吲哚酚促进神经炎症并表现出促炎作用。
小鼠表现出情绪障碍和神经元退化的行为证据,如焦虑、抑郁和认知功能障碍。这些相应的器质性病变伴随着这些行为变化。这也表明硫酸吲哚酚和中枢神经系统疾病之间似乎存在病理联系。
吲哚对神经和神经精神疾病的影响
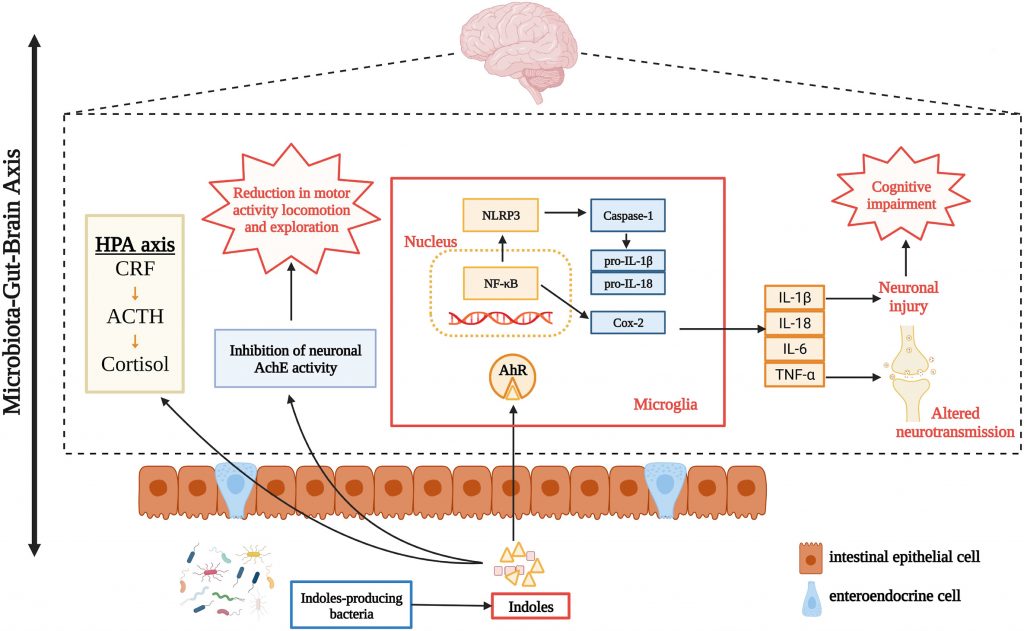
编辑
Zhou Y,et al.Neuropharmacology.2023
硫酸吲哚酚对大脑产生有益或有害的影响取决于含量:
通过比较患有抑郁症的志愿者和健康参与者,鉴定出了22种尿液代谢物,并且两组受试者之间的丰度存在差异。患有严重抑郁症的个体尿液中硫酸吲哚酚浓度低于健康人。在志愿者中,血清硫酸吲哚酚浓度与精神焦虑和相关的基于功能磁共振成像的神经特征有关。
在实验性自身免疫性脑脊髓炎的小鼠模型中,每日腹腔注射硫酸吲哚酚(10mg/kg)可调节星形胶质细胞活性,并通过芳基烃受体对中枢神经系统发挥抗炎作用。相反,当大鼠口服较高剂量(100和200mg/kg)的硫酸吲哚酚时,观察到空间记忆受损以及运动和探索活动减少。
另一项研究发现,单次腹腔注射更大剂量(800mg/kg)的硫酸吲哚酚会引起与神经元坏死相一致的大脑组织学变化。硫酸吲哚酚在15-60微摩尔范围内使用时,会诱导原代星形胶质细胞产生自由基氧,并导致海马神经元细胞死亡。在10微摩尔浓度下,硫酸吲哚酚通过氧化应激诱导人星形胶质细胞凋亡。
在慢性肾病(CKD)患者中,硫酸吲哚酚与心血管健康和全因死亡率相关。硫酸吲哚酚是血液透析患者发生心血管疾病的重要因素。在血液透析中,发现游离形式硫酸吲哚酚水平与成纤维细胞生长因子23呈正相关,与C-C基序趋化因子15、补体成分C1q受体、串珠素(Perlecan)、博来霉素水解酶、分化簇166抗原和信号淋巴细胞活化分子家族成员5呈负相关。
这些蛋白质在血管修复和内皮生长中发挥着至关重要的作用。硫酸吲哚酚还可以通过增加血小板过度活跃、提高血浆促凝血水平和产生促凝血颗粒来导致血栓形成和动脉粥样硬化。硫酸吲哚酚通过增加炎症、心脏纤维化、心肌细胞增殖来影响心脏组织。
并且强有力的证据表明,硫酸吲哚酚在肾功能不全的情况下积累时是有害的。硫酸吲哚酚会损害近端肾小管细胞并诱导炎症和纤维化发展。肾脏通过肾小管分泌实现硫酸吲哚酚的高清除率,而硫酸吲哚酚与血浆蛋白结合超过90%,受到蛋白结合的限制,并且血液透析患者的血浆水平相对较高,这也表明与肾脏疾病密切相关。
啮齿动物口服新霉素(一种广谱抗生素)会降低脑内羟吲哚含量,因此表明羟吲哚至少部分源自肠道微生物群代谢活动。
有趣的是,最近在人类粪便样本中发现了羟吲哚,从而证实肠道微生物是这种代谢物的来源。在人类粪便中存在的细菌代谢物中,羟吲哚被发现是主要的芳基烃受体激活剂之一。
大鼠海马切片的体外实验表明,羟吲哚可能与电压门控钠通道相互作用,增加产生动作电位的阈值,从而大大降低神经元的兴奋性。
大胆推测
这些结果说明羟吲哚可能与已知的神经抑制化合物具有一些共同特征,从而能够降低神经元的兴奋性
吲哚及其衍生物可以影响肠道健康甚至是全身其他器官的健康,而过高或过低的水平都可能带来不利影响,导致一些疾病的发生。
调节人体内吲哚水平的机制主要有3种:首先,它可以通过调节与吲哚产生相关的肠道微生物群的丰度来影响吲哚的水平。
先前的研究发现,特定的肠道微生物群成员在色氨酸转化为吲哚的过程中发挥着不可或缺的作用,例如产孢梭菌(Clostridium sporogenes),它已被证明可以通过氧化和还原途径产生吲哚乙酸和吲哚-3-丙酸。在大肠杆菌中也发现了吲哚活性转运蛋白,在乳杆菌中也发现了色氨酸酶的表达,并有助于将色氨酸转化为吲哚。
其次,饮食调整可以增加或减少色氨酸的摄入量,从而间接影响体内的吲哚水平。
最后,直接作用于AHR受体激动剂的药物可能会发挥与吲哚类似的作用。
益生菌可以通过多种机制与体内微生物群相互作用,对宿主健康产生有益影响。
临床试验表明,益生菌可以抵消肠道微生物失衡造成的负面影响,并可导致吲哚产生属及其衍生物的富集,如乳杆菌(Lactobacillus)。在另一项临床试验中,具有抗炎作用的双歧杆菌属在益生菌治疗后的丰度也显著增加。
益生菌还有可能成为神经和精神疾病的治疗辅助药物。在一项针对抑郁症和海马体功能变化的研究中,接受益生菌治疗的小鼠表现出肠道微生物生态失调的显著缓解,以及较低的肠道炎症介质如IL-1β、TNF-α和iNOS的表达水平。此外,情感行为也被检测到与肠道微生物群组成变化相关的显著改善。
益生元是宿主细菌可以选择性利用并提供健康益处的底物。常见的益生元包括低分子量碳水化合物、多酚和多不饱和脂肪酸。
在实验室和临床试验中,益生元(包括菊粉和低聚半乳糖)已被证明可以显著提高产生吲哚的益生菌(例如双歧杆菌和乳酸杆菌)的丰度。在对肠道微生物失衡进行一定纠正后,吲哚作为具有调节作用的配体,对炎症反应具有抑制作用。
合生元是益生菌和益生元的混合物。在大鼠实验中,合生元治疗逆转了肠道微生物特定菌群丰度的变化。
经过合生元(乳杆菌、双歧杆菌、链球菌和菊粉的组合)治疗的患有慢性肾病的大鼠中,肠道中产生吲哚的产气荚膜梭菌的丰度降低至正常水平。此外,合生元治疗可通过改变肠道微生物丰度来有效调节粪便中吲哚及其衍生物的水平。
合生元治疗由于其对吲哚水平的间接影响,可能成为许多神经系统和神经精神疾病的辅助治疗。最近一项针对阿尔茨海默病患者的临床实验表明,在使用合生元后,神经炎症有所减轻。
值得注意的是,合生元干预的改善效果优于单独的益生元或益生菌干预,这表明复合干预的组合更有可能成为临床治疗的实际应用。
粪便微生物移植(FMT)在先前的几项研究中已被证明对缓解胃肠道微生物失调具有不可否认的作用。通过FMT将产生吲哚的益生菌(乳酸杆菌、双歧杆菌等)移植到患者的肠道中,可以改善肠道微生物群。据观察,肠道中产生吲哚的微生物群的丰度增加,吲哚及其衍生物的水平受到间接影响。
吲哚是色氨酸代谢途径上的关键配体,可以调节和抑制神经和精神疾病的炎症。粪菌移植已被证明在各种疾病和临床治疗中对改善患者的肠道生态起着至关重要的作用,它可以通过吲哚在中枢神经系统疾病治疗中间接发挥作用,并被认为是一种潜在的辅助疗法。
肠道微生物通过色氨酸调节吲哚水平。色氨酸是人体必需氨基酸之一,需要完全依赖饮食摄入。
以下是一些富含色氨酸的常见食物:
蛋白质食物:肉类(牛肉、猪肉、鸡肉、火鸡)、鱼类(鳕鱼、鲑鱼、金枪鱼)、贝类(蛤蜊、扇贝)、蛋(鸡蛋、鸭蛋)、豆类(大豆、黑豆、红豆)、奶制品(牛奶、乳制品)等。
蔬菜和水果:菠菜、花椰菜、蘑菇、芝麻、松子、香蕉、菠萝、樱桃、草莓、蓝莓等。
谷物和豆类:燕麦、大麦、小麦、黑米、糙米等。
坚果和种子:核桃、杏仁、腰果、葵花子、南瓜子、亚麻籽等。
研究表明,0.4%膳食色氨酸可显著减少衰老小鼠的神经退行性病变和神经炎症。在此过程中,补充色氨酸可以通过改变肠道微生物组成并增加阿克曼氏菌等有益细菌的丰度来增强肠粘膜屏障功能。肠道菌群产生的吲哚类物质(如吲哚乙酸、吲哚-3-丙酸)显著升高,可以激活AHR,从而抑制神经炎症。
通过血清靶向代谢组学分析和16s rRNA测序对全身色氨酸代谢和肠道微生物组进行了分析,进一步证实了微生物群-肠-脑轴在调节色氨酸饮食产生吲哚的潜在作用。
虽然色氨酸饮食是一种可能的治疗策略,但过多的色氨酸摄入也被证明具有有害作用,应注意适量摄入。
此外,据报道,间歇性禁食还可能通过调节肠道微生物组成来影响血浆吲哚,从而对神经和神经精神疾病的预后产生有益影响。饮食调节可能是改善吲哚含量的一种简单但有效的方法。
最近的综合研究表明,吲哚通过作用于靶细胞来调节炎症反应和细胞生理。近年来,开发吲哚类作用靶点的药物一直是一个重要的研究热点。
吲哚及其衍生物充当AHR的配体,并通过AHR间接调节星形胶质细胞和小胶质细胞的炎症。因此,直接作用于AHR的激动剂也可以作为内源性AHR配体(如吲哚)的替代品,并参与色氨酸AHR介导的炎症抑制途径。
体外合成的AHR配体2-(1′H-吲哚-3′-羰基)-噻唑-4-羧酸甲酯在一项临床研究中被证明具有抑制自身免疫性脑脊髓炎进展的能力。对AHR具有激动作用的药物有可能取代色氨酸代谢产物,并作为外源性AHR配体激活自限制途径和修复神经和神经精神疾病。
除了靶向AHR,影响吲哚作用途径中其他靶点的药物也可能具有治疗作用,这需要在未来进行更多的研究。
由于吲哚是肠道细菌常见的代谢产物,其重要的生物学作用不容忽视。吲哚与肠道菌群的稳态和肠道健康直接相关,吲哚还可以间接影响其他系统和人体的整体健康。
吲哚对人类健康具有有益影响,通过激活免疫细胞释放抗炎因子如IL-22来调节肠道屏障并帮助维持肠道稳态,抑制包括肠出血性大肠杆菌在内的病原菌的定植,以及增加MUC表达以增强粘液屏障功能。
此外,吲哚在调节肠道微生态方面也起着关键作用。它们抑制有害菌株,并以影响基因表达的方式改变肠道致病菌的毒力,这有助于缓解出血性结肠炎等疾病。同时,吲哚及其衍生物在激活AhR和PXR介导的抗炎途径中至关重要。例如吲哚-3-丙酸通过PXR介导肠细胞TNF-α的下调,并上调编码生长抑素的mRNA,以调节炎症中的肠道通透性和肠道屏障功能。这意味着吲哚及其衍生物有前景的治疗途径。
尽管如此,吲哚也是一把双刃剑,少数衍生物也存在一定的缺点。例如,吲哚肝脏代谢产生的硫酸吲哚酚在高浓度时具有肾毒性和心血管毒性,可通过促进氧化应激、炎症等病理变化而导致多系统功能障碍。
吲哚在肠道稳态和人类健康中发挥着至关重要的作用。许多肠道细菌被鉴定为将色氨酸代谢为吲哚。尽管如此,仍可能存在未鉴定的细菌,因此未来需要使用代谢组学和宏基因组学来进一步表征未知的吲哚产生细菌及其相关代谢途径。
不同浓度的吲哚发挥着不同的生理功能。健康成年人的粪便吲哚浓度是已知的,而人类肠道和血液中许多吲哚衍生物的浓度仍然未知。缺乏在不同环境中测定吲哚及其衍生物浓度的定量方法,未来需要开发简单快速的测定方法。
未来仍需进一步研究吲哚与人类健康之间的关系。需要研究多种吲哚组合对宿主生理学的影响,并将这些发现与临床环境相结合,以开发相关疾病的新疗法。
主要参考文献
Zhou Y, Chen Y, He H, Peng M, Zeng M, Sun H. The role of the indoles in microbiota-gut-brain axis and potential therapeutic targets: A focus on human neurological and neuropsychiatric diseases. Neuropharmacology. 2023 Nov 15;239:109690.
Tennoune N, Andriamihaja M, Blachier F. Production of Indole and Indole-Related Compounds by the Intestinal Microbiota and Consequences for the Host: The Good, the Bad, and the Ugly. Microorganisms. 2022 Apr 28;10(5):930.
Ye X, Li H, Anjum K, Zhong X, Miao S, Zheng G, Liu W, Li L. Dual Role of Indoles Derived From Intestinal Microbiota on Human Health. Front Immunol. 2022 Jun 17;13:903526.
Lee JH, Lee J. Indole as an intercellular signal in microbial communities. FEMS Microbiol Rev. 2010 Jul;34(4):426-44.
Jiang H, Chen C, Gao J. Extensive Summary of the Important Roles of Indole Propionic Acid, a Gut Microbial Metabolite in Host Health and Disease. Nutrients. 2022 Dec 28;15(1):151.
Kumar A, Sperandio V. Indole Signaling at the Host-Microbiota-Pathogen Interface. mBio. 2019 Jun 4;10(3):e01031-19.
Chimerel C, Emery E, Summers DK, Keyser U, Gribble FM, Reimann F. Bacterial metabolite indole modulates incretin secretion from intestinal enteroendocrine L cells. Cell Rep. 2014 Nov 20;9(4):1202-8.
Lu Y, Yang W, Qi Z, Gao R, Tong J, Gao T, Zhang Y, Sun A, Zhang S, Ge J. Gut microbe-derived metabolite indole-3-carboxaldehyde alleviates atherosclerosis. Signal Transduct Target Ther. 2023 Oct 4;8(1):378.