-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康
便秘的全球患病率为14%。慢性便秘会影响生活质量。健康人相比,便秘还会导致粪便重量降低,且与结直肠癌风险增加相关。
饮食调整是治疗便秘的主要方法之一。世界胃肠病学协会建议通过饮食建议或补充来增加纤维摄入量。在英国,健康专家指南建议食用水果,包括富含山梨醇的水果,例如杏子、桃子和李子,以及相应的果汁。然而,关于水果对肠道运动和便秘的作用机制的证据有限。
本文旨在探讨水果和水果制品对肠道微生物群、肠道运动和便秘的作用机制和有效性。
关于水果中的成分,离不开以下几大成分。
▌ 膳食纤维
水果是膳食纤维的极好来源。
纤维是什么?
纤维是指三个或三个以上单体单元的聚合物碳水化合物加上木质素在小肠中不被消化或吸收的总和。纤维不是一个分子,而是一系列在溶解度、粘度和发酵性方面不同的分子。
纤维会增加粪便体积和生物量
不可发酵纤维完好无损地到达下消化道,而粘性纤维具有较高的水结合能力,因此,这两种纤维都能有效地增加粪便体积。
粪便体积的增加进一步导致管腔扩张并触发蠕动。可发酵纤维增加肠道微生物群的丰度,从而增加粪便生物量,并增加短链脂肪酸的产生。
纤维使粪便变软
这导致结肠渗透负荷增加,从而增加粪便的含水量,从而导致粪便变软。研究发现,各种高纤维食物可以缩短整个肠道的转运时间。
▌ 山梨糖醇
山梨醇是一种在植物中发现的糖醇。山梨醇在小肠内不被消化或吸收,并且能够在其分子中保持水分,从而导致肠腔中水分的增加,从而软化粪便,从而缓解排便。
在一项随机对照试验(RCT)中,与安慰剂相比,每天40克山梨醇持续6天会导致粪便水和粪便重量显著增加。此外,未被吸收的山梨醇到达结肠,在结肠中被肠道微生物群发酵,增加短链脂肪酸的产生,并可能改变微生物群。
虽然这一假设尚未在人类身上得到验证,但与低聚果糖和对照组相比,大鼠体内的山梨醇增加了粪便、结肠和盲肠乳酸杆菌AD102和粪便罗氏乳酸杆菌,并且结肠和盲肠样本中的丁酸含量高于对照组。
▌ (多)酚类
多酚是植物性食品和饮料中的一大类化合物,包括水果、蔬菜、谷物、茶、咖啡和葡萄酒。它们的结构由一个或多个与芳香烃基团结合的羟基组成。
只有一小部分低分子量(聚)酚在小肠中被吸收,而那些高分子量的酚到达结肠时不受影响,在结肠中它们可被肠道微生物群发酵,从而将较大的(聚)酚分解成较小的可吸收分子,可能对多种健康有益。
此外,现有证据表明,(多)酚有可能通过增加有益细菌(如双歧杆菌和乳酸杆菌)来积极改变肠道微生物群,或者通过抑制潜在致病菌的生长。虽然有人假设,由于其抗炎能力(多聚)酚类可能有助于治疗炎症性肠病或肠易激综合征,但目前没有足够的数据显示其对便秘的直接影响。
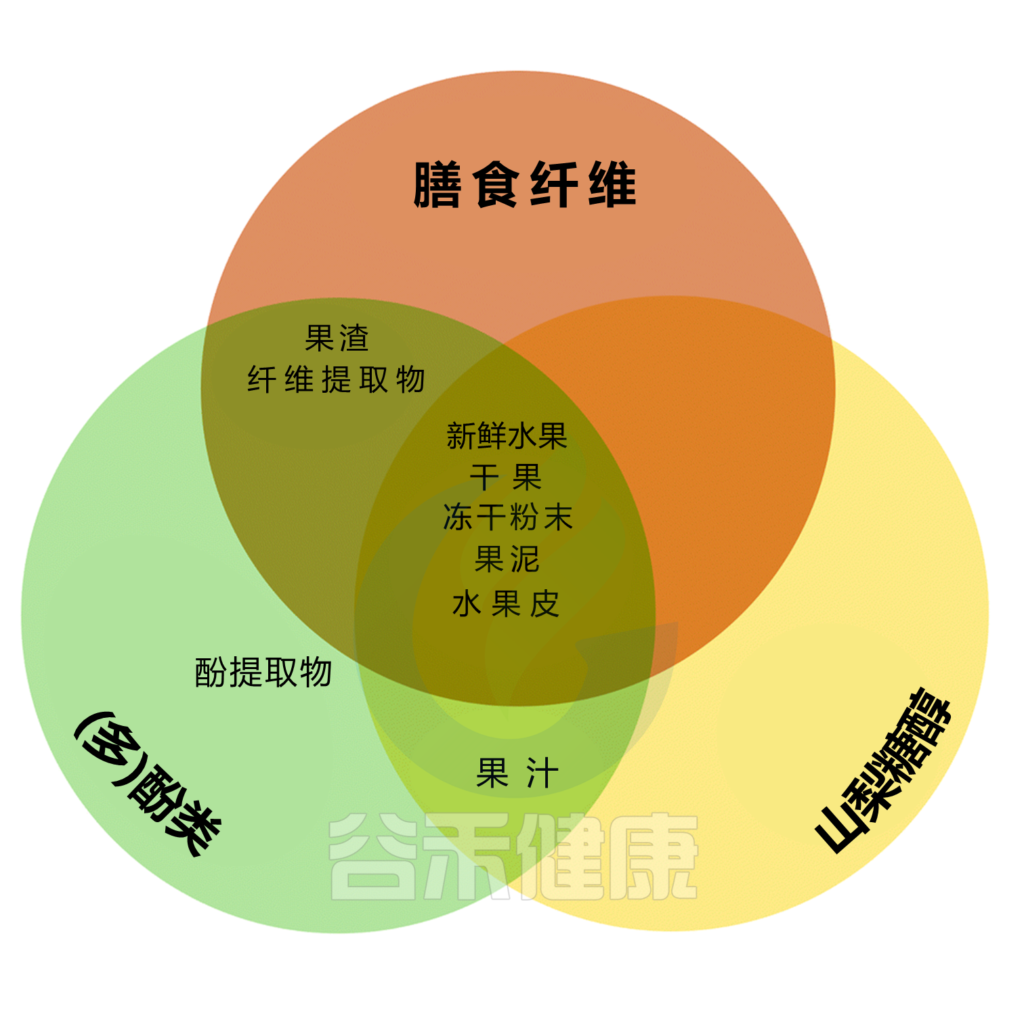
纤维、山梨醇和(多)酚是介导水果对肠道微生物群、肠道运动和肠道功能影响的主要成分,因此可能影响便秘。
不同的水果含有不同数量的纤维、山梨醇和(多)酚类物质,而水果的加工过程可能对这些物质产生重大影响。例如,新鲜或干果、果肉或全果冰沙(用新鲜、纯水果制成的饮料)可能含有大量的纤维、山梨醇和(多)酚,而浓缩果汁或浓缩果汁可能含有山梨醇和(多)酚,但纤维很少。
谷禾健康
关于微生物的研究已成为研究热点之一,在生物医学、环境、动植物等各个领域都有它的应用。
与谷禾合作的客户研究成果都已逐步显现,目前已有上百篇文章发表在各个期刊,我们选了其中几篇文章分享给大家,文末有前沿科学家的写作经验供大家参考。
肝纤维化
研究肠道微生物群与肝纤维化的关系,建立检测和分期肝纤维化的微生物群生物标志物。
将131只大鼠分为肝纤维化组(n=66)和对照组(n=65),其中肝纤维化(四氯化碳诱导4,6,8,12周)分为4个阶段,F1(n=15),F2(n=22),F3(n=11),F4(n=18),对其粪便样本进行16srRNA测序(V4区),在正常肝大鼠和肝纤维化大鼠之间发现了肠道微生物群的显著变化。
该研究证实了肠道微生物群是检测和分期肝纤维化的潜在生物标志物,具有较高的诊断准确率。
谷禾健康
生物累积(Bioaccumulation)是什么?
分解这个术语“生物累积”时,你会得到什么?accumulation ‘’积累’ 这个词意味着某种东西的积累,而前缀 bio- 意味着生命。把它放在一起,就是生物体内积累一些东西来定义生物累积。
那么生物物累积用于指生物体内化学物质的积累。这些物质没办法被代谢 ,便累积于生物体内,经由食物链中各阶层消费者的食性关系而累积,越高级消费者的体内其累积浓度越高的现象。
生物累积是如何发生的?
一种方法是让一定数量的化学物质进入生物体的速度快于生物体分解和使用它的速度。这意味着进入的比排出的多,导致化学物质在生物体内积累。
另一种主要方式是化学物质进入生物体,而生物体无法以某种方式将其分解或排泄。于是化学物质会继续积累,直到最终对生物体变得致命。
所有生物体面临的问题都是吸收和消除之间动态平衡的结果。生物蓄积发生的程度决定了所表现出的毒性作用。在某些情况下,保护机制免受沉积在特殊部位的某些不良物质的影响,以防止它们参与代谢反应。
本文的生物累积描述是用于评估药物及其有害代谢物的在肠道细菌内的积累。
治疗药物会对肠道微生物群产生强烈影响,反之亦然。潜在的药物-细菌相互作用可以通过生物转化降低微生物适应性或改变药物可用性。后者可以对药物活性和功效产生正面或负面影响。尽管洛伐他汀( lovastatin )和柳氮磺吡啶(sulfasalazine)等药物会被肠道细菌化学转化为活性形式,但细菌代谢可以使地高辛(digoxin)等药物失活,或者像伊立替康(irinotecan)一样引起毒性作用。为了进一步增加易感药物的多样性,最近报道,肠道细菌对一百多个分子进行了化学修饰。然而,这些相互作用的机制观点主要限于药物生物转化。
洛伐他汀(Lovastatin)
洛伐他汀(Lovastatin)可使胆固醇的合成减少,也使低密度脂蛋白受体合成增加,主要作用部位在肝脏,结果使血胆固醇和低密度脂蛋白胆固醇水平降低,由此对动脉粥样硬化和冠心病的防治产生作用(来源:百度百科)。
柳氮磺吡啶(sulfasalazine)
柳氮磺吡啶(sulfasalazine)磺胺类抗菌药。属口服不易吸收的磺胺药,吸收部分在肠微生物作用下分解成5-氨基水杨酸和磺胺吡啶。5-氨基水杨酸与肠壁结缔组织络合后较长时间停留在肠壁组织中起到抗菌消炎和免疫抑制作用,如减少大肠埃希菌和梭状芽孢杆菌,同时抑制前列腺素的合成以及其他炎症介质白三烯的合成(来源:百度百科)。
地高辛( digoxin)
地高辛是中效强心苷类药物,为白色结晶或结晶性粉末;无臭;味苦。在治疗时,对心脏的作用表现为正性肌力作用, 减慢心率,抑制心脏传导。适用于低输出量型充血性心力衰竭、心房颤动、心房扑动、阵发性室上性心动过速。
肠道中的细菌可以调节治疗药物的可用性和功效。然而,药物与细菌之间相互作用潜在机制是微生物对药物的化学转化或生物转化。
德国海德堡和英国剑桥科学家团队刚刚发在《Nature》上,成功研究了 25 种代表性肠道细菌菌株对 15 种结构不同的药物的消耗。揭示了 70 种细菌和药物相互作用,其中 29 种以前没有报道过。超过一半的新相互作用可归因于生物积累。
肠道细菌的这种生物累积可能是改变药物可用性和细菌代谢的常见机制,该机制会对个体微生物群组成、药代动力学、副作用和药物反应产生影响。
为了更详细调查药物与细菌之间相互作用的系统图谱,科学家使用两种分析方法核磁共振(NMR)光谱和液相色谱-质谱联用(LC-MS)系统地分析了 15 种人类靶向药物和 25 种人类肠道细菌(长双歧杆菌、大肠杆菌和均匀拟杆菌的其他亚种或同种菌株之间的相互作用)。选择细菌种类以涵盖代表健康微生物群的广泛系统发育和代谢多样性。在药物方面,选择了 12 种口服小分子药物(分子量小于 500 Da),这些药物可以基于超高效液相色谱结合紫外检测(UPLC-UV)进行定量,以跨越不同的化学,适应症区域和副作用概况。
细菌-药物相互作用网络。左网络:肠道细菌对药物的生物转化或生物累积。右网络:至少在两个独立筛选中检测到的药物对肠道细菌生长的影响(Student’s t检验,α = 0.05)。文中未提及的细菌全名如下:脆弱拟杆菌、普通拟杆菌、动物双歧杆菌亚种。乳杆菌BI-07, Coprococcus come ,梭菌,梭菌,副干酪乳杆菌, Ruminococcus gnavus。
谷禾健康
收样安排
五一节放假时间为4月30-5月4日。
五一期间接收快递,但实验室不接收样本进行实验。4月30-5月4日收到样本会在5号进行实验。

杭州谷禾信息技术有限公司
2022年4月29日

谷禾健康
疾病表现、进展和治疗反应的可变性一直是医学的核心挑战。尽管宿主因素和遗传学的变异性很重要,但很明显,在迈向个性化治疗的过程中,必须考虑肠道微生物组具有巨大的遗传和代谢多样性。
疾病表现、治疗反应和治疗不良反应的个体差异是有效管理疾病和患者安全的主要挑战。这种认知是精准医学的基础,其最简单的形式可以这么说,用个性化方法为合适的患者确定合适的治疗方法,无需反复试验。
将肠道微生物组与人类遗传学区分开来的一个方面是它代表了我们健康的动态组成部分,通过复杂的网络不断与宿主和环境因素相互作用。虽然存在潜在挑战,肠道微生物组的可塑性也提供了一个独特的机会,使其成为精准医学的一个有吸引力的目标。
本文支持使用肠道微生物组作为精准医学工具的当前证据,并建议未来需要将微生物组作为个体化治疗或干预工具的工作。
该研究团队选择了六个广泛的疾病组,这些组具有相对较强的证据表明肠道微生物组的作用。 尽管每个疾病组都有不错的发展,但在考虑临床影响时,不同疾病组的前景和成熟度各不相同(下图)。
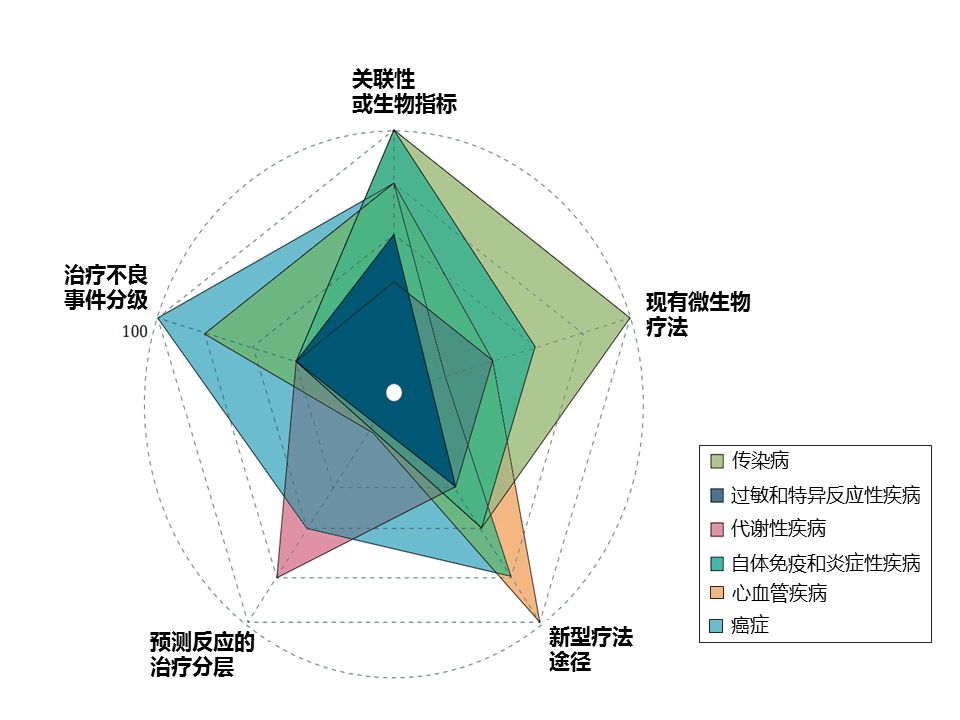
抗生素诱导的肠道微生物组破坏会促进机会性和医院感染的机制。最常见的院内腹泻感染艰难梭菌为例,强调可能解释临床结果的个体差异的微生物组和病原体特异性特征。
复发性艰难梭菌感染 (CDI) 一直是微生物组研究的中心焦点。CDI 出现最常见的原因是使用抗生素,但矛盾的是,CDI 的一线治疗也包括抗生素。
抗生素对一般人相当有效,但为什么部分患者出现治疗失败,或是成功治疗后复发?
这可能与宿主特征(例如高龄)或药物的使用(例如质子泵抑制剂)有关, 以及肠道微生物组中特定病原体的特征有关。
除了宿主因素外,肠道微生物群的破坏也是 CDI 的关键因素。
· 与健康对照个体相比,CDI患者的肠球菌、韦永氏菌、乳杆菌、γ-变形菌属的相对丰度较高,而拟杆菌属、毛螺菌科、瘤胃球菌科的含量较低。

谷禾健康
普雷沃氏菌属存在于人类中,帮助分解蛋白质和碳水化合物食物。也可作为条件致病菌,引起牙周和牙齿、肠道炎症、类风湿性关节炎、细菌性阴道炎等问题。


普雷沃氏菌(Prevotella)是革兰氏阴性菌、非运动性、杆状、单细胞,可在厌氧条件下生长。
Prevotella是一个大属,包括50多个不同的物种。大多数可以从口腔和肠道中分离出来。


普雷沃氏菌有助于分解蛋白质和碳水化合物食物。
普雷沃氏菌可以通过结合或附着在上皮细胞以外的其他细菌上定植,在以前感染的区域产生更大的感染。
普雷沃氏菌的另一种生存机制是天然抗生素抗性基因,防止被消灭(TIGR)。


接下来,我们从肠道、口腔、阴道三个部位,来了解普雷沃氏菌与人体健康或疾病的关系。
普雷沃氏菌通常被认为是一种与健康的植物性饮食相关的菌,在人体中发挥“益生菌”的作用。普雷沃氏菌属的减少与某些疾病相关。
自闭症
被诊断患有自闭症的儿童的普雷沃氏菌属丰度较低。婴儿普雷沃氏菌水平的下降与两岁时的行为问题有关。
过敏
研究人员分析了从2010年到2015年收集的数据,这些数据观察了怀孕36周的母亲以及她们1个月、6个月和12个月大的婴儿的粪便样本。有食物过敏的儿童(58名)与没有食物过敏的儿童(258名)进行了比较。
他们发现,20%没有过敏的婴儿的粪便样本中含有P.copri,而过敏婴儿的粪便样本中只有8%含有P.copri:母亲的粪便样本中含有P.copri,意味着婴儿产生过敏的风险更低。
多发性硬化症
在多发性硬化症患者的肠道微生物群中,缺乏Prevotella histicola。
2019年开展的一项研究表明,单独使用Prevotella histicola治疗转基因小鼠或与多发性硬化药物Copaxone联合治疗,减少了与该疾病相关的促炎症反应。
因此,该菌可能在多发性硬化症(MS)患者中有潜在的治疗作用,多发性硬化症是一种影响大脑和神经的自身免疫性疾病。多发性硬化症患者通常表现为微生物群失衡。
普雷沃氏菌是健康肠道生物群中必不可少的菌。然而,这种细菌是不是越多越好?
答案是否定的。

谷禾健康
随着科学技术的巨大进步,产生了大量的“组学”数据。理解生物系统各个层次产生的大量序列和结构数据是关键,由此产生了“生物信息学”。
“生物信息学”是一个跨学科领域,主要是用计算算法来组装、评估、理解、可视化和归档与生物分子相关的数据。
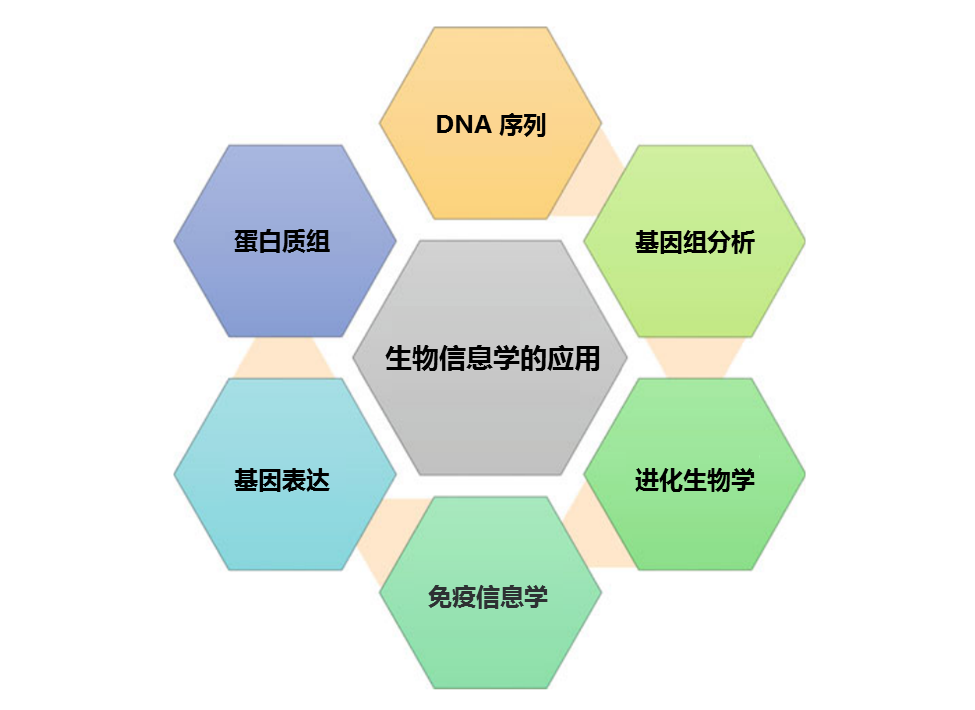
从基因组测序、基因及其功能预测到蛋白质分析,如蛋白质结构和功能预测、系统发育研究、药物和疫苗设计、生物体鉴定,以及支持和推进生物技术领域的研究,都需要用到生物信息学。
今天给大家推荐一本书《Advances in Bioinformatics》,帮助广大科研工作者更容易进行研究,从而对生物学有新的见解。

该书共23个章节。涵盖了蛋白质组学、代谢组学、DNA测序和NGS技术、基因组分析、生物计算、神经网络分析、大数据分析、软计算、人工智能到进化生物学、疫苗和药物设计、生物合成学和癌症生物学应用等,从这些领域出发,综述了生物信息学在其中的应用、发展、帮助和已经获得的成果,并探讨了未来的发展方向。
1. 生物信息学简介及其应用
生物信息学在DNA测序与分析、基因组测序及其注释分析、进化生物学的计算、比较基因组学、基因和蛋白表达分析、蛋白质和DNA、RNA的结构分析、免疫信息和药物设计的技术应用。
2. 生物信息学工具和软件
重点介绍了Banqit、Spin、WEBIN、Sequin、Sakura等序列提交软件;ADIT、PDB_Extract等分子结构提交软件;SRS、Entrez、Getentry等序列检索工具。此外,还详细讨论了BLAST、CLUSTALW/X等序列比对工具,以及Swiss-Model、Modeller、JPred、3D-Jigsaw和ModBase等结构预测工具。
3. 生物信息学在生物科学中起到的作用
重点介绍了生物信息学在基因组学、转录组学、蛋白质组学和代谢组学等主要“组学”领域的作用,以及在其它领域,营养基因组学、化学信息学、分子系统发生学、系统学和合成生物学的应用。还讨论了生物信息学在这些领域的多样化发展。
4. 蛋白质分析:从序列到结构
介绍与蛋白质序列和结构分析相关的各种数据库和方法。这类研究的主要应用之一是在药物发现和开发方面。
5. 进化生物学
进化是一个物种或种群经历遗传特征变化的动态过程。对进化的研究被称为进化生物学。进化生物学研究的关键是序列变异,这是通过比较DNA或蛋白质序列来检测的。迄今以及开发了不同的计算工具来比对所获得的序列和识别序列变异。进化基因组学的应用正在从研究人类进化到研究各种病毒的进化。许多病毒对人类健康具有严重威胁。本章介绍了PAML、PhyML、MrBayes、RAxML、MSA、MUSCLE 、MAFFT 等用于系统发育分析的计算工具。并详细讨论了进化的计算原理。
6. 基于web页面的调控序列分析的生物信息学方法
本章主要概述了在线分析哺乳动物基因组中调控序列方法,以及用于调控序列分析的在线生物信息学工具。
7. 用于SNP分析的生物信息学资源综述
基因变异是导致生物物种内多样性的关键。单核苷酸多态性(SNPs)是遗传变异的主要形式。单核苷酸多态性在理解生物表型差异的进化过程中至关重要,而且还被用于各种疾病的诊断和治疗。本章详细介绍了用于人类和其他非人类基因组的SNP分析。此外,还讨论了在生物信息学领域需要解决的挑战和差距,以便在未来有效地研究SNPs。

谷禾健康


现如今,甲状腺疾病的发病率逐年递增,而女性更是甲状腺疾病的重灾区,据统计,每八名女性中就有一人患有甲状腺疾病。而甲状腺问题和肠道问题往往是齐头并进的。
本文我们将探讨肠道与甲状腺联系背后的科学事实,并提供有用的干预措施,助你走上改善肠道和甲状腺健康的道路。
甲状腺是位于颈部底部的一个小而重要的内分泌腺。
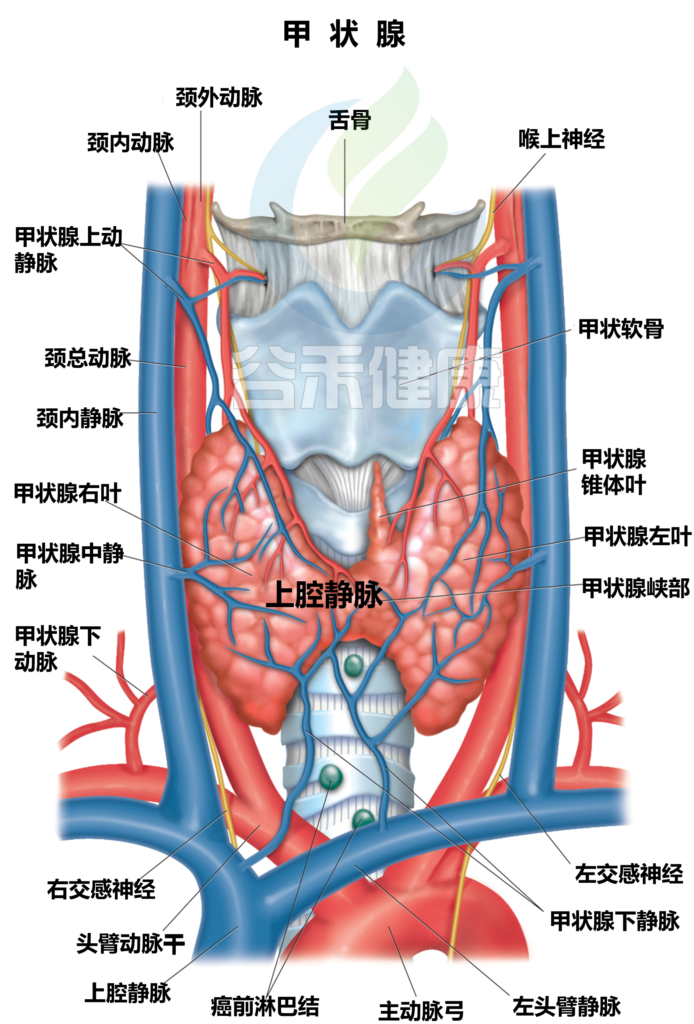
甲状腺如何参与调节新陈代谢?
甲状腺的工作是确保体内的细胞正常工作,通过激素来发送信息到每一个细胞。它生产两种重要的激素,分别是T4和T3(具体意思见本节末“名词小讲堂”),激素的工作是指导体内的细胞工作,何时该消耗氧气养分,从而维持体内新陈代谢的过程。
人体内的每个细胞都有这些激素的受体,甲状腺的作用包括:
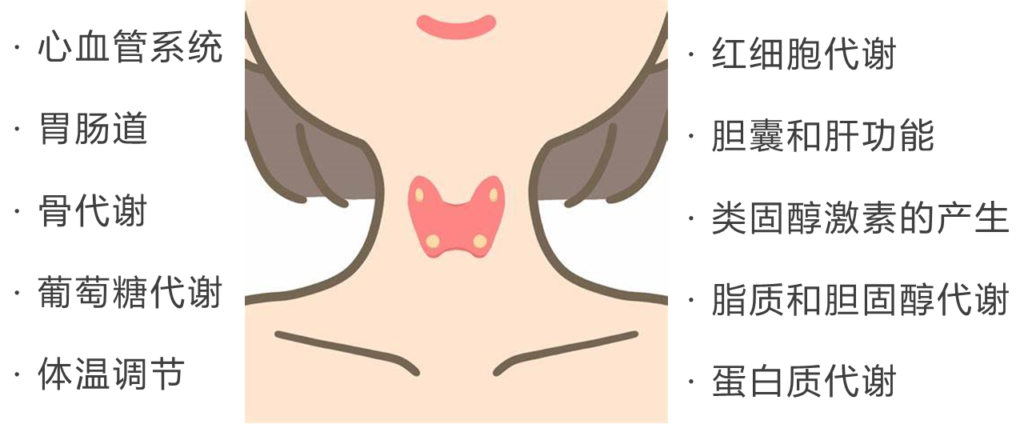
同时呢,甲状腺也是受约束的,它何时发送信息是被脑部的垂体监控着的,垂体会感受血液中的激素含量高低,以促甲状腺激素(TSH)的形式发出指示。可以看到,这是一个精密运作的系统。
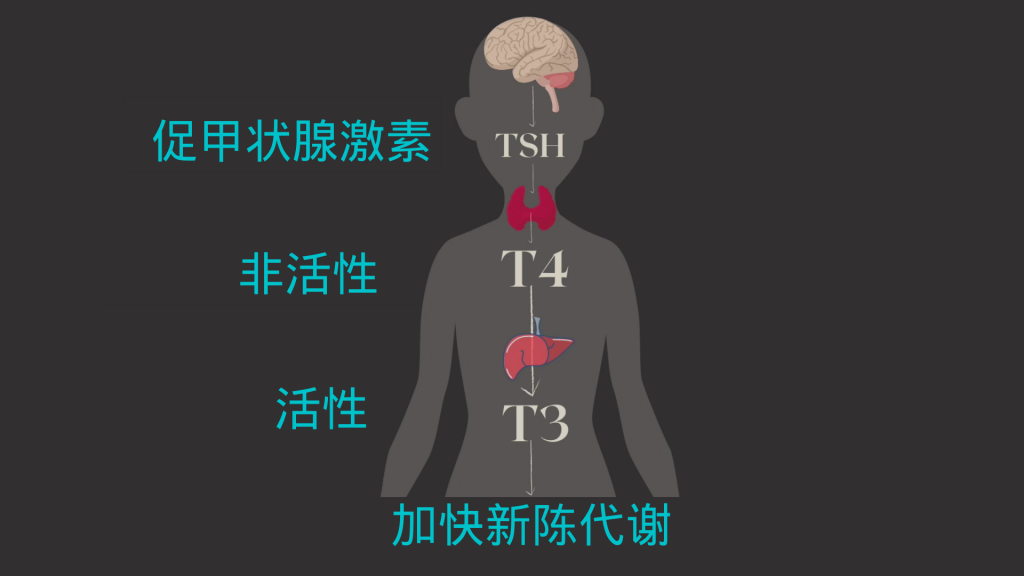

然而,甲状腺激素的产生、转化和摄取过程非常复杂,如果任何一步出错,就会出现甲状腺功异常的症状。
▲甲状腺过度活跃——甲亢
甲状腺过度活跃,发出太多激素时,就会出现甲亢,新陈代谢就会加快,表现为心率过快,持续饥饿,体重下降,出汗,焦虑,难以入睡。
▲甲状腺活动不足——甲状腺功能减退症
甲状腺活动不足时,就会出现甲状腺功能减退症,也就是甲状腺发出激素太少,没有足够的量去唤醒体内细胞,新陈代谢减慢,表现为体重增长,怕冷,关节肿大,感觉忧郁。
名词小讲堂:
促甲状腺激素释放激素 (TRH):这种激素在下丘脑中产生,它向垂体发出信号以产生多少促甲状腺激素 (TSH)。
促甲状腺激素(TSH):由垂体产生,TSH 刺激甲状腺产生甲状腺激素 T4 和 T3。TSH 水平升高可能表明甲状腺功能低下。需要注意的是,并非所有甲状腺功能减退症患者都符合这种模式。如果甲状腺激素水平低,TSH 水平低也可能表明存在问题。
甲状腺素 (T4) 和三碘甲状腺原氨酸 (T3):统称为“甲状腺激素”,T4 和 T3 由甲状腺产生。T4 是甲状腺激素的非活性形式,必须先转化为 T3,然后身体才能使用它。
总 T4 和 T3:它们与蛋白质载体结合,可以在血液中循环
游离 T4 和 T3:它们与载体分离,可以与细胞受体结合并发挥其功能 。
甲状腺结合球蛋白 (TBG): TBG 是与 T4 和 T3 结合并通过血液运输这些激素的蛋白质载体。过多的 TBG 会导致游离 T4 和 T3 水平降低。
甲状腺抗体:在自身免疫的情况下,这些抗体会攻击并损害甲状腺。我查看甲状腺过氧化物酶 (TPO) 抗体 (Ab)和甲状腺球蛋白 (Tg) Ab。这些抗体可以在临床甲状腺功能减退症发生前数年出现,因此及早发现它们尤为重要。
目前研究表明,良好的甲状腺健康始于肠道。随着我们对肠道微生物群的了解不断加深,可以看到研究表明,生活在人体内的微生物群在甲状腺功能减退症和自身免疫性甲状腺疾病中发挥着重要作用。
甲状腺疾病,例如桥本甲状腺炎或甲状腺功能减退症,通常与不孕症、荷尔蒙失衡、体重增加、疲劳或焦虑等症状有关。

谷禾健康
油炸是一种流行的烹饪方法,它使食物在质地和香味上更有吸引力,从而改善食物口感。


油炸食品通常被认为不健康的,因为油炸可能会增加食物的能量密度,从而增加能量摄入,并通过氧化和氢化过程使油变质,导致不饱和脂肪酸如亚油酸和亚麻酸的损失,但反式脂肪酸、油脂降解和晚期糖基化终产物的增加。
在流行病学研究中,油炸食品的高摄入量与多种不良健康后果有关(包括2型糖尿病),虽然结果并不完全一致。
可能原因:
油的类型
据报道,在主要使用橄榄油制备油炸食品的人群中,油炸食品和2型糖尿病风险之间没有关联,橄榄油比其他普通油(如玉米油)更抗氧化。
食物成分 、条件
被油炸食物的不同成分和油炸条件(温度、持续时间)也可能引发不同的结果。
混杂因素
各种混杂因素(如体重增加、高血压和高脂血症)增加了不一致观察结果的复杂性,这些因素与食用油炸食品和2型糖尿病风险相关。
综上,油炸食品和2型糖尿病之间的不良关联背后的潜在机制在很大程度上仍然未知。很多途径包括体重增加、炎症和脂质代谢等都有可能参与其中。
近年来,越来越多的数据表明,肠道微生物群可能在饮食因素(包括油炸食品)与宿主健康联系中发挥着关键作用。
对人类和动物模型的研究表明,油炸食品的摄入量或油炸和热加工的副产品与肠道微生物群的多样性和丰富性有关。然而,评估油炸食品对糖代谢影响的随机临床试验仍然缺乏。
近日,《糖尿病护理》杂志上,一项随机对照喂养试验旨在测试油炸肉类摄入对葡萄糖稳态的影响。
研究人员将117名年龄在18-35岁的青少年超重(BMI > 24 kg/m2)的成年人随机分为两组,分别提供等热量膳食和一致的食物,交替健康饮食指数(AHEI)评分>85。干预组是油炸,对照组是煮,蒸,或用调味汁拌。
葡萄糖代谢指数变化:
在两组的干预过程中,包括胰岛素生成指数(IGI)、肌肉胰岛素抵抗指数(MIRI)和胰岛素水平在内的几项葡萄糖代谢指数均有所改善,同时能量摄入也有所减少。
与对照组相比,油炸食品组的4周干预在IGI、MIRI和胰岛素曲线下面积(AUC)方面的改善较少,并且在HbA1c、C肽和葡萄糖的曲线下面积没有差异。作者得出结论,油炸肉的摄入损害了葡萄糖稳态。
肠内毒素和炎症标志物变化:
次要结果中,油炸肉干预组显示肠内毒素和全身炎症生物标志物减少较少,调节饱腹感和糖摄入的肝因子FGF21增加较少。
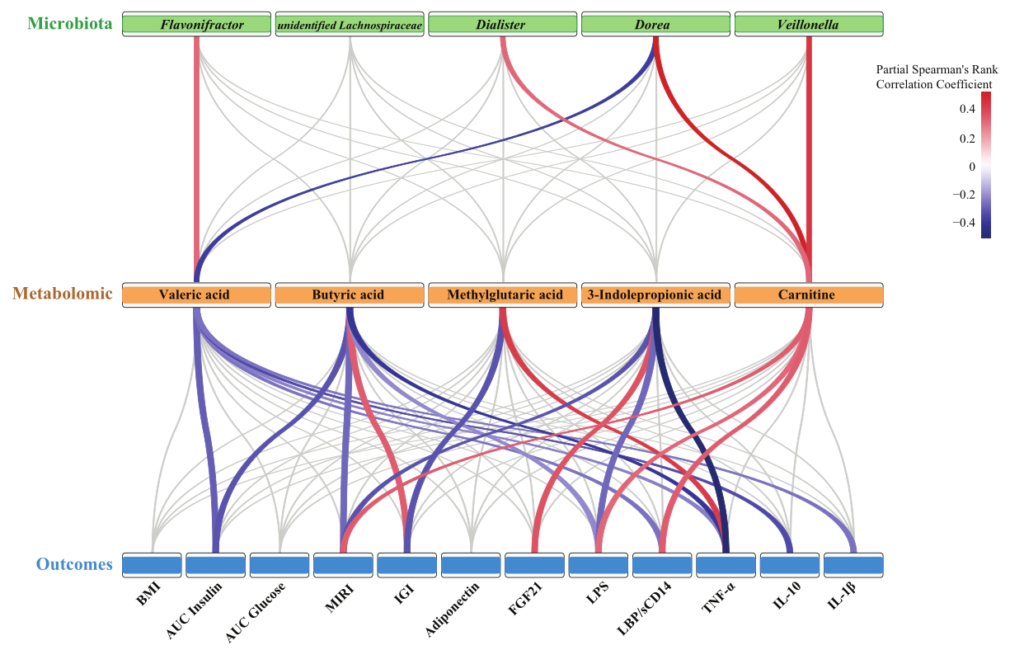
肠道微生物群变化:
发现油炸肉组的肠道微生物群丰富度低于对照组;总体微生物结构和组成以及微生物组预测的与葡萄糖稳态相关的途径在这两组之间也是不同的。
油炸组降低了Lachnospiraceae和Flavonifractor的丰度。
谷禾健康
在与细菌性疾病的斗争中,抗生素是不可避免的。抗生素是人类使用了几十年的治疗细菌感染的救命药物。

虽然抗生素挽救了数百万人的生命,但它们也会消灭一些常驻细菌。现有文献已经确定了与抗生素相关的肠道微生物群失调(多样性降低)。也就是说过度使用抗生素会影响肠道健康。
抗生素对肠道菌群的不同影响
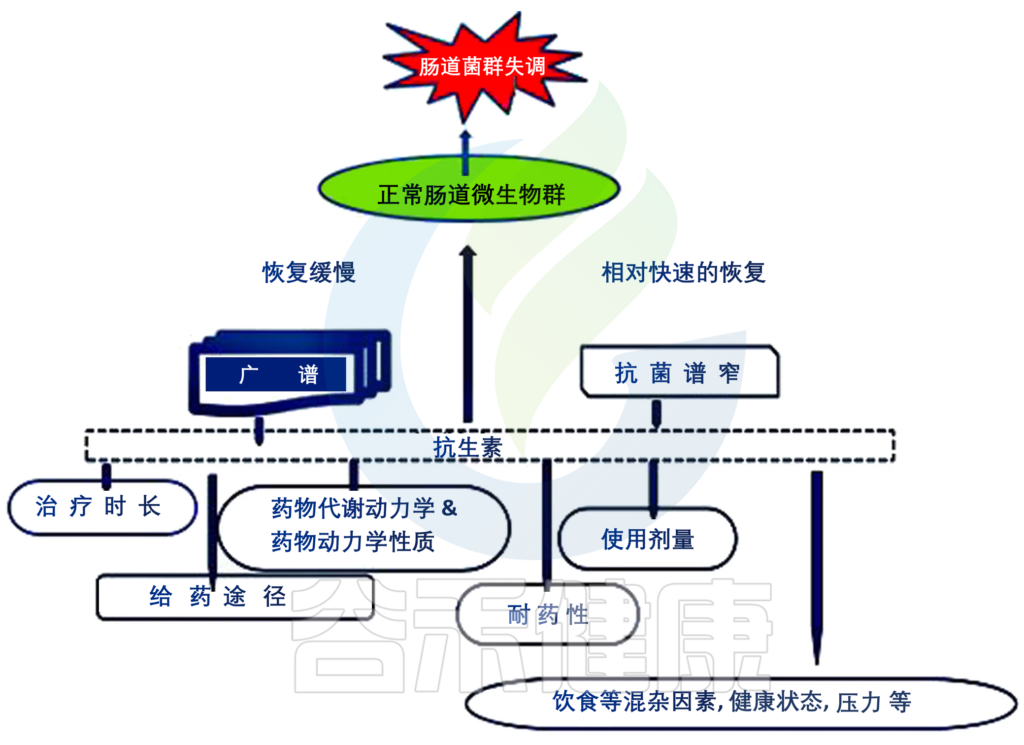
Yang L, et al., AMB Express. 2021
药物使用时间越短,肠道菌群恢复越快,反之亦然。然而,虽然一些物种将在几个月后恢复,但有些灭绝的物种在试验期间没有恢复。每个疗程中抗生素浓度的比例恢复是一种现象,它将从体积上说明所使用的抗生素的疗效。

♞ 抗生素滥用正导致耐药性危机日益增长
构成抗生素滥用的一种形式与食用动物有关。由于抗生素的过度使用,微生物的遗传能力随着耐药基因的增加而增加。显然,细菌对几乎所有可用的抗生素都产生了多种机制的耐药性。
抗生素滥用所造成的神经毒性效应,既有局部作用,也有更显著的全身作用。
滥用抗生素的不良反应除了增加微生物耐药性之外,还有微生物群异常,所谓微生物群异常也就是菌群失调。
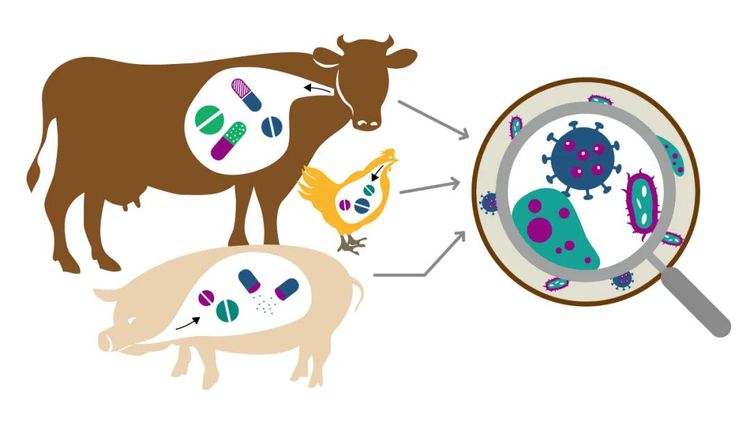
☟ 菌群失调
可导致自身免疫性疾病、胃肠道疾病、过敏、感染、关节炎、哮喘、癌症、肥胖等疾病。
过量服用抗生素会改变肠道微生物群,据研究,这与神经和精神疾病的发展有关。在控制抗生素滥用和耐药性的同时,为了减少抗生素的使用,抗菌肽(AMPs)与一些传统抗生素联合使用是有前景的。
从最近的研究来看,水平基因转移(HGT)和抗生素耐药基因在胃肠道中大量发生。这带来了许多临床意义,因为益生菌、共生菌和条件致病菌可以通过转移耐药基因在抗菌药物中生存。
◢ 在耐药性癫痫患者中的菌群失调
由于肠道生态失调被发现与黏液消耗、炎症、血脑屏障破坏导致神经调节剂改变有关,与药物敏感患者和健康对照相比,耐药性癫痫患者的肠道微生物组组成发生了改变。
低碳水化合物饮食疗法通过改变肠道微生物组减少一些耐药性癫痫患者的癫痫发作。益生菌补充剂通过改变微生物群的组成,可以减少某些患者的癫痫发作。
◢ 健康人肠道肠杆菌科出现替加环素耐药性
在新加坡健康人群肠道微生物群的队列研究中,通过替加环素灭活酶Tet (X4)对宿主的作用检测到高水平的替加环素耐药性。大肠杆菌2EC1-1和94EC分别携带Tet(X4)、IncI1型质粒p2EC1-1和p94EC-2。产 Tet(X4) 肠杆菌科细菌的粪便携带率为 10.1%(95% CI = 5.1%-17.3%),表明 Tet(X4) 广泛分布于新加坡健康个体的肠道微生物群。
接下来我们详细了解下,目前常用的抗生素对肠道菌群会带来哪些影响。
人类肠道微生物群在平均稳定的群落中是的,而对所有物种来说都是没有弹性的。从某种意义上说,就像突变的效果一样,一旦基因发生突变,就不会有恢复。因此,在使用后果不明的药物削弱肠道微生物群时,必须高度谨慎。
表1 抗生素使用对肠道菌群变化的不同影响

克林霉素
克林霉素是常用的广谱抗生素之一。它主要针对对维持肠道健康至关重要的厌氧菌。