-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com


谷禾健康

从生命早期到成年,微生物群对婴儿的健康起着至关重要的作用。生命早期的微生物群不仅是婴儿健康的关键调节剂,而且与长期健康有关。怀孕到生命早期是婴儿微生物群建立的黄金时期,受环境和遗传因素的影响。
最近,关于微生物群在人类疾病中作用的 研究呈爆炸式增长,但在疾病或健康方面的应用相对有限,因为人类微生物群的许多方面仍然存在争议,尤其是婴儿微生物群。
在怀孕期间,母体微生物群会影响胎儿的发育,尤其是大脑发育,如子宫微生物群、阴道微生物群、胃肠道微生物群、胎盘微生物群(有争议的)和口腔微生物群。值得注意的是,母体微生物群的紊乱会导致不良妊娠结局,严重威胁后代的健康。出生后,受环境和遗传因素影响的婴儿微生物群迅速建立,以确保健康成长。
孕期母体微生物群
以前,胎儿宫内感染的罪魁祸首被认为是来自阴道的微生物,如细菌、病毒和真菌。然而,随着科学技术的发展,人们发现口腔和肠道微生物群也与胎儿的健康有关,因为这些部位的微生物群可以通过血液传播。
在怀孕期间,母亲的肠道、口腔和阴道微生物群都会发生变化。这些变化相关的各种因素,包括饮食,抗生素的使用,感染,应激和宿主基因(下图)。
影响母婴微生物群的因素以及微生物群调节的机会窗口

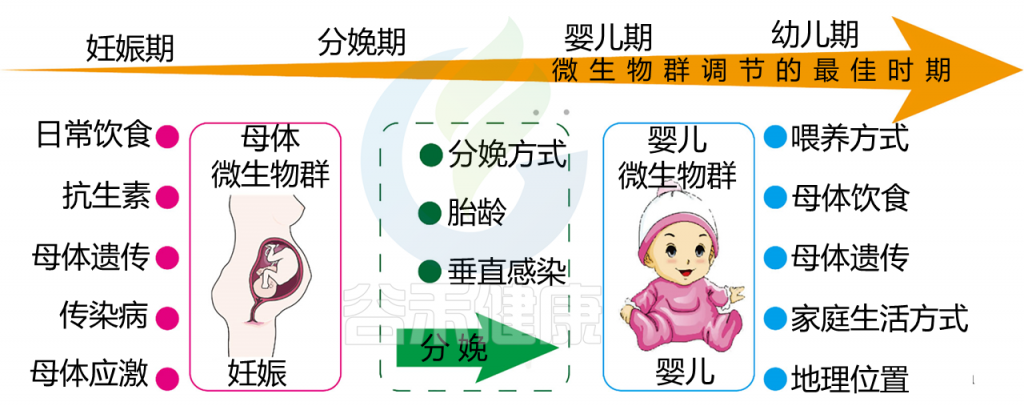
Yao Y, et al. Front Immunol. 2021
研究已经揭示健康孕妇阴道微生物群的稳定性高于健康非孕妇,乳酸菌是健康孕妇阴道微生物群的主要成分。传统上,子宫被认为是无菌的,但这个概念直到最近几年才被打破。子宫内膜有自己的微生物群,尽管这些微生物群的生物量比较低。遗憾的是,目前对子宫内膜微生物群的了解有限,其在胎儿发育和妊娠结局中的作用仍有待充分阐明。
新出现的证据表明,孕妇肠道菌群的丰富度和均一性与正常女性没有显着差异,但分布和组成发生了明显变化。此外,患有妊娠并发症的孕妇肠道微生物群的多样性降低,这对母亲和胎儿的健康都是不利的。例如,妊娠期慢性高血压大鼠的肠道微生物重塑受损。先兆子痫孕妇肠道微生物群中产生短链脂肪酸的粪球菌的丰度降低。
谷禾健康
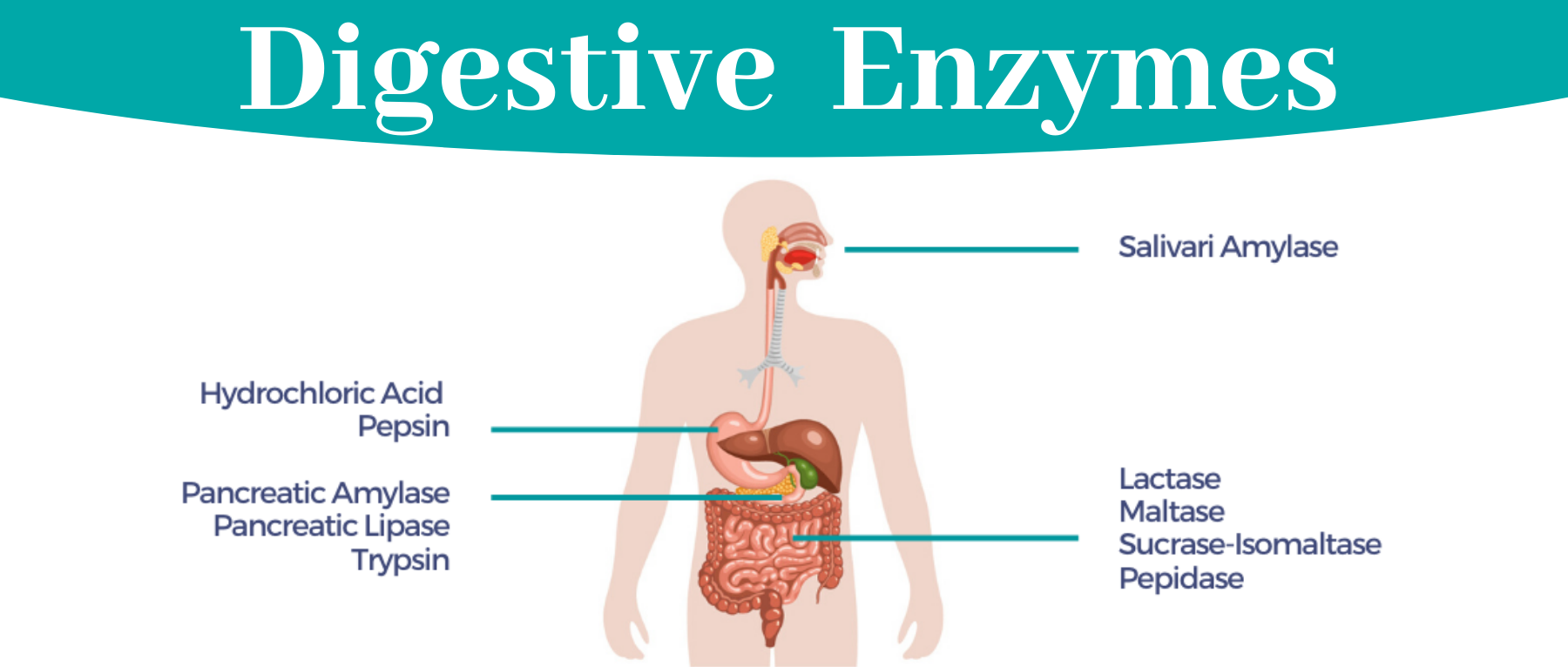
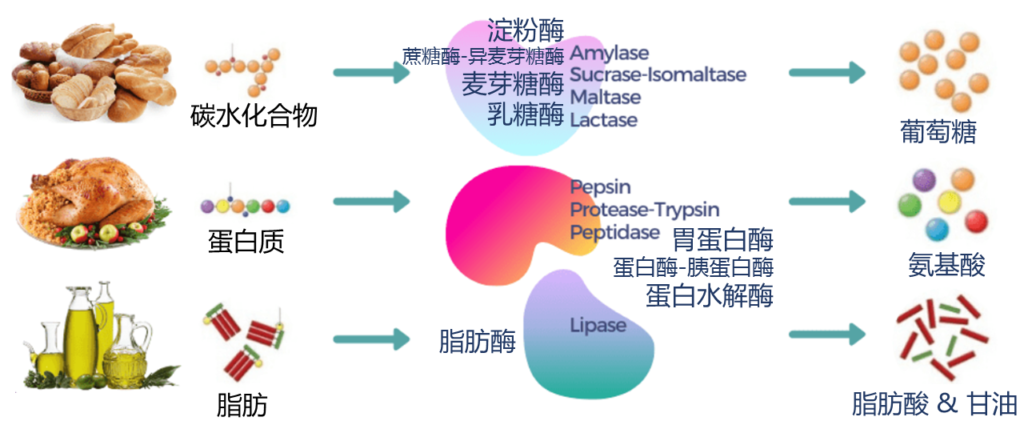
消化酶分解脂肪、蛋白质和碳水化合物,便于身体吸收营养。大部分消化酶由胰腺产生,其次是小肠、胃和口腔。
本文主要介绍不同酶的工作原理、酶水平背后的遗传因素和消化系统疾病以及自然促进消化的方法。
消化酶是一组广泛的酶,可将脂肪、蛋白质和碳水化合物等大营养素分解成更容易被身体吸收的较小营养素。人体在胰腺中自然产生大部分酶,而胃、小肠和口腔中产生少量酶。
以下酶可以帮助人体从特定的食物中吸收营养:
▪ 蛋白酶把蛋白质分解成氨基酸
▪ 脂肪酶把脂肪分解成脂肪酸
▪ 淀粉酶将碳水化合物分解成单糖,如葡萄糖
它们的主要作用是帮助消化,这些酶在你体内的数量和活性取决于一系列复杂的因素。初步研究表明,消化酶可能有助于缓解各种消化疾病、减轻炎症、肠道感染等。
消化酶水平低表明胰腺不能正常工作,难以产生重要的消化化合物。
在严重的时候,这种情况被称为外分泌胰腺功能不全(EPI)。这时,胰腺中产生消化酶的细胞会随着时间的推移而被破坏。
当然,EPI不是唯一的原因,各种因素都会导致消化酶水平降低:

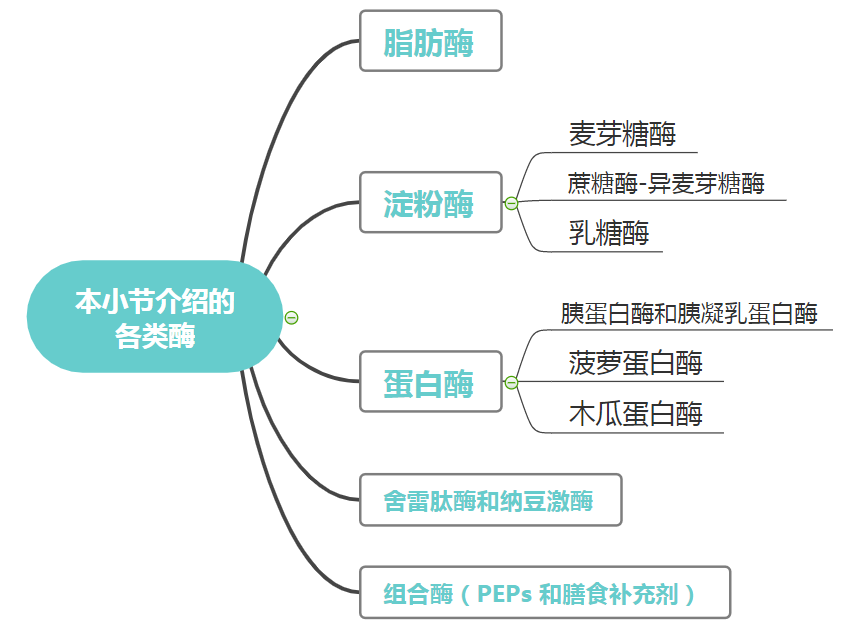
消化酶主要分三大类:脂肪酶、淀粉酶、蛋白酶
脂肪酶
脂肪酶是分解脂肪的酶,帮助维持胆囊功能正常。它们在胰腺中产生,但也可以从植物、动物和真菌中提取,并且足够稳定。
各种类型的脂肪酶参与不同的过程,例如脂肪代谢、运输、细胞信号传导、炎症等。

✓ 用于肠易激综合征(IBS)患者
含有脂肪酶和其他胰酶的补充剂可以帮助减少餐后的腹胀、胀气和饱腹感,尤其是脂肪含量高的食物。这些症状通常与消化问题有关,如肠易激综合征(IBS)。研究还表明,一些肠易激综合征患者可能存在胰腺外分泌功能不全,即由于胰腺产生的消化酶缺乏而无法正确消化食物。

谷禾健康

近年来,多重耐药病原体数量的增加以及食品安全已成为全球面临的严重问题,寻找或开发新一代抗菌药物或保存剂变得越来越重要。科学家们发现细菌产生的细菌素,能够控制临床相关的敏感菌和耐药菌,纯化的细菌素可以作为天然防腐剂或保存剂添加到食品中。细菌素可以作为抗病原体添加剂添加到动物饲料中,以保护牲畜免受病原体损害。医学上细菌素具有替代抗生素作为抗菌药物的潜力,是一种新型的抗癌药物。
本文将详细介绍细菌素相关的知识,包括:什么是细菌素、分类、生态与进化、抗病原菌以及抗病机制、食品保存和控制食物中毒发展、制药和替代抗生素潜力、益生菌特性、重塑肠道菌群以及影响健康的目标。
细菌素是一种小的、热稳定的、核糖体合成的抗菌肽,由细菌产生,对其他细菌具有活性,并且生产者对其免疫。它们在大小、结构、作用机制、抑制谱、免疫机制和靶细胞受体方面表现出相当大的多样性。
它们表现出针对产生它们的相同细菌菌株或针对密切相关物种的菌株的抗微生物活性。细菌素的合成发生在位于质粒或染色体 DNA 中的基因的控制下,这些基因同时包含生产者对产生的细菌素的抗性的遗传决定因素。
编码活性蛋白质的基因,编码蛋白质抗性的基因,负责细菌素从细胞中输出的基因,偶尔编码参与细菌素翻译后修饰酶的基因同时表达。
细菌素由革兰氏阳性菌(乳酸杆菌、乳球菌、链球菌、肠球菌、明串珠菌、片球菌和丙酸杆菌)和革兰氏阴性菌(大肠杆菌、志贺氏菌、沙雷氏菌、克雷伯菌和假单胞菌)产生。
“细菌素(Bacteriocin)”是指由任何类型的细菌产生的有毒蛋白质或肽,它对相关细菌有活性,但不会伤害生产细胞。这是描述的第一种由大肠杆菌产生的细菌素。在这种情况下,后缀cin被添加到生产物种中,例如pyocins来自Pseudomonas pyocyanea。属名也被用来命名细菌,例如klebicins(来自克雷伯氏菌),lactococcins(来自乳球菌)。尽管细菌素对细菌具有毒性,但不应将其与“毒素”(外毒素)混淆。
肠菌素是第一个被鉴定的细菌素,由发现者André Gratia 于 1925 年命名,当时他注意到一种大肠杆菌菌株产生一种有毒的可扩散物质,可杀死邻近的大肠杆菌。
从那时起,已经描述了成百上千种肽和蛋白质细菌素,它们是由革兰氏阴性和革兰氏阳性细菌制成的各种天然抗菌化合物库的一部分,以抵御竞争对手。为了与细菌素研究的起源保持一致,大肠菌素仍然是研究最多的,特别是在细菌素如何破坏细菌强大的防御机制方面。
大肠杆菌素通过多种机制杀死细胞,这些机制分为两种细胞毒性类别;酶促大肠菌素切割核酸或肽聚糖前体,而成孔大肠菌素使细胞质膜去极化。
在复杂和过度拥挤的环境中,微生物顽强地相互竞争领土和营养,因此形成了过多的防御机制。其中,细菌素被认为是目前分布最广的机制。根据定义,细菌素是原核来源的分泌的、核糖体合成的肽,具有抗菌特性。
从人类健康的角度来看,细菌素代表了一个经过 30 亿年进化磨练的潜在先导化合物库。它们的靶标范围窄、活性高、稳定性惊人且毒性低,使其成为现有小分子抗生素的可行替代品或补充品。它们使用这些强大的武器在微生物战争中茁壮成长。为了完成这个武器库,产生细菌素的菌株被赋予了有效的策略来逃避被自己的毒素杀死。大多数细菌素在皮摩尔或纳摩尔范围内具有活性,并且靶向在系统发育上与生产菌株接近的细菌物种,尽管有些细菌素表现出更广泛的活性谱。
事实上,例如,许多细菌素的活性谱很窄,对与生产者密切相关的菌株表现出抗菌活性,而其他细菌素则表现出对多种不同属的抗菌活性。细菌素生产的调节可能很复杂,在某些情况下会受到环境条件的影响,例如 pH 值、温度和生长培养基等。
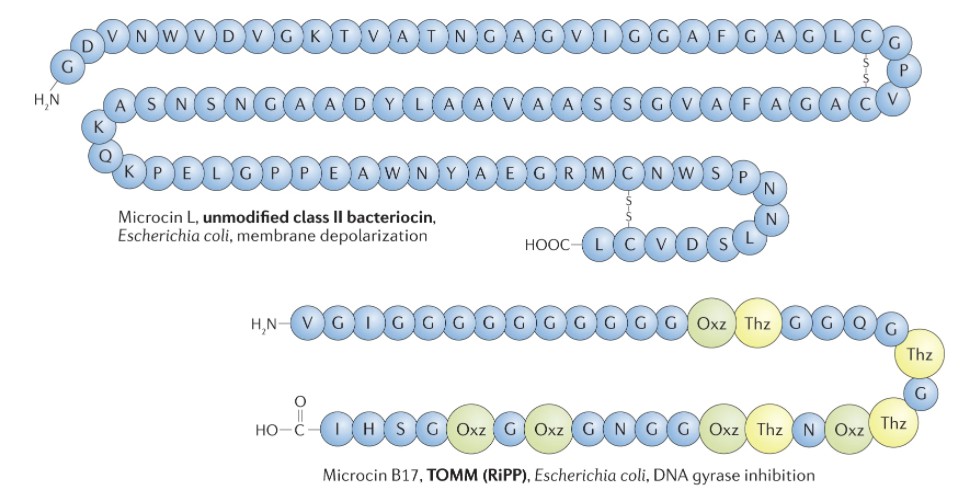
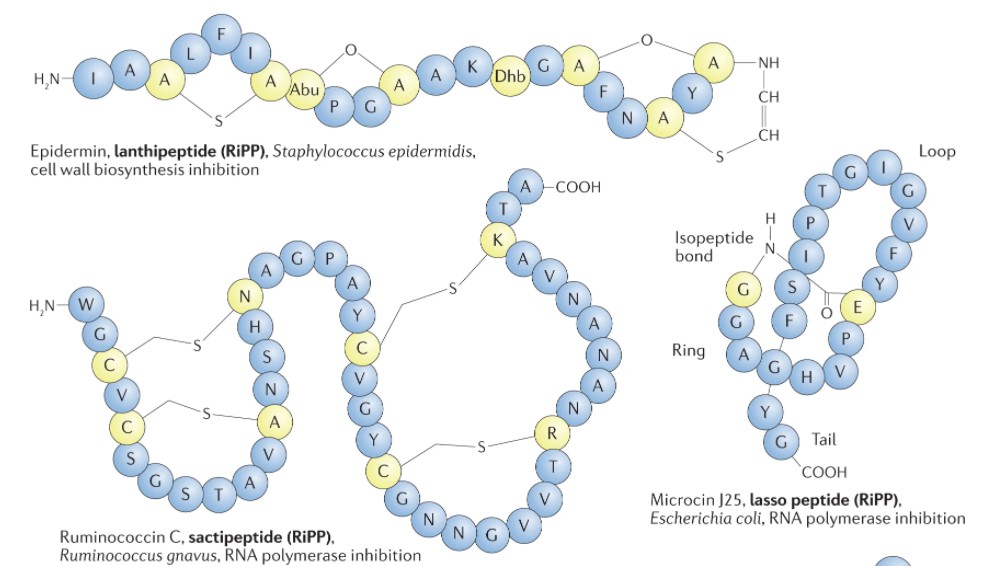
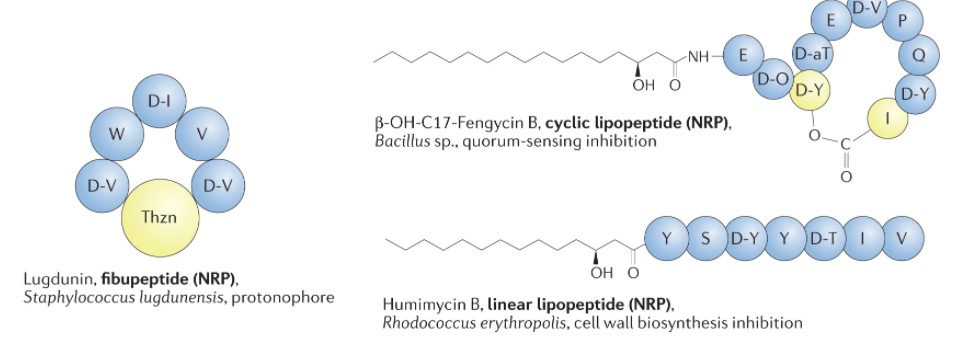
Heilbronner S, et al., Nat Rev Microbiol. 2021

谷禾健康
乳酸菌属益生菌是使用最广泛的益生菌之一。罗伊氏乳杆菌( L. reuteri ) 是一种经过充分研究的益生菌,可以在大量哺乳动物中定殖。
罗伊氏乳杆菌是一种革兰氏阳性杆状细菌,已在各种食物中发现,尤其是肉类和奶制品。在人类中,罗伊氏乳杆菌存在于不同的身体部位,包括胃肠道、泌尿道、皮肤和母乳。罗伊氏乳杆菌的丰度因个体而异。
可能的优势
● 可能促进皮肤光泽和头发浓密
● 可能有助于对抗感染
● 可能减少炎症并加强免疫
● 可能会改善肠道健康(减少 IBS 和 IBD 症状)
● 可能降低胆固醇
可能的副作用
● 可能对组胺不耐受的人不利
● 可能导致某些人体重增加
● 缺乏大规模临床研究
● 长期补充的安全性未知
罗伊氏乳杆菌(Lactobacillus reuteri)名字来自德国微生物学家 Gerhard Reuter,他在1960 年代在人类肠道和粪便样本中发现了它。1960 年代被发现时,罗伊氏乳杆菌自然存在于 30-40% 的人体内,现在大约降至10-20%。科学研究者将这种下降与生活方式的改变联系起来。我们不像以前那样吃发酵食品,如酸菜,而是使用防腐剂,这会杀死食物和体内的细菌。
罗伊氏乳杆菌在发酵过程中能够产生葡聚糖和果聚糖。其中一种葡聚糖,α-1,4/1,6 葡聚糖,分子量为 40 MDa,支化度约为 16%,似乎是一种饱腹感诱导剂,对胰岛素和血糖水平有良好的影响在人类。葡聚糖不会在胃和空肠中降解,而是在结肠中完全降解。由于其慢淀粉特性,这种葡聚糖可能是烘焙应用中一种促进健康的成分。
不同菌株的Lactobacillus reuteri已被证明具有不同的生理作用。例如,Lactobacillus reuteri DSMZ 17648用于治疗幽门螺杆菌(H. pylori),而Lactobacillus reuteriNCIMB 30242 用于治疗高胆固醇。
但是,罗伊氏乳杆菌补充剂尚未获得批准用于医疗用途,而且有的益处和副作用缺乏可靠的临床研究。法规制定了补充剂的制造标准,但不保证它们是安全或有效的。
大多数关于Lactobacillus reuteri的研究是在动物或细胞中进行的。临床研究很少,而且大多数是低质量的、小规模的或可能存在偏见的。此外,使用的确切菌株因不同研究而异。
尽管罗伊氏乳杆菌在人类中是正常的,但并不是每个人的胃肠道中都有它。口服补充剂可以增加和补充胃肠道罗伊氏乳杆菌,然而它不一定会长期留在那里。
同样,罗伊氏乳杆菌的良好来源是乳制品和肉类,素食者和大多数素食者都避免食用这些食品,因此补充剂很重要。在母乳喂养时服用罗伊氏乳杆菌补充剂的女性更有可能将这些有益细菌转移给婴儿。
为消化和吸收而建,胃肠系统的某些部位已发展为对微生物定植不利。这方面的例子可以在由小肠上部胃酸和胆汁盐引起的低 pH 条件下看到。因此,在胃肠道定植的第一步就是在这样的环境中生存。幸运的是,罗伊氏乳杆菌对低 pH 值和胆汁盐具有抵抗力。这种抗性被认为至少部分取决于其形成生物膜的能力。
谷禾健康
自身免疫性疾病,包括炎症性肠病、多发性硬化和类风湿性关节炎,具有不同的临床表现,但肠道微生物群紊乱和肠道屏障功能障碍的潜在模式相同。但宿主因素的个体差异使得统一的方法不太可能。
我们往往会有这样的错觉:“自然”的东西一定是对健康有益的。我们是不是只要吃最原始的食物就能恢复健康?
事实上,优化人类健康的途径并不像照搬我们祖先的饮食或增加我们对微生物的接触那样简单。归根结底,过去和现在的环境是根本不同的,也许是不可逆转的。
因此,针对微生物群的饮食干预的目标不应是回到祖先的状态,而是操纵微生物群,优化宿主健康,直接适应日益工业化的世界。
本文介绍了关于饮食-局部炎症中的微生物群相互作用、肠道微生物群失衡和宿主免疫失调的知识。通过了解并结合个别饮食成分对微生物代谢输出和宿主生理的影响,研究了基于饮食的自身免疫性疾病预防和治疗的潜力。讨论了针对肠道微生物群的工具,如粪便微生物群移植、益生菌和正交小生境工程等。这些方法强调了在自身免疫性疾病不断增加的情况下,利用饮食精确操纵肠道微生物组的途径。
在肠道中,对微生物威胁作出适当反应的免疫平衡行为,对共生微生物和自身抗原的耐受,在生命早期尤其重要,这是定植微生物群和宿主免疫系统相互作用的窗口,引发促炎或抗炎倾向,可能对终身健康产生影响。
★ 婴儿期影响菌群的因素
看过我们文章的都知道,早期生活因素,如剖腹产、配方奶粉喂养和抗生素的使用,可能会干扰微生物群生长过程。
婴儿期母乳中的人乳低聚糖有助于双歧杆菌的定植,双歧杆菌在早期肠道微生物群中占主导地位,产生岩藻糖、乙酸盐、丙酮酸盐和1,2-丙二醇,通过交叉喂养支持微生物群的扩增,有助于对共生细菌的免疫耐受。
人类通常在6个月大时开始转变为固体食物饮食,自此菌群显著扩大,产生更多数量和种类的代谢物(例如,丁酸盐,它促进结肠粘液屏障的成熟,并阻止具有高致病潜力的细菌的生长)。
★ 抗性淀粉缓解系统性红斑狼疮
在一项涉及149名健康志愿者的研究中,脂多糖通过诱导TLR4耐受来抑制先天免疫过程。微生物抗原对TLR的不当刺激也可能影响自身免疫疾病的进展,并为其修复提供线索,如系统性红斑狼疮过度表达TLR7的小鼠模型中,通过添加抗性淀粉来改变微生物代谢产物和分类组成,狼疮进展得到缓解。
★ 微生物变化引起的免疫反应
在自身免疫性疾病患者中也发现了由微生物引起的适应性免疫反应失调。哺乳动物肠道中的共生微生物群,尤其是梭菌群IV和XIVa,它们将膳食纤维代谢成丁酸盐以诱导Treg细胞发育,与小鼠模型中的结肠炎严重程度呈负相关。
通常,自身免疫性疾病患者表现出产生耐受性IL-10的CD25 FOXP3 T细胞的丰度降低,自身反应性效应性T细胞亚群(如TH1细胞和TH17细胞)的丰度增加,从而使Treg细胞与效应性T细胞的比率偏离稳态水平。这些改变可能通过过度产生促炎细胞因子(如TNF和IL-17)进一步促进肠道通透性的增加,这些促炎细胞因子可调节紧密连接蛋白的表达。
自身免疫疾病共同的潜在驱动因素鼓励通过饮食干预或微生物群调节策略(下文讨论)来进行肠道微生物群工程,以将肠道微生物群转变为功能多样的“健康”状态。
饮食可直接影响肠道微生物群,调节其组成或代谢输出,从而可能促进疾病或形成稳态。
谷禾健康
在过去的研究中,健康科学的突破表明人类肠道菌群可以受到环境因素的影响(饮食、生活方式等),而环境因素反过来又可以影响人类健康和疾病。
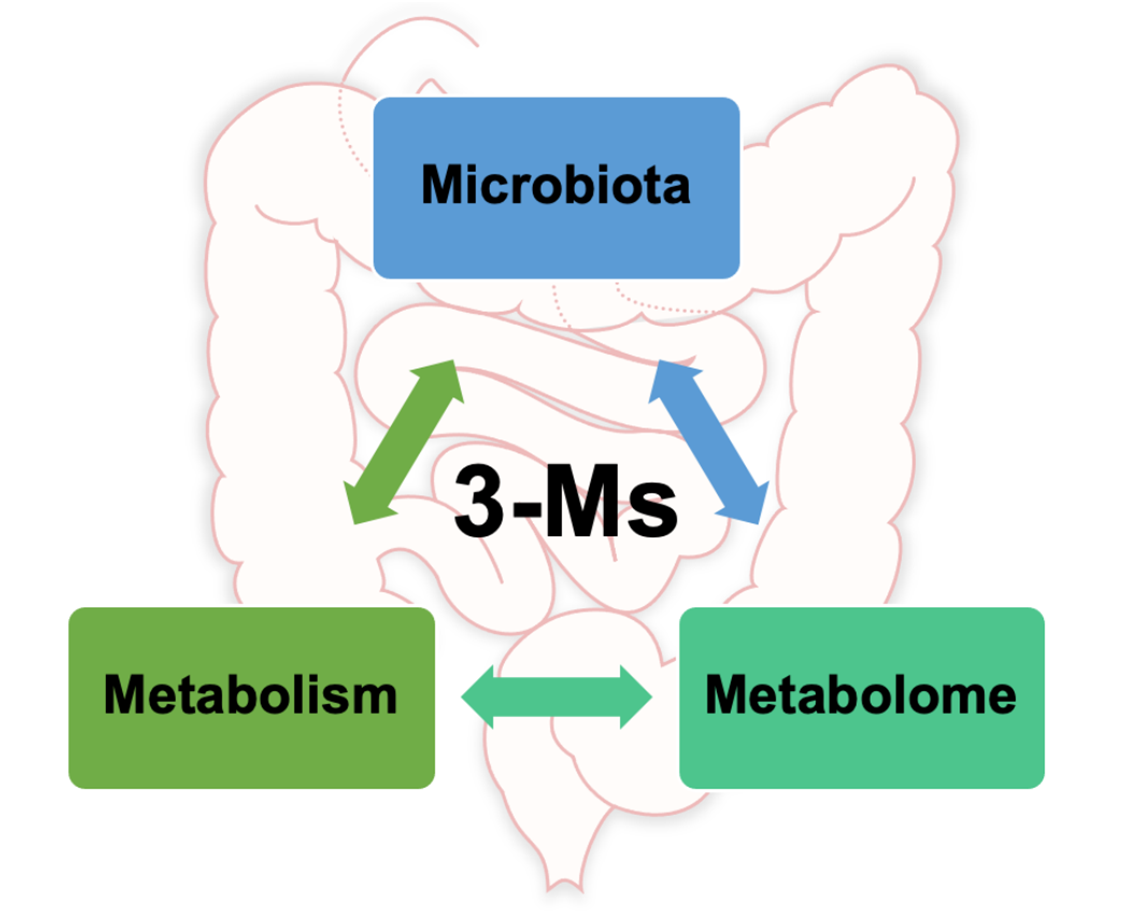
现在,更全新的概念肠道3M已经提出,即微生物组、代谢和代谢组(microbiota, metabolism, metabolome),对人类健康具有重要意义。
本文重点介绍3M相关的主要概念,提供了关于调节肠道健康的全面更新概述,有助于我们对肠道与人类健康和疾病之间的关系的基本理解。同时也定义了3M相互作用的挑战及讨论未来前景。
在阐述微生物群之前首先了解一下它的发展历史。
关于微生物群的历史
Dey P, et al., Free Radic Biol Med. 2021
人类微生物群
随着下一代高通量16S rRNA测序技术的出现,与人类健康和疾病相关的以微生物群为中心的研究有了巨大的增长。
数量
目前的估计表明,一个体重为70公斤的人平均含有39万亿细菌,而人体细胞则为30万亿。此外,在遗传水平上,在整个人类微生物群中基因数量超过1000万个基因,比人类基因(2万-2.5万)还要多很多,仅肠道微生物群基因就有330万。由于与生命各个方面相关的宿主-微生物相互作用的数量巨大,人类微生物群关联通常被称为“超有机体”。
分布
肠道细菌在肠道长度上的定植偏好是区域特异性的,可能是由缺氧肠腔内宿主的理化参数的变化(如pH值、营养有效性、pO2、胆汁酸、抗菌剂、粘蛋白)决定的。此外,在肠的特定区域内,管腔和绒毛隐窝之间的微生物种群多样性可能不同。比如说,厚壁菌和拟杆菌在盲肠中占优势,而隐窝主要由变形杆菌门和脱铁杆菌门定植。
“核心微生物群”
当然,由于与环境的不同相互作用,包括饮食模式的差异,文献中的微生物概况往往不一致。因此,人类微生物组项目的一个关键目标是确定一个“核心微生物组”,作为所有个体的普遍微生物分类学特征,该特征大部分保持不变。

谷禾健康
在过去的 20 年中,膳食(多)酚类化合物作为具有预防慢性疾病的抗氧化剂受到了关注。而临床试验中证据很弱,主要因为个体间的差异很大。多酚的生物利用度低(5%–10% 被小肠吸收)。这些化合物的生物利用度严格取决于肠道微生物的酶促机制。
最近,鉴定参与肠道多酚转化的细菌越来越受到关注。已经表征了几种催化酚类物质代谢的菌群及其分解代谢途径。大多数描述的参与多酚转化的属是双歧杆菌、拟杆菌和厚壁菌。微生物会受到外部因素(例如饮食、药物和体育锻炼)、宿主的地理分布和个体差异的影响。肠道微生物群组成的年龄相关变化可能会影响某些营养素的生物利用度,包括其代谢活性介质。
特定的代谢型会产生对健康影响的生物活性代谢物。代谢型还可以反映肠道微生物群的组成和代谢状态,并且可能是通过肠道微生物群介导的潜在多酚健康影响的生物标志物。
基于最近的研究成果,本文将综合介绍膳食多酚以及其生物利用转化与肠道菌群的互作对人体健康的影响,为我们如何结合肠道微生物群选择和摄入膳食多酚提供一些视野。
膳食多酚是一组生物活性植物化学物质,主要存在于各种水果、蔬菜、种子、草药和饮料(啤酒、葡萄酒、果汁、咖啡、茶和巧克力)中,少量存在于干豆类和谷物中。食物中,这些化合物具有多种生态作用,从抵御生物和非生物压力因素到界内交流。
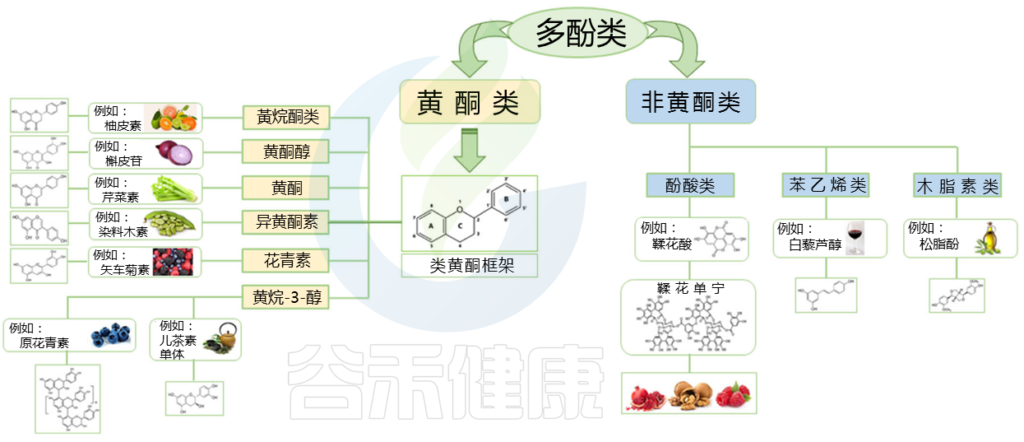
基于中心吡喃环的氧化态将黄铜类细分为几个亚类:
类黄酮类黄酮主要是黄烷醇、黄酮、花色素、黄烷酮、黄酮醇和异黄酮
非类黄酮
非黄酮类化合物主要类别是酚酸,可细分为苯甲酸衍生物,例如没食子酸和原儿茶酸,肉桂酸衍生物,包括香豆酸、咖啡酸、阿魏酸。
第二个主要组主要由芪类组成,白藜芦醇是主要代表,以顺式和反式异构形式存在。另一个重要的非黄酮类化合物是由两个苯丙烷单元氧化二聚产生的木脂素。
黄烷醇是食物中最常见的类黄酮形式,其最丰富的来源是洋葱、西兰花、茶、苹果、红酒、蓝莓、杏仁和开心果。
黄烷酮丰富的食物包括柑橘类水果,如柠檬、葡萄柚和橙子。
花青素是存在于红色水果和蔬菜(例如覆盆子、接骨木莓、草莓、石榴、卷心菜、红洋葱)中的水溶性黄酮类化合物。黄酮来源的有针叶樱桃、杏、橄榄油、蜂蜜、苹果、木瓜和芒果。
异黄酮是主要存在于豆科植物中的生物活性化合物,少量(浓度低于 0.1 毫克/千克)存在于杏、干枣、醋栗、芒果、李子、新鲜椰子和芝麻,而发现二苯乙烯存在于葡萄、红酒和浆果中。
红色、深色水果和蔬菜,如草莓和黑莓、黑萝卜、洋葱和茶,是也是酚酸的重要来源。
红葡萄酒中常含有芪,豆制品中常含有异黄酮。 亚麻籽和芸苔属蔬菜中含有大量木脂素; 其他,如松脂醇、落叶松脂醇通常也存在于许多食物中。
Davinelli S &Scapagnini G. Biofactors. 2021
谷禾健康
在开始本文之前,首先祝祖国母亲生日快乐!
是祖国的繁荣昌盛
成就我们无限可能
目光所致皆为华夏
五星闪耀皆为信仰


-正文-
我们知道肠道微生物群影响健康。然而肠道的内部工作机制较为复杂,不仅有各种微生物,也包括它们产生的气体,比如二氧化碳、氢、甲烷和硫化氢以及各种微量气体,都是由肠道内的化学相互作用和微生物群产生的。
分析这些肠道气体及其对饮食变化的反应可以揭示肠道微生物群的产物和功能及其对人类健康的影响。更好地理解肠道内产生气体的复杂相互作用将提高我们预防、诊断、治疗和监测许多胃肠道疾病的能力。
本文主要概述了肠道气体的生理学及其在胃肠道疾病中的作用,并讨论了目前对肠道主要气体成分、其产生来源及其与饮食摄入的关系的认识。
了解肠道气体的生理学,无论是由微生物发酵还是化学活动产生的,是了解其对人类健康影响的第一步。
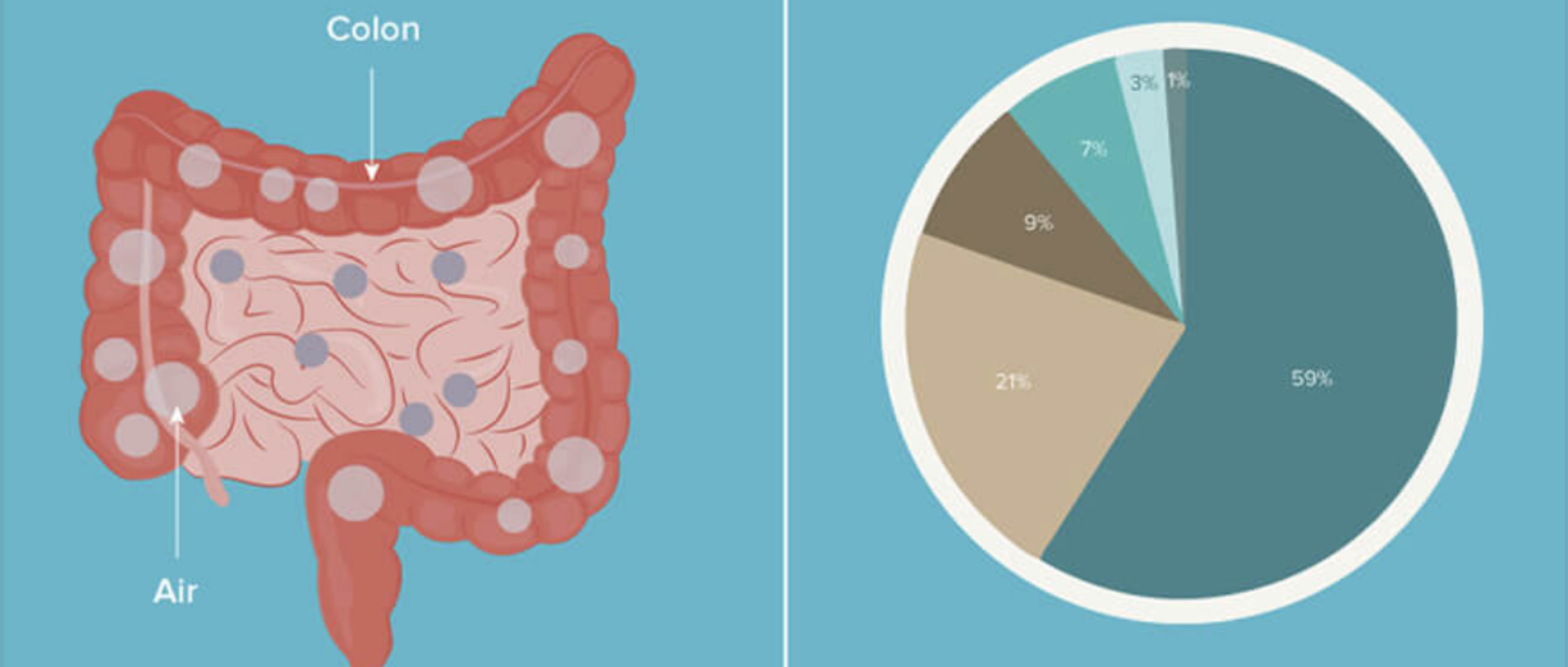
气体和食物沿着肠道的运输
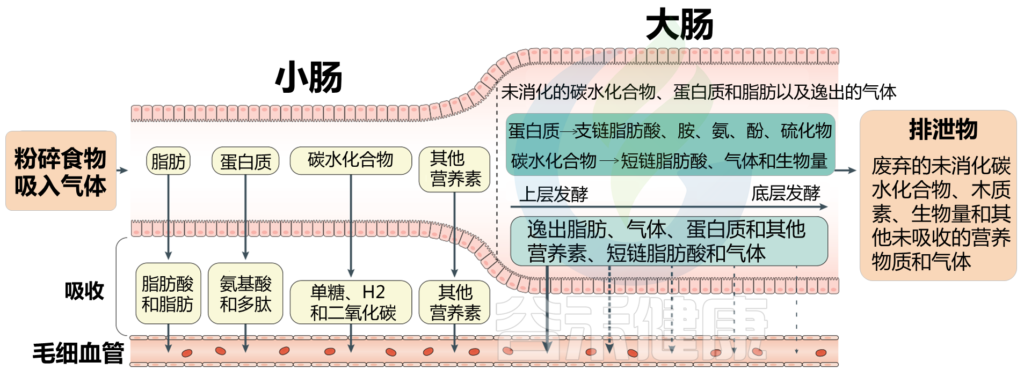
» 气体成分
主要的肠道气体:
氮气(N2)、氧气(O2)、二氧化碳(CO2)、氢气(H2)、甲烷(CH4)
其他气体(以微量浓度存在):
硫化氢(H2S)、一氧化氮(NO)和含硫化合物等
» 肠道气体产生与分布
肠道气体可溶解在肠道的液体成分中,或以气体形式保留在顶空气体中。气体可能通过吞食进入肠道,也可能通过内部化学反应或细菌发酵产生。
在肠道中,氧气(完全)和N2(大部分)来自吞食的空气(尽管少量N2是通过结肠中蛋白质的微生物反硝化作用产生的)。由于N2在通过肠道期间未被吸收或代谢,因此其水平在整个胃肠道内保持合理稳定。
相反,肠道在整个长度内变得越来越无氧,随着管腔微生物数量的增加,氧气浓度逐步降低;因此,结肠中的氧气浓度非常低。(见下图 ↓↓)
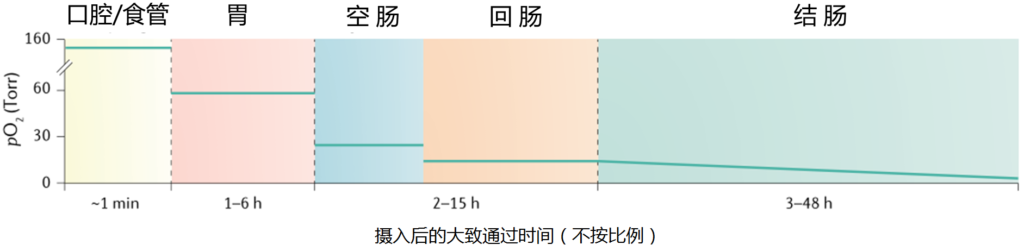
胃和小肠中的二氧化碳部分是通过化学反应产生的,虽然一些吞咽的呼吸性二氧化碳确实会与胃中的气体成分混合。
在近端小肠中,碳酸氢盐(HCO3−), 由胰腺分泌,与十二指肠胃食糜中的盐酸(HCl)发生化学反应,使管腔内pH值从酸性(~3)增加到接近中性(~7.4),并产生CO2.
谷禾健康
收样及报告安排
公司安排2023年1月15至2023年1月28日放假,年后初八开始恢复上班。
13号及之前收到样本安排上机出具报告,14号之后暂停接收样本,1月28号恢复接收样本。
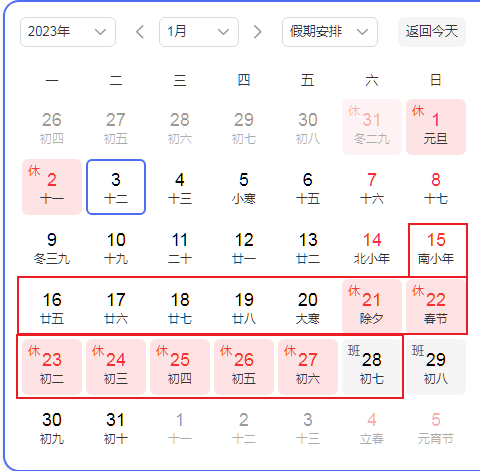
杭州谷禾信息技术有限公司
2023年1月3日
谷禾健康
出于消化和吸收的目的,哺乳动物已经形成了一个非常复杂且高度专业化的胃肠系统,由粘膜屏障维持。
越来越多的数据强调了肠道屏障和肠道通透性对健康和疾病的重要作用。然而,这些术语定义不明确,其评估存在争议,临床意义也没有明确确定。
肠漏症正在成为一种流行病。虽然它尚未被医学界广泛认可,但全球数亿人可能患有与肠漏综合征相关的症状和疾病。
本文讨论的是关于粘膜屏障及其在疾病预防和治疗中的作用的知识。
首先,定义相关术语“肠到屏障”、 “肠道通透性”、“肠漏”。
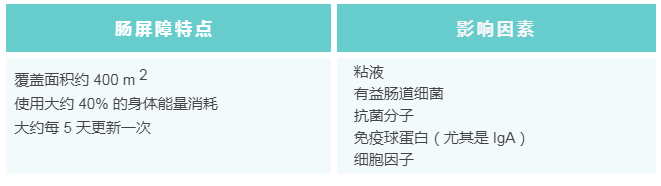
其次,描述了影响肠道通透性的肠道屏障的关键要素。这个屏障代表了一个巨大的粘膜表面,数十亿细菌面对着身体最大的免疫系统。一方面,完整的肠道屏障保护人体免受微生物和毒素的侵袭,另一方面,这个屏障必须打开以吸收必需的液体和营养。这两个看起来矛盾的目标是通过肠道屏障组成的复杂解剖和功能结构来实现统一的,这正是我们常听说的“肠道通透性”。
第三,描述了饮食和细菌对肠道通透性的调节。特别是,必须考虑潜在的屏障破坏因素,例如肠道灌注不足、感染和毒素,还必须考虑选择过量的营养物质、药物和其他生活方式因素。
第四,讨论肠道菌群对肠道通透性的互相影响,以及与通透性增加相关的特定疾病,例如危重病、炎症性肠病、乳糜泻、食物过敏、肠易激综合征,以及肥胖和代谢疾病。所有这些疾病都以炎症为特征,炎症可能由管腔成分易位到宿主中引发。
最后,讨论了评估肠道通透性的方法。手段差异很大,可能评估屏障的不同功能组件。
“肠道屏障”和“肠道通透性”描述了同一解剖结构的两个不同方面。
“肠道通透性”是一个术语,主要由电生理学家使用动物或人类的组织外植体在 Ussing 室中研究上皮通透性而形成。
注:Ussing Chamber(尤斯室,也叫尤斯灌流室)是研究跨上皮转运的工具,可用于包括离子转运、营养物质转运及药物转运等的研究。通过跨上皮转运的研究,可以了解上皮的离子通道机制、营养成分及药物透过上皮的吸收、影响上皮屏障功能以及通透性的因素等等。来源:百度百科
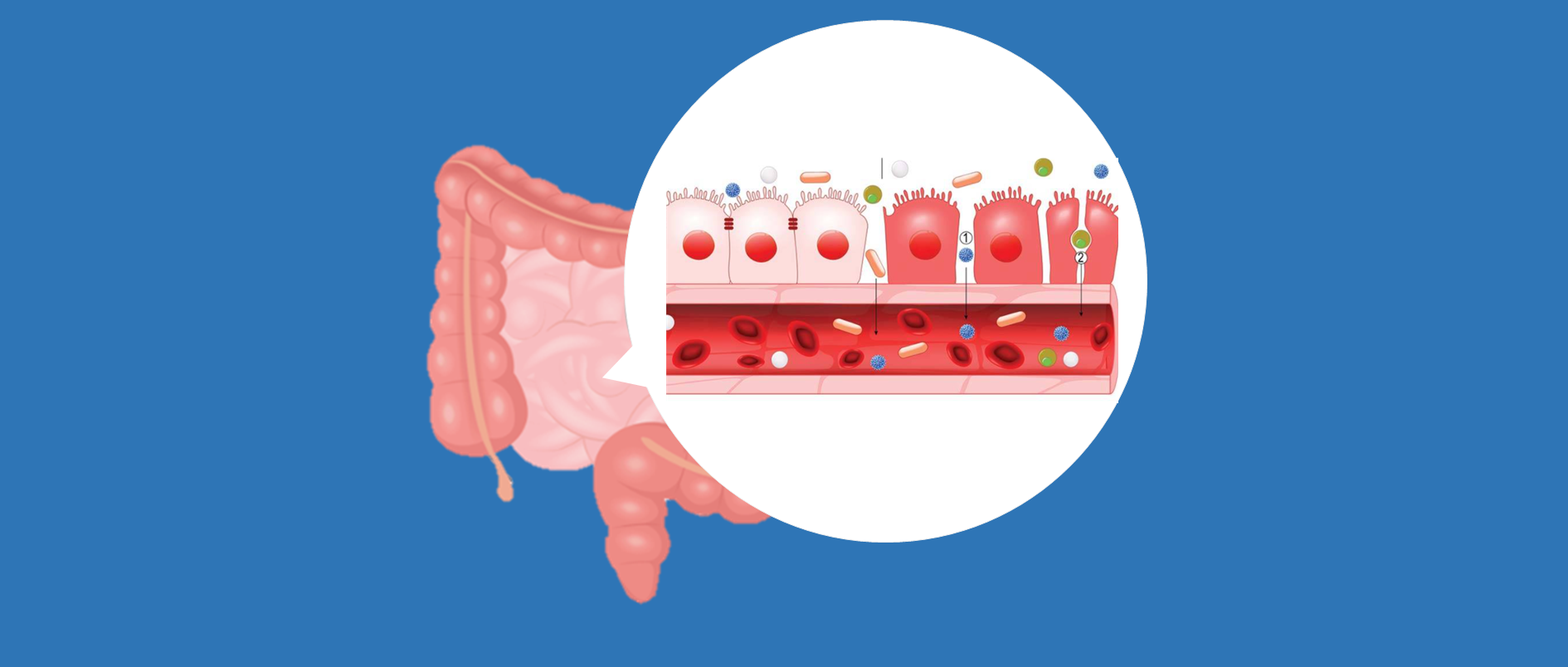
“肠道屏障”用来描述将内部环境与管腔环境分开的复杂结构。物理屏障包括由血管内皮、上皮细胞和粘液层组成。除了这个物理屏障之外,化学物质也参与了屏障功能。它们由消化分泌物、免疫分子、细胞产物如细胞因子、炎症介质和抗菌肽组成,主要由小肠隐窝中的潘氏细胞(Paneth cell)产生。
肠道屏障基本上将肠道内容物与身体分开。它由单层细胞(上皮细胞、传感细胞以及产生酶和神经递质的细胞)组成。这些细胞通过紧密连接(TJ)蛋白连接起来。
肠道屏障是一个动态实体,由多个元素组成,与各种刺激相互作用并做出反应。
首先,在管腔中,细菌和抗原被胆汁、胃酸和胰液降解,共生细菌通过产生抗微生物物质来抑制病原体的定植。
屏障的另一个要素是由未搅拌的水层、糖萼和粘液层组成的小环境,它们通过免疫球蛋白 A (IgA) 分泌以及糖萼和粘液提供的物理屏障防止细菌粘附。由顶端连接复合物连接的上皮细胞具有运输管腔内容物的能力,但它们也通过分泌氯化物和抗菌肽对有害刺激作出反应。
▃ 上皮层