-

 CNAS L23010
CNAS L23010

 国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业
国家高新企业 | ISO9001认证 | 肠道健康精准检测高新技术研发中心 | 专精特新企业 二级病原微生物安全实验室
二级病原微生物安全实验室- 联系电话:+13336028502
- +400-161-1580
- service@guheinfo.com

谷禾健康
过去十余年,新一代测序(NGS)极大推动了人类微生物组研究,我们开始能以前所未有的分辨率,观察人体各生态位的细菌、古菌、真菌、病毒等。
研究发现,微生物组与肥胖代谢问题、消化道疾病、精神健康问题、癌症治疗响应、肝胆问题等健康状况都存在关联,这些前沿发现催生了如肠道菌群检测等专业服务,也推动学界提议将微生物组作为疾病辅助诊断、预后评估、治疗反应预测和监测、指导个性化的饮食方案的新工具。
但现实中,从科研发现到临床应用的转化之路并非坦途。样本差异很大、流程细节多、污染隐蔽、统计学和机器学习容易踩坑、不同实验室做法不一,导致可重复性与可比性不足,临床效用难以确认。
这场“从科研到临床”的转化难,其根源并非微生物本身潜力的匮乏,而在于我们缺乏一套能够驾驭其复杂性的严格“交通规则”。
因此,微生物组要想真正服务临床,需要建立一套覆盖“分析前—分析中—分析后”的全流程质量管理,包含:标准操作程序(SOP)、质量控制(QC)、内部质量评估(IQA)、外部质量评估(EQA)与严格的分析/临床验证。
本文将为你揭示微生物组检测走向临床的关键挑战和解决方案,特别是质量管理框架的构建,为这一前沿领域的规范化发展提供路径。
从使用场景出发,理解“需要什么”远比“能做什么”更重要。微生物组数据可转化的临床用途,例如以下:
辅助筛查/诊断
识别疾病状态下的特征性菌群变化,例如肠道炎症、某些感染相关的菌群失衡。
指标示例:
特定物种丰度、菌群之间比值(如拟杆菌门/厚壁菌门)、功能通路、耐药/毒力基因等。
预后分层
用微生物组的“多样性与稳定性”预测疾病结局或加重风险,如囊性纤维化、消化系统或呼吸系统慢病的加重倾向。
比如说囊性纤维化患者:
菌群多样性下降 → 预测6个月内急性发作风险增加↑
指标示例:
α-多样性、β-多样性等。
微生物组分析指标作为生物标志物
预测治疗反应
典型案例:肿瘤免疫检查点抑制剂(ICIs)的疗效与基线肠道菌群结构相关;部分有益菌群富集与更好的应答有关。
具体来说,癌症治疗响应中,肠道菌群会通过调节免疫系统影响抗癌药物疗效。
指标示例:
响应者群体中特定属/种的富集模式、多样性高低、免疫调节相关功能基因与代谢产物等。
疗效监测/辅助治疗定制
动态随访微生物组,观察治疗(含饮食干预、FMT等)后的菌群变化轨迹;为供/受体匹配提供安全评估。
指标示例:
纵向β-多样性变化、特定功能通路恢复、致病菌负荷下降等。
注:这些指标不是金标准,可以认为是“窗口”,辅助筛查、分层或跟踪,但仍需在具体疾病与人群中完成临床验证与参考区间/阈值的建立。
正因如此,微生物群分析有望为疾病的早期诊断、个性化治疗以及健康管理提供全新的视角和方法,其作为生物标志物的潜力也备受关注。不过,微生物群分析的道路并非一帆风顺,诸多挑战横亘其中。
微生物组自身的善变与复杂性
微生物群本身具有高度的复杂性,它由数量庞大、种类繁多的微生物组成,包括细菌、真菌、病毒等,这些微生物之间以及它们与宿主之间存在着错综复杂的相互作用。
你的饮食、生活方式、服用的药物,都会引起肠道菌群的波动。这种看起来“牵一发而动全身”的特性,使得捕捉一个稳定且有代表性的快照变得困难。
数据分析的挑战
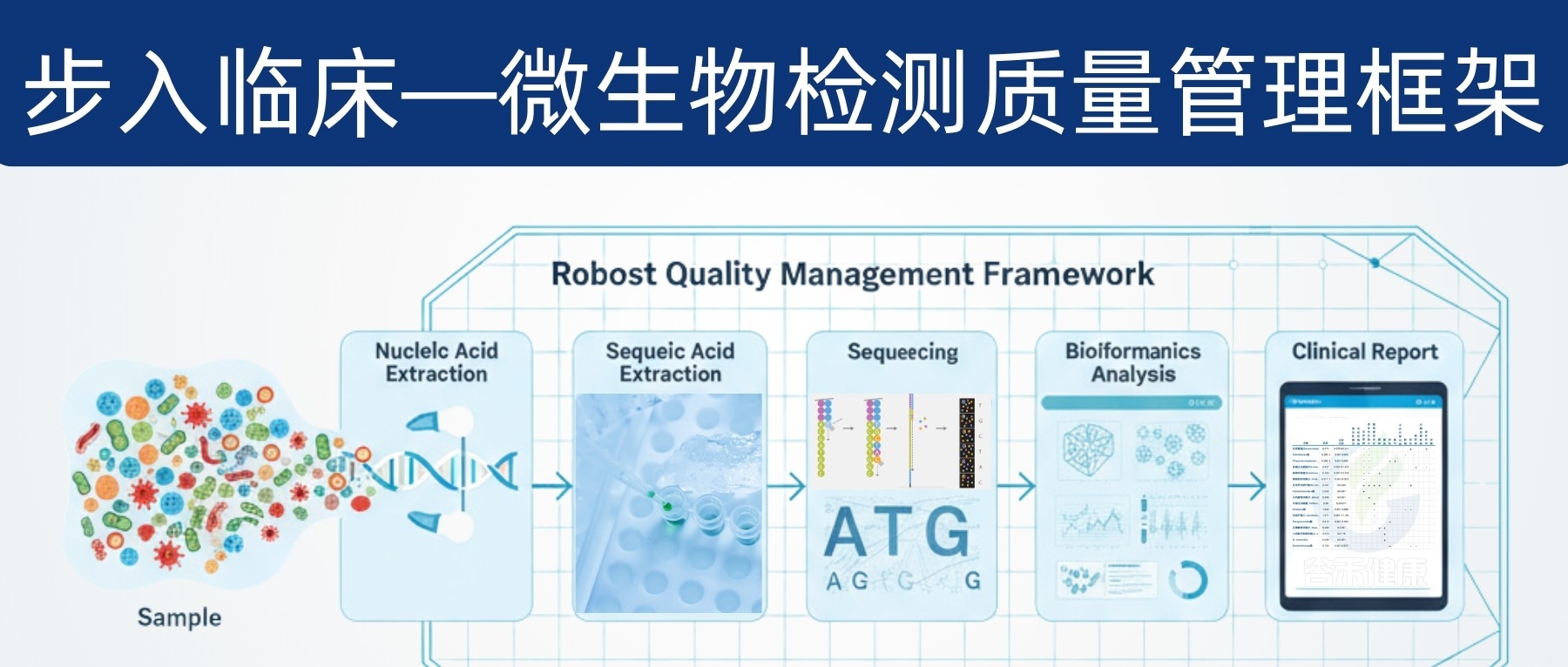
通过高通量测序(NGS)技术,我们能获得海量的微生物组数据。经过复杂的生物信息学处理,才能转化为可解读的分类、功能或多样性指标。
指标繁多:一个样本中可能包含成千上万种微生物或功能基因,为了简化,研究者会使用一些概括性指标,比如“α-多样性”(衡量菌群的丰富度和均匀度),但这可能会丢失关键信息。
统计陷阱:直接将成千上万个菌种与临床表型做关联分析,很容易因为数据本身的特点(高维度、稀疏性)而产生错误的、虚假的结论。
新工具的挑战:虽然机器学习等新工具很有前景,但它们也带来了新的复杂性,比如模型难以解释、验证困难等。
检测技术的细节
从样本离开人体到最终报告生成,整个流程遍布“雷区”,例如在样本储存条件、DNA 提取(方法、试剂盒品牌)、测序(引物的选择)等过程中,任何一个环节的细微差异,都可能对结果产生显著影响,从而导致研究结果的不一致性。
污染问题:尤其是在处理微生物含量低的样本时,实验中微量的污染物可能会导致错误的研究结论。
缺乏标准:目前没有统一的标准化实验流程。从样本处理到DNA提取,操作步骤的微小改动都会显著影响最终结果,导致不同研究之间的数据很难进行比较。
正是这些技术上的不统一和不可控,使得微生物组检测领域迫切需要一个标准化的质量管理体系,以确保未来临床检测的准确性和可靠性。
面对微生物群分析中的诸多挑战,建立一套完善的质量管理框架显得尤为重要。这个框架涵盖了从实验设计到结果解读的各个环节,旨在确保微生物群分析的准确性、可靠性和可重复性。它不仅为科研人员提供了标准化的操作流程,也为临床医生提供了更准确的诊断依据,有助于推动微生物群分析在精准医疗中的应用。
验 证:迈向精准医疗的基石
在微生物群分析中,新检测方法的引入如同打开一扇通往未知领域的门,但这扇门能否通向正确的方向,需要通过严格的验证来确保。
分析验证是对检测方法本身性能的评估,包括准确性、精密度、灵敏度、特异性等指标。
临床验证则是将检测方法应用于临床样本,验证其在实际临床环境中的有效性和可靠性。
临床效用评估是从临床实践的角度出发,评估检测方法对患者治疗决策和健康结果的影响。
临床效用——临床有效性,有用
微生物组检测必须同时满足两个条件:
这一验证阶段通常通过前瞻性临床试验进行,专门评估检测带来的实际改善。
虽然大型机构可能主导全面评估,但各临床实验室仍需验证自己的实施方案。
验证参考样本和数据的可用性对于充分评估任何新检测的实施至关重要。
目前微生物组分析还缺乏明确指导方针。现有的基因测序应用建议可作为基础,但验证过程必须根据特定方法和临床目的调整。尽管已提出许多潜在应用,但尚无一种完成正式临床验证。
标准操作程序:确保结果一致性的关键
严格标准流程的重要性
为整个微生物群分析工作流程定义严格的标准操作程序(SOPs)是确保结果一致性和可靠性的关键。
SOPs 好比一份详细的地图,为实验人员提供了清晰的操作指南,从样本采集到结果报告的每一个环节都有明确的规定和要求。
在湿实验室协议方面,任何细微的差异都可能对实验结果产生显著影响,因此需要严格控制实验条件,确保实验操作的一致性。
生物信息学工具的特殊需求
除了SOPs的基本要求(如书面文档和过程可追溯性)外,宏基因组学分析还特别需要生物信息学工具和参考数据库的稳定性。为了支持微生物组分析的各种应用,分析软件应具备可配置性和灵活性,同时保持易用、快速、稳定和可追溯的特点。
在宏基因组学分析的实际应用中,我们通过灵活组合构建定制化的数据分析脚本,满足不同分析需求的可配置性要求,确保分析工具既具备强大的统计分析能力,又保持了高效的数据处理性能。
谷禾所有生物信息学脚本的版本控制采用集中化管理策略,统一存储和备份于公司服务器中。对于分析所需的软件环境,本地统一部署管理,同时,建立了镜像作为备份方案,这种双重保障机制有效提升了分析环境的可靠性和可重现性。
这种基于官方资源的配置策略不仅保证了分析结果的标准化和可比较性,还为后续的方法验证和结果重现提供了坚实的技术基础。
质量控制:保障分析可靠性的支柱
质量控制(QCs)是质量管理的基石。它通过检查阴性和阳性对照样本,确保从采样到数据分析的全过程顺利完成。同时,设置“检查点”来监控关键步骤,只有满足标准才能继续。
核心原则
全流程把关:用阳性和阴性对照验证“采样→提取→建库→测序→分析”是否顺利完成。
检查点门槛:每个关键步骤设定接受标准,未达标则暂停或重做。
确保可重复与可比:严格执行标准,保证实验室内一致性和对外有效性。
内 部 质 量 控 制
阳性对照——验证能检出
最佳选择是标准化“细胞模拟”群落(已知组成),可覆盖提取到分析的全链条。
其他形式:DNA 模拟物、计算机模拟读段(不能覆盖湿实验室环节)。
平台特定补充,如 Illumina 常加 PhiX 作标定。
阴性对照——监测污染
污染物来源于实验室环境、试剂(“试剂盒”)或邻近样本(交叉污染),并在采样、DNA 提取、文库制备和测序(“索引跳跃”)过程中累积。
必备
提取空白、建库空白;必要时加“采样阴性”(例如清水、试剂空白)。
建议
所有阴性对照都进行测序,即便检测不到 DNA,也用于评估随机污染。
管理
建立污染物追踪库,长期记录与预警异常事件。
处理
策略
过滤阴性对照中出现的菌群(注意避免过度过滤导致假阴性)。
设阈值过滤或用专用工具(如 decontam、microDecon、Recentrifuge)识别并剔除污染;其中Recentrifuge能识别交叉污染。
注意
事项
低生物量样本(皮肤、下呼吸道)更易受污染影响;基于低丰度物种计数的指标更脆弱,像 α 多样性这类“更广义”的指标相对稳健。
关 键 检 查 点( 示 例 )
1
是否该做这个检测
核对临床指征、限制与必要咨询,避免无效或重复检测。
2
样本是否合适
采样部位要与临床问题匹配(肠道/皮肤/下呼吸道等生态位差异巨大)。
采样时机需控制(用药间隔、治疗/操作后多长时间、是否空腹/进食等),避免节律与饮食干扰。
采样质量判定(如直肠拭子是否有粪染、痰是否黏稠非唾液等)。
3
DNA 提取量与可用性
–工具:qPCR、荧光法(如 Qubit)、分光光度法(如 NanoDrop)。
–高生物量样本(粪便):若文库或测序读数异常偏低,提示提取/建库失败。
–低/中生物量样本(呼吸道、皮肤):常规前瞻性定量,关注污染风险。
–补充:靶向 qPCR(细菌 16S、真菌 ITS)可校正宿主 DNA 干扰,并将相对丰度换算为近似绝对量。
4
文库定量与标准化
–目的:测序前做等摩尔混合,也可侧面反映样本质量/微生物负荷。
–方法:荧光定量+片段长度分析。
5
测序质量
常用的测序数据指标:每样本读数、错误率、Q30 占比、读长等;
使用平台指标与 FastQC 辅助诊断问题。
6
生物信息学处理质量
流程:质控、过滤、修剪、聚类/ASV、组装、比对等;
检查各步骤的日志与评分,确认成功产出预期结果(分类、功能、耐药/毒力等)。
7
最终临床审核
由熟悉这方面的临床微生物学家把关,确认流程完整、报告恰当(必要时展示稀有但临床相关的菌群)。
早期需为临床医生提供解读支持;长期通过培训提升全科室对结果的理解与应用。
外部和内部质量评估:提升分析水平的助力
外部质量评估(EQA/能力验证)
核心做法是向多家实验室发放同一份参考材料,然后回收并比较各家的检测结果。可以理解为由权威第三方机构组织的 “全国统考”。
微生物组许多指标对流程细节非常敏感,因此需要定制化的 EQA 方案。
提出的两种解决方案:
结论导向 EQA:看不同实验室在临床结论上是否一致。
可能出现中间指标不一致(如多样性指数不同),但基于各自参考范围/阈值,最终临床判断一致(如粪菌移植失败风险高)。
举例:A、B两个实验室用了不同的方法,测出的多样性指数分别是3和5,数据不同。但他们根据各自的参考标准,可能都得出了“此患者粪菌移植失败风险高”的相同结论。这样就算通过。
局限:目前微生物组检测在临床上还没有太多公认的应用,所以这种考核很难开展。
方法导向 EQA:不讨论临床结论,只比对技术表现是否一致。看大家用同样的方法、同样的样本,能否得到同样的结果。
内 部 质 量 评 估(IQA)
简单来说,内部质量评估就是实验室内部的 “自我检查” 机制,用来确保自己的检测流程稳定可靠。
具体怎么做?
重复检测参考样本:定期把一个已知的、标准的参考样本拿出来重新检测一遍,看结果是不是和以前一样。这能保证实验流程长时间内没有发生偏移。
检测平行样本:把一份临床样本分成两份(或多份),当作两个独立的样本来检测。理论上,这两份得到的结果应该高度一致。这能检验操作的精密度和可重复性。
为什么要这么做?
因为目前针对微生物组分析的官方 “外部质量评估” (EQA)(也就是由权威机构组织的全国性或全球性大比对)还很缺乏。
解决方法:通过实验室内部的自我检查,以及组织几个本地或合作实验室之间搞 “比对测试”,可以很好地补充或弥补这个空缺,从而保证检测质量。
目前尚无充分验证的生物标志物可应用于常规实践,基于微生物组的生物标志物验证仍是一项持续而复杂的工作。下面的三个案例展示了常见挑战与可行做法。
案例一
Akkermansia muciniphila 预测免疫治疗反应
某些肠道菌与免疫检查点抑制剂疗效相关,Akkermansia muciniphila的存在被认为可能提示更好的应答。
► 分析验证(如何确保检测准确)
这个验证相对直接,因为目标很明确:只需要评估整个流程能否可靠地找到、识别并量化 A. muciniphila 这一种细菌。
具体做法是:
► 临床验证(这个指标在病人身上真的有用吗)
这需要通过设计良好的临床试验来证明。
可以这样做:
一个重要的提醒:
在做临床验证时,必须特别注意研究人群的构成。因为 种族、地理位置、饮食习惯 等因素都会显著影响肠道菌群。这些是混杂因素,如果不加考虑,可能会导致错误的结论。
案例二
用肠道菌群多样性指标,预测造血干细胞移植的预后
这个案例关注的是一个叫 “α-多样性”(Alpha-diversity) 的指标。简单来说,它衡量的是你肠道里微生物种类的“丰富程度”和“均匀程度”。就像一个生态系统,种类越多、分布越均匀,多样性就越高。
有研究发现,在接受造血干细胞移植前,如果患者肠道菌群的辛普森多样性指数的倒数(Inverse Simpson Diversity)越高,他们之后的总体生存率也越高。
► 分析验证(如何确保多样性算得准)
验证这类指标的难点在于,我们永远不知道样本里到底有多少种细菌(也就是说没有标准答案)。所以验证方法是直接看,我们测出的“多样性指数”和“病人的生存结果”之间有没有稳定的关联性。
► 临床应用与解读 (怎么用这个结果)
得到一个多样性指数后,怎么判断是高是低呢?
挑战:混杂因素
和上个案例一样,最大的挑战还是“混杂因素”。
人种、地区、饮食都极大地影响肠道菌群。
这个研究的优点是它的数据来自三大洲,说明结论可能具有普遍性。但反过来,这种大杂烩式的方法也可能掩盖了只针对某个特定人群的规律。
案例三
机器学习分析肠道菌群用于肝细胞癌筛查
这个案例探讨的是一个更前沿的方法:训练一个人工智能(AI)模型,让它通过分析一个人的肠道菌群数据,来判断他患上肝癌的风险。
核心思路
训练模型:先收集大量已知是“健康”或“肝癌患者”的肠道菌群数据。
让AI学习:把这些数据喂给AI模型,让它自己学习和总结出区分健康和患病人群的菌群模式。
预测:模型训练好后,当输入一个新人的菌群数据时,它就能根据学到的规律给出一个判断,这个人得肝癌的风险有多高。
如何验证这种AI模型?(这是最复杂的部分)
– 检查AI的学习方法
要确保AI不是过度训练得出的结论,比如,它可能学到了一些无关的线索或过度拟合部分线索,总的来说,AI需要比较大量的数据输入才可靠,此外对输入数据的准确性,描述等要求非常高。
– 严格的考试
绝对不能用训练过的数据来测试模型。这就像学生考试,不能用做过的练习题来当考卷。必须用一个全新的、独立的“验证数据集”来测试它的准确性。
– 在真实世界中检验
理想情况下,要把这个模型拿到一个更大、更多样化的人群中去检验,看它是否依然有效。
如何评价疾病预测模型的好坏?
评价一个疾病预测模型的精准性,远非一个“准确率”数字那么简单,其核心在于模型的性能是否与它的临床应用目的相匹配。我们必须综合评估一系列关键指标,并理解它们之间的内在权衡关系。
首先,要区分几个概念:
关键在于,这些指标的应用价值取决于场景:
对于疾病筛查(如早期癌症筛查):我们担心的是假阴性(把病人判为健康),这会延误宝贵的治疗时机。
因此,一个好的筛查模型必须追求灵敏度高,哪怕这会牺牲一部分特异度,导致一些假阳性(把健康人判为有病)。因为假阳性者可以通过后续更精确的检查(如CT、活检)来排除,只是虚惊一场,但漏诊的代价是巨大的。
对于疾病的确诊或指导重大治疗:此时,我们担心的是假阳性(误诊)。
给一个健康人施以不必要的、甚至有害的治疗是不可接受的。因此,一个用于确诊的模型必须拥有很高的特异度和阳性预测值(PPV),确保诊断的可靠性。
总的来说,一个模型的优劣是相对的。因此,最适合特定任务的模型,就是最佳模型。
评估微生物组的预测模型时,不能孤立地看某个指标,而应结合ROC曲线和AUC值来综合判断其区分能力,并根据其在临床路径中的具体角色(是用于大规模初筛,还是用于辅助诊断,辅助干预),来决定我们更看重哪个维度的性能。
关于这方面内容,详见谷禾之前的文章:
这套质量管理框架,为临床实验室未来如何开展微生物组检测指明了方向。
有些做法是现成的,比如必须使用“阴性对照”样本来监控污染。还有一些可以直接借鉴其他领域的经验,比如开展“内部质控”(IQA)和“外部质评”(EQA)。
因为如果科研人员在发表研究成果(比如发现一个新的生物标志物)时,就能提供详细的标准化操作流程(SOPs)和完整的质控数据,那么临床实验室就能更快、更容易地把这项新技术“接过去”并复现出来。
这将大大加快从基础研究成果,到病人能从中受益的诊断工具的转化过程。
编辑
目前,行业标准化已不再是纸上谈兵,本文所探讨的质量管理框架源自长期的实践与迭代。围绕实验室的人员、设备、环境、方法及管理流程等关键环节,谷禾进行了持续的系统化改进,建立并执行一套可追溯、可评估的质量管理体系,推动相关工作走向规范化。
在既有质量控制与验证基础上,谷禾将继续推进临床与微生物领域的转化应用,持续完善流程与方法,稳步提升落地能力与服务可用性。
主要参考文献
Scherz V, Greub G, Bertelli C. Building up a clinical microbiota profiling: a quality framework proposal. Crit Rev Microbiol. 2022 May;48(3):356-375.
Caminero A, Tropini C,et al., Credible inferences in microbiome research: ensuring rigour, reproducibility and relevance in the era of AI. Nat Rev Gastroenterol Hepatol. 2025 Jul 31.
Britton RA, Verdu EF, Di Rienzi SC, Reyes Muñoz A, Tarr PI, Preidis GA; Biotherapeutics Subcommittee of the AGA Center for Gut Microbiome Research and Education. Taking Microbiome Science to the Next Level: Recommendations to Advance the Emerging Field of Microbiome-Based Therapeutics and Diagnostics. Gastroenterology. 2024 Nov;167(6):1059-1064.